Практикум 8
Построение профиля и поиск подходящих последовательностей
В этом првктикуме подбирался профиль, который успешно опознавал бы последовательности рибосомального белка RL1
протеобактерий. Для построения профиля я взял 7 последовательностей белка различных протеобактерий (seqs_for_HMM.fasta), и применил
к ним команды hmm2build и hmm2calibrate. Получившийся профиль доступен поссылке profile.out.
Далее я искал в БД "Swissprot" удовлетворяющие моему профилю последовательности командой hmm2search с пороговым E-value 10.
Все находки представлены в таблице на втором листе.
Анализ результатов поиска
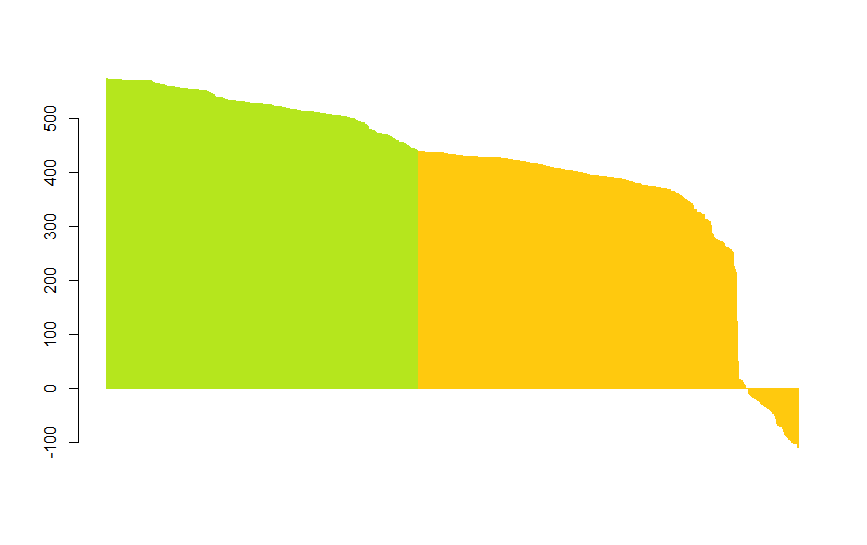
На ниже представленном рисунке представлена гистограма весов всех находок. Зелёным помечена область, последовательности
в которой прнадлежат только протеобактериям. Как видно из графика, большого перепада в весе находок между протеобактериями
и другими бактериями не наблюдается.
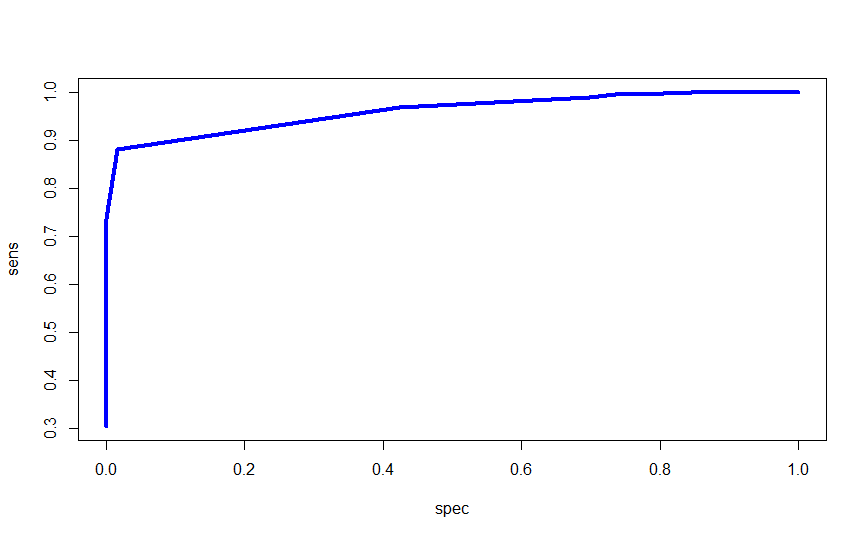
 Следующий график отображает ROC-кривую находок.AUC приведённой ROC-кривой построенного профиля имеет достаточно большой,
что указывает на хорошую предсказательную способность профиля. При граничном E-value 1*e-130 (вес выравнивания более 450)
профиль имеет высокую чувствительность и специфичность.
Следующий график отображает ROC-кривую находок.AUC приведённой ROC-кривой построенного профиля имеет достаточно большой,
что указывает на хорошую предсказательную способность профиля. При граничном E-value 1*e-130 (вес выравнивания более 450)
профиль имеет высокую чувствительность и специфичность.
| >=450 | True | False |
| Positive | 375 | 7 |
| Negative | 51 | 442 |
Sensitivity --
Specificity --
Precision -- 0.98
Accuracy -- 0.93
© Максим Григорьян, 2016
 Следующий график отображает ROC-кривую находок.AUC приведённой ROC-кривой построенного профиля имеет достаточно большой,
что указывает на хорошую предсказательную способность профиля. При граничном E-value 1*e-130 (вес выравнивания более 450)
профиль имеет высокую чувствительность и специфичность.
Следующий график отображает ROC-кривую находок.AUC приведённой ROC-кривой построенного профиля имеет достаточно большой,
что указывает на хорошую предсказательную способность профиля. При граничном E-value 1*e-130 (вес выравнивания более 450)
профиль имеет высокую чувствительность и специфичность.
