
Эволюционные домены. Банки Pfam и InterPro
Банки семейств доменов Pfam и InterPro позволяют находить и характеризовать родственные белки. Для этого анализируют аминокислотные последовательности белков.
Белки родственны по последовательности, потому что порядок аминокислот закодирован в ДНК, в которой редко бывают ошибки (хотя, разумеется, есть исключения). После расхождения генов одного и того же белка по каким-либо причинам (например, существование видов в разных условиях) последовательности начинают накапливать различия (например, из-за мутаций), что уменьшает их сходство.
Для определения родства важно оценить 3D-структуру белка, которая является более консервативной, чем последовательность.
И, наконец, если белки родственны по последовательности, то они, как правило, будут выполнять одинаковые функции. Но это правило выполняется не всегда.
Для структурирования белков и удобства поиска их родственников, вводят специальные обозначения:
- Эволюционный домен белка – это консервативный фрагмент его последовательности, который можно найти при выравнивании последовательностей
- Домен – длинный консервативный участок в выравнивании нескольких белков (с общим консервативным участком), который выделяется на фоне неконсервативных участков, т.е. это структурная единица, которую можно найти во множественном выравнивании
- Семейство – коллекция гомологичных белков
- Клан – группа родственных записей
- Повтор – короткая нестабильная единица, которая при множественном копировании образует стабильную структуру
- Мотив – короткий консервативный участок, который отвечает за какую-то функцию белка
Все эти разделения и понятия используются при поиске и описании доменной архитектуры белка TENI_BACSU по банкам Pfam и InterPro.
Таблица 1
Описание доменной архитектуры белка TENI_BACSU по данным Pfam
Схема из Pfam

|
|||||
| Схема из Pfam | |||||
| № | Pfam AC | Pfam ID | Семейство доменов | Положение в последовательности | Клан |
| 1. | PF02581 | TMP_TENI | Тиамин монофосфат синтазы (состоит из фермента ТМР и регуляторного белка TENI) | 3-179 | Семейство является членом клана TIM_barrel (CL0036), в состав которого входит всего 57 членов |
Данные о домене
В банке Pfam содержится информация только информация о 31 доменной архитектуре с рассматриваемым доменом. Информацию о нем можно найти здесь.
Домен TMP_TENI входит в состав 5722 последовательности, описание которых можно найти здесь.
В банке Pfam есть 62 белковые последовательности с известной 3D-структурой, в состав которых входит изучаемый домен. Эти структуры доступны по этой ссылке.
Pfam позволяет получить множественное выравнивание (в формате HTML сохраненная версия) с исследуемым доменом.
Описание частоты встречаемости доменов в организмах по отдельности
Один и тот же домен может встречаться в архитектурах разных белков.
В архитектуре белка TENI_BACSU только один домен. Поэтому для описания частоты встречаемости доменов рассмотрим THIED_COREF (Q8FTH8) – белок, который катализирует фосфорилирование HMP-P в HMP-PP и HMP в HMP-P.
В состав архитектуры этого белка входит три домена (Рис.1).

Pис.1. Доменная структура белка THIED_COREF
Таблица 2
Описание частоты встречаемости доменов в организмах по отдельности
| № | Pfam AC | Pfam ID | Полное название семейства доменов | Положение в последовательности | Клан |
| 1. | PF02581 | TMP_TENI | См. Таблицу 1 | ||
| 2. | PF08543 | Phos_pyr_kin | Фосфометилпуримидин киназы | 232-479 | Это семейство является членом клана Ribokinase (CL0118), который всего включает в себя 6 членов |
| 3. | PF03070 | TENA_THI-4 | TENA/THI-4/PQQC | 539-736 | Это семейство является членом клана HO (CL0230), который всего включает в себя 5 членов |
Таблица 3
Встречаемость доменов белка THIED_COREF по данным Pfam
| Название домена | |||
| TMP_TENI | Phos_pyr_kin | TENA_THI-4 | |
| Количество доменных архитектур с доменом | См. в разделе «Данные о домене» | 34 | 23 |
| Количество последовательностей с доменом | 8058 | 3227 | |
| Количество последовательностей с известной 3D- структурой, в которых есть домен | 38 | 70 | |
| Количество видов, у которых есть домен | 4351 | 2223 | |
Согласно информации в таблице, рассматриваемые домены встречаются часто. Проанализируем встречаемость доменов в конкретных группах организмов (Таблица 4).
Таблица 4
Представители домена PF08543 и домена PF03070 в организмах разных таксонов
| Таксон | Количество белков с доменом PF08543 | Количество белков с доменом PF03070 | |
| Эукариоты | Зеленые растения | 69 | 61 |
| Грибы | 328 | 244 | |
| Животные | 38 | 4 | |
| Остальные эукариоты | 53 | 12 | |
| Археи | 159 | 147 | |
| Бактерии | 7342 | 2716 | |
| Вирусы | 0 | 0 | |
Домен PF08543 (Phos_pyr_kin) встречается в природе чаще, чем домен PF03070 (TENA_THI-4). Оба домена встречаются преимущественно в белках бактерий, меньше – в белках архей, еще меньше – в белках эукариотических организмов и совсем отсутствуют в вирусных белках.
Для рассматриваемых доменов нет дерева, так как они встречаются в большом количестве белков. Однако, визуализацию их распространенности отображают круговые диаграммы (Рис. 2 и Рис. 3)
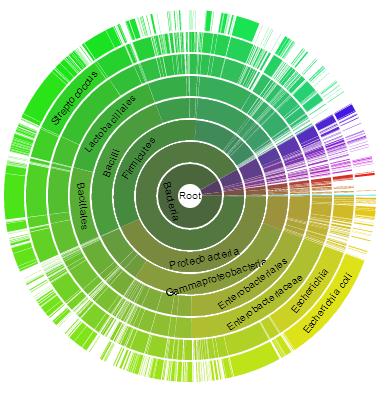
Pис.2. Диаграмма распространенности домена Phos_pyr_kin
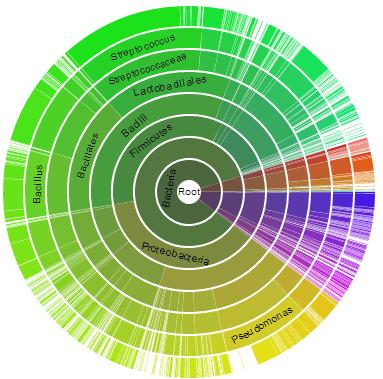
Pис.3. Диаграмма распространенности домена TENA_THI-4
Сравнение описания мотивов в разных банках семейств, по данным InterPro
InterPro (integrated resource of protein families, domains and functional sites) – это единая интегрированная база данных, включающая в себя информацию о семействах белков, доменах, мотивах и тд и составленная на основе содержимого других БД (например, Pfam, Prosite).
С помощью InterPro можно получить карту, на которой будут отражены все сайты, мотивы и тд (Рис. 4).

Pис.4. Карта с разметкой всех мотивов, сайтов, доменов для последовательности белка TENI_BACSU, интегрированные в InterPro
Самый короткий мотив – TMP_TBNI (PF02581). Он начинается 4ым аминокислотным остатком и заканчивается 179ым (длина: 175 АО). Этот мотив отвечает за фосфорилирование тиамина. Мотив TMP_TBNI описан в банке PROSITE. На карте он обозначен белым цветом.
Самый длинный мотив – PTHR20857. Начинается 1ым аминокислотным остатком, а заканчивается 205ым (длина: 205АО). Мотив PTHR20857 описан в банке PROSITE. На карте он обозначен серым цветом.
Кроме того, интегрированы еще и известные структуры белков, взятые из разных банков. Границы структурных доменов не отличаются от границ доменов Pfam.