
MEME – программа для построения множественного локального выравнивания. В настоящее время является самой популярной в своей области применения.
Актуальность программы заключается в том, что локальное выравнивание в ряде случаев имеет больший биологический смысл, чем глобальное, потому что позволяет точнее определить функционально значимый участок (мотив, сайт или домен), искажение которого влечет за собой изменение функций белка (например, сайт связывания с лигандом или активный центр). Такие участки консервативны (содержат мало эволюционных изменений или же не содержат их вовсе), потому что выгодны с точки зрения эволюции.
Программа MEME отличается от программ глобального множественного выравнивания (например, MUSCLE) тем, что не увеличивает вес выравнивания за счет добавления гэпов, сохраняя тем самым биологический смысл.
С помощью MEME был проведен поиск мотивов (блоков достоверного выравнивания) в белке TENI_BACSU и его гомологах.
Таблица 1
| Мотив | Число последо-ватель-ностей в нем | Длина мотива | E-value | |
| MOTIF 1 | 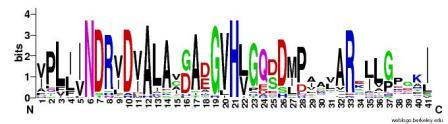 |
25 | 41 | 1.7e-402 |
| MOTIF 2 |  |
25 | 21 | 7.1e-181 |
| MOTIF 23 | 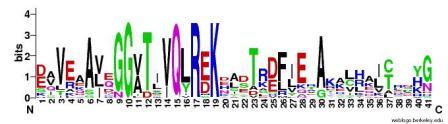 |
20 | 41 | 6e-237 |
Для сравнения были созданы три fasta-файла, которые содержали описанные мотивы по отдельности. Действие было выполнено с помощью команды –seqret пакета EMBOSS.
JalView помогает визуализировать сравнение мотивов и выравнивания.
Сперва было проанализировано совпадение с выравниванием каждого из мотивов.
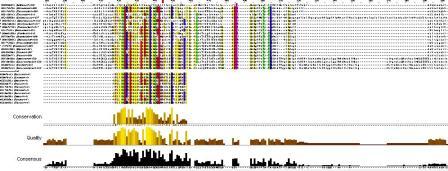
Как видно на рисунке, выравнивание MUSCLE и мотив 1, полученный программой MEME абсолютно идентичны (за исключением отдельных единичных аминокислот в некоторых белках).

Выравнивание MUSCLE и мотив 2, полученный программой MEME, также имеют Несущественные различия из-за того, что программа MUSCLE вставила колонку гэпов.
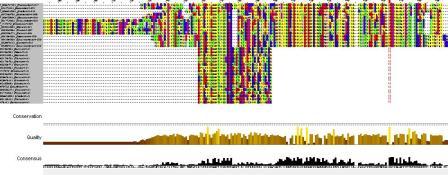
Выравнивание MUSCLE и мотив 3, полученный программой MEME, имеют некоторые различия. Например, первые две последовательности имеют 3 аминокислоты, которых нет в других последовательностях.
С помощью программы MAST был проведен поиск мотивов в выравнивании, полученном программой Pfam (см. Эволюционные домены. Банки Pfam и InterPro)
На вход программы был подан msf-файл с последовательностями. Полученный результат можно просмотреть здесь.
Мотивы искались в 89 последовательностях, содержащихся в файле с выравниванием.
Все три мотива встречаются в 71 последовательности.
Мотивы 1 и 2 встречаются в 11 последовательностях.
Только мотив 3 встречается в одной последовательности.
Мотив 3 и 2 встречаются в 6 последовательностях.
При визуальном сравнении можно сказать, что выравнивание Pfam хорошо соотносится с мотивами. Подтверждением этого является наличие всех трех мотивов в большом количестве белков из выравнивания.
Литература:
Timothy L. Bailey and Michael Gribskov, "Combining evidence using p-values: application to sequence homology searches", Bioinformatics, 14(48-54), 1998.