
Комплекс ДНК-белок
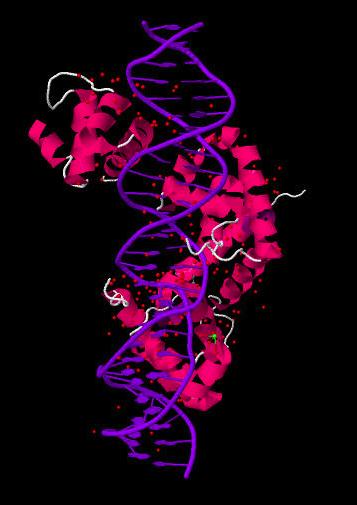
В банке данных PDB был получен файл 1RIO.pdb с ДНК-белковым комплексом (Рис.1).
С помощью инструментов JMol определим несколько множеств атомов ДНК в рассматриваемом файле (Рис.2, Рис.3, Рис.4).
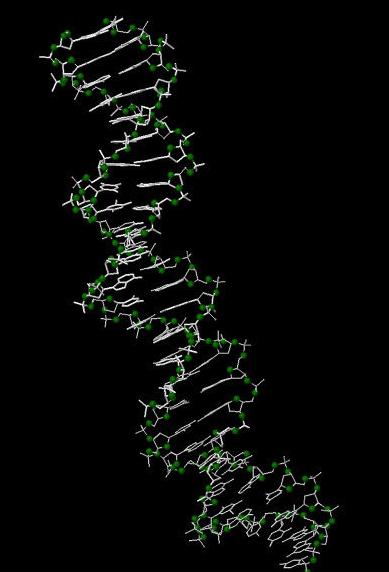
На Рис.2 изображена ДНК с выделенным зеленым цветом множеством атомов set1 (атомы кислорода 2'-дезоксирибозы).
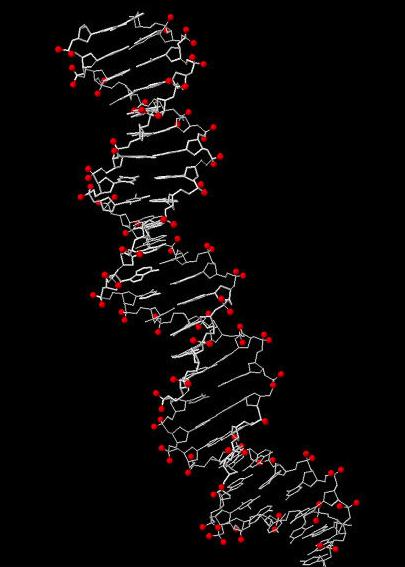
На Рис.3 изображена ДНК с выделенным красным цветом множеством атомов set2 (атомы кислорода в остатке фосфорной кислоты ).
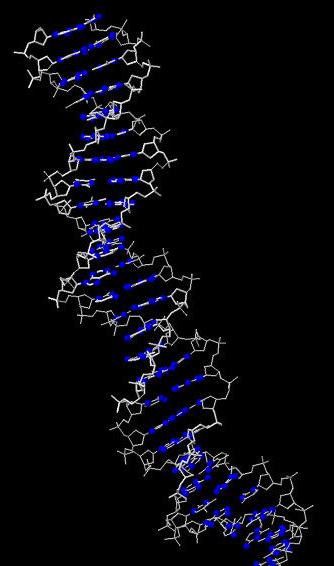
На Рис.4 изображена ДНК с выделенным красным цветом множеством атомов set3 (атомы азота в азотистых основаниях).
Все представленные отображения наборов атомов можно получить с помощью скрипта.
С помощью выбранных наборов атомов можно описать ДНК-белковые контакты в рассматриваемой структуре 1RIO.pdb.
Будем считать полярными атомы кислорода и азота, а неполярными атомы углерода, фосфора и серы. Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5Å.
Таблица 1
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 1 | 38 | 39 |
| остатками фосфорной кислоты | 8 | 7 | 15 |
| остатками азотистых оснований со стороны большой бороздки | 1 | 7 | 8 |
| остатками азотистых оснований со стороны малой бороздки | 2 | 0 | 2 |
Наибольшее число контактов наблюдается у остатков 2’-дезоксирибозы, что связано с ее наиболее доступным (из рассматриваемых групп) пространственным положением. Кроме того, для остатков 2’-дезоксирибозы характерно большое количество неполярных контактов, что связано с большим количеством неполярных атомов в белке. Также с пространственным положением, но наименее доступным, связано наименьшее число контактов белка с остатками азотистых оснований со стороны малой бороздки. Можно еще отметить, что остатки азотистых оснований образуют больше контактов (особенно, неполярных) со стороны большой бороздки.
Визуализировать контакты ДНК с белком можно еще и с помощью программы nucplot. Получая на входе файл в формате (старом) pdb, программа выдает несколько файлов в различных форматах. Нас интересует файл с популярной схемой (Рис.5) отображения всех контактов нуклеотидов ДНК с другими молекулами (с аминокислотными остатками белка в том числе).
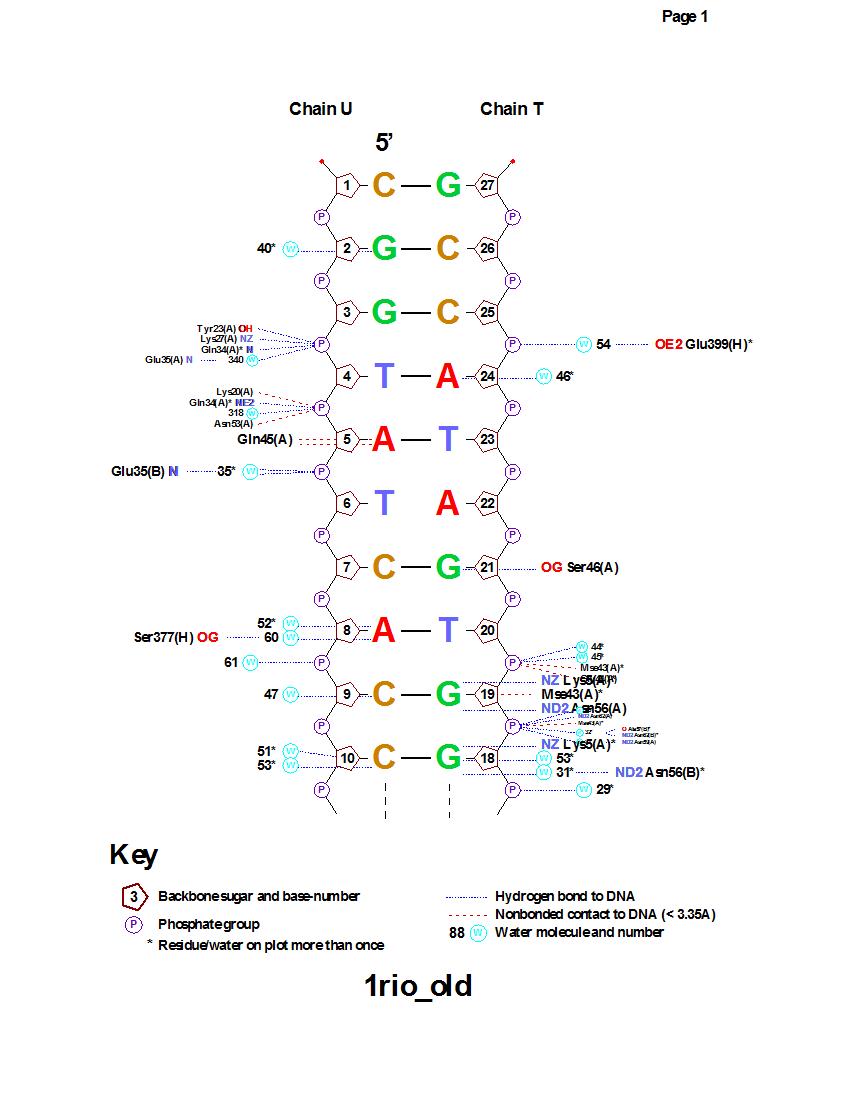
Рассмотрим контакты ДНК с Н-цепью белка.
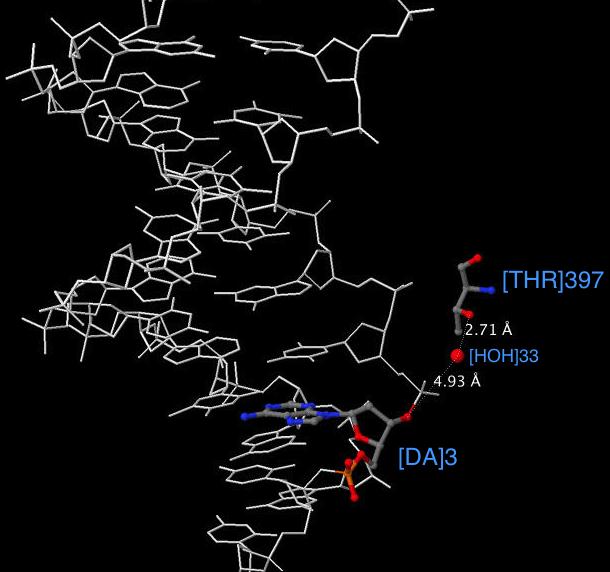
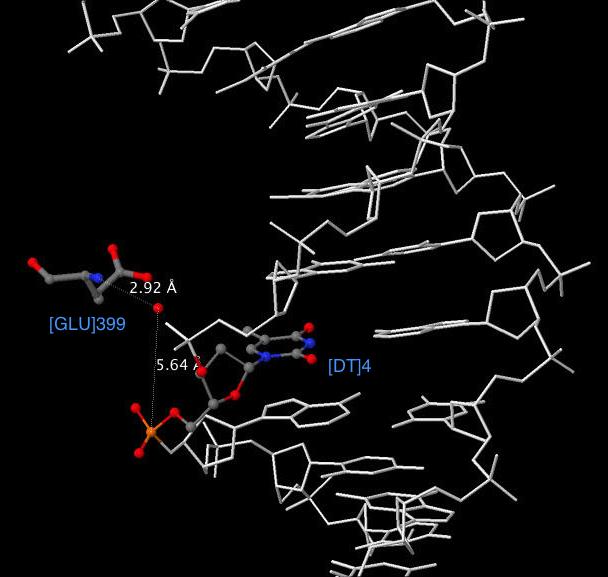
Согласно выше представленной схеме наибольшее число взаимодействий наблюдается с Thr 397(H) и Glu 399(H). Контакты этих аминокислотных остатков с ДНК представлены на Рис.6 и Рис.7, которые получены с помощью JMol.
Представленные на Рис.6 и Рис.7 взаимодействия осуществляются посредством молекул воды. Пунктиром обозначены контакты, и приведены длины связей в ангстремах.
Для распознавания ДНК в структуре важны аминокислотные остатки Arg 409(H) (связывается с основанием G5) и Gln 414(H) (связывается с основанием Т19), контакты с ДНК которых представлены на Рис.8 и Рис.9 (получены с помощью JMol).
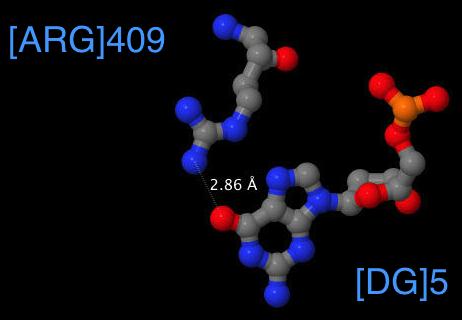
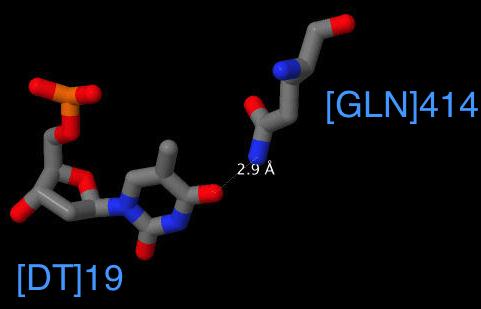
На Рис.8 и Рис.9 пунктиром изображены взаимодействия ДНК и белка, и указаны длины связей в ангстремах.
Предсказание вторичной структуры заданной тРНК
Предсказание вторичной структуры будет проводиться для структуры 1F7V.pdb.
1. Предсказание вторичной структуры тРНК путем поиска инвертированных поворотов
Программа einverted из пакета EMBOSS позволяет найти инвертированные участки в нуклеотидных последовательностях. При изменении параметров, можно получать различные результаты. Наиболее удачными были следующие параметры:
- Gap penalty [12]: 5
- Minimum score threshold [50]: 20
- Match score [3]: 10
- Mismatch score [-4]: -5
Полученный файл сохранен в файле 1f7v2.inv.
2. Предсказание вторичной структуры тРНК по алгоритму Зукера
Программа mfold из пакета EMBOSS реализует алгоритм Зукера.
Единственным параметром, который следует менять, является параметр Р. Он указывает, на сколько процентов выдаваемое предсказание структуры может отличаться по своей вычисленной энергии от оптимального. Чем больше значение этого параметра, тем больше вариантов предсказания будет выдано.
Заметим, что, чем ниже значение параметра Р, тем удачнее оказываются предсказания. Самое удачное предсказание представлено на Рис.10
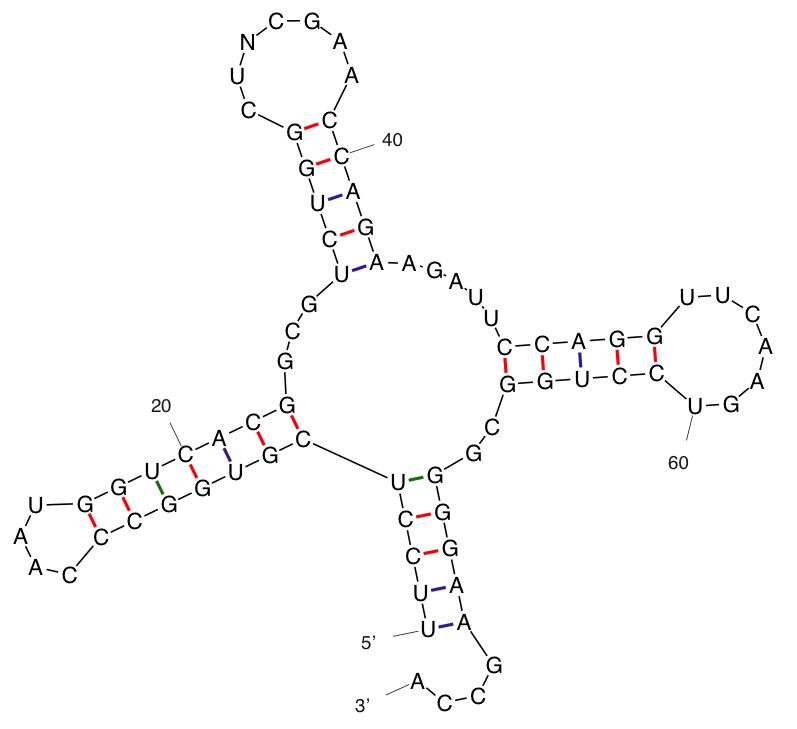
Таблица 2
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | (5') 1-7 (3') (5') 66-72 (3') всего 7 пар |
Предсказано 5 пар из 7 реальных | Предсказано 5 пар из 7 реальных(кроме 6ой и 7ой) |
| D-стебель |
(5') 10-12 (3') (5') 23-25 (3') Всего 3 пары |
Предсказано 2 пары из 3 реальных | Предсказано 3 пары из 3 реальных |
| T-стебель |
(5') 49-52 (3') (5') 62-65 (3') Всего 4 пары |
Предсказано 3 пары из 4 реальных | Предсказано 4 пары из 4 реальных |
| Антикодоновый стебель |
(5') 37-44 (3') (5') 26-33 (3') Всего 8 пар |
Предсказано 5 пар из 8 реальных | Предсказано 5 пар из 8 реальных |
| Общее число канонических пар нуклеотидов | 22 | 15 | 17 |