
Реконструкция филогенетических деревьев
Для работы были отобраны бактерии. Для них было составлено филогенетическое дерево
(Рис. 1)
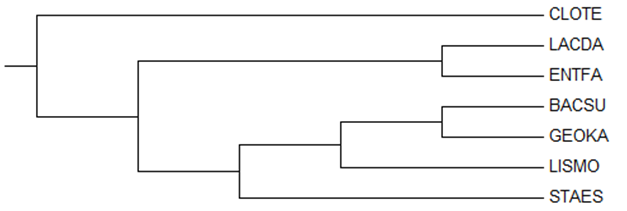
Рис. 1 Филогенетическое дерево выбранных бактерий
Пользуясь таксономическим сервисом NCBI определяем, к каким таксонам относятся отобранные бактерии (Таблица 1)
Таксономическая принадлежность бактерий
| Мнемоника | Название | Тип | Класс | Отряд | Семейство |
| BACSU | Bacillus subtilis | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| ENTFA | Enterococcus faecalis | Firmicutes | Bacilli | Lactobacillales | Enterococcaceae |
| GEOKA | Geobacillus kaustophilus | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| CLOTE | Clostridium tetani | Firmicutes | Clostridia | Clostridiales | Clostridiaceae |
| LISMO | Listeria monocytogenes | Firmicutes | Bacilli | Bacillales | Listeriaceae |
| LACDA | Lactobacillus delbruecki | Firmicutes | Bacilli | Lactobacillales | Lactobacillaceae |
| STAES | Staphylococcus epidermidis | Firmicutes | Bacilli | Bacillales | Staphylococcaceae |
На дереве есть ветви, которые отделяют отдельные таксоны, которые на Рис. 2 соответствуют раскраске в Таблице 1.
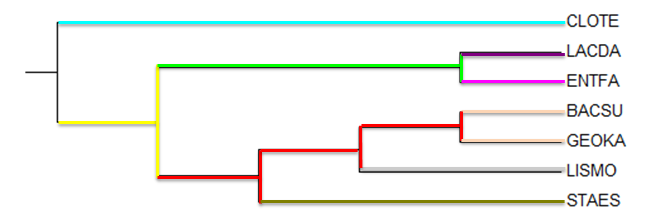
Рис. 2 Филогенетическое дерево выбранных бактерий с обозначением ветвей, выделяющих таксоны
Для дальнейшей работы была выбрана функция IF2 (фактор инициации трансляции 2). Филогенетическое дерево будет реконструировано по белкам этого семейства.
Из Swiss-Prot, используя команду –seqret, были получены последовательности белков с данной функцией из рассматриваемых бактерий.
С помощью программы JalView было выполнено выравнивание полученных последовательностей (Рис. 3), на котором раскраска зависит от процента идентичности. Изображение приведено в «блочной» форме.
С помощью программы JalView можно реконструировать филогенетическое дерево четырьмя способами. Изображения деревьев были получены программой Mega.
Average Distance Using % Identity
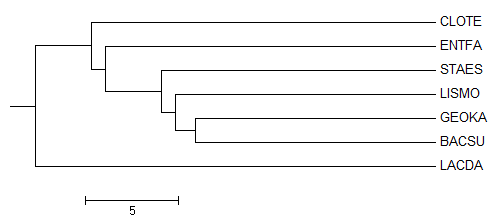
Рис. 4 Реконструированное филогенетическое дерево
Дерево на Рис. 4 отличается от правильного дерева:
- Наличием {CLOTE, LACDA} vs {ENTFA, STAES, LISMO, GEOKA, BACSU, LACDA}
- Отсутствием {LACDA, ENTFA} vs {CLOTE, BACSU, GEOKA, LISMO, STAES}
Neighbour Joining Using % Identity
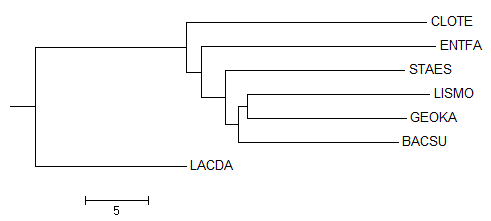
Рис. 5 Реконструированное филогенетическое дерево
Дерево на Рис. 4 неукорененное отличается от правильного дерева:
- Наличием {CLOTE, LACDA} vs {ENTFA, STAES, LISMO, GEOKA, BACSU, LACDA}
- Отсутствием {LACDA, ENTFA} vs {CLOTE, BACSU, GEOKA, LISMO, STAES}
- Присутствием {GEOKA, LISMO} vs {CLOTE, ENTFA, STAES, BACSU, LACDA} (в правильном дереве есть ветвь {GEOKA, BACSU} vs {CLOTE, ENTFA, STAES, LISMO, LACDA}).
Average Distance Using BLOSUM62
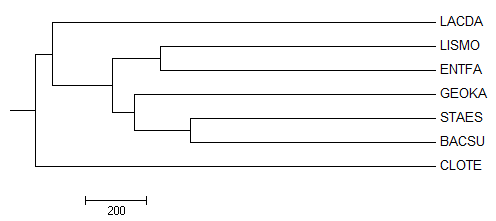
Рис. 6 Реконструированное филогенетическое дерево
Нетривиальные ветви, отличающие дерево на Рис. 6 от правильного:
- {LACDA, CLOTE} vs {LISMO, ENTFA, GEOKA, STAES, BACSU}
- {LACDA, LISMO, ENTFA, CLOTE} vs {GEOKA, STAES, BACSU}
- {LACDA, LISMO, ENTFA, CLOTE, GEOKA} vs {STAES, BACSU}
- {LISMO, ENTFA} vs {LACDA, GEOKA, STAES, BACSU, CLOTE}
Neighbour Joining Using BLOSUM62
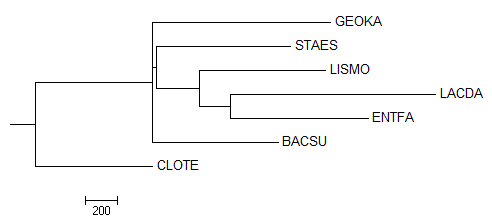
Рис. 7 Реконструированное филогенетическое дерево
Дерево на Рис. 5 неукорененное и отличается от правильного дерева присутствием:
- {GEOKA, CLOTE} vs {LISMO, ENTFA, GEOKA, STAES, BACSU},
- {GEOKA, CLOTE, BACSU} vs {STAES, LISMO, LACDA, ENTFA},
- {GEOKA, CLOTE, BACSU, STAES} vs {LISMO, LACDA, ENTFA}.
В правильном дереве присутствует только одна нетривиальная ветвь дерева с Рис. 7: {LACDA, ENTFA} vs {GEOKA, STAES, LISMO, BACSU, CLOTE}.
Используя метод "Maximum Parsimony" в программе Mega, выполнили еще одну реконструкцию дерева, укоренив его в одну из ветвей (Рис. 8)
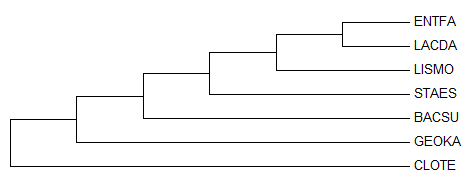
Рис. 8 Реконструированное филогенетическое дерево
Отличия от правильного дерева:
- только одна нетривиальная ветвь, которая есть в правильном дереве:
{LACDA, ENTFA} vs {GEOKA, STAES, LISMO, BACSU, CLOTE} - нетривиальные ветви, которых нет в правильном дереве:
{ENTFA, LACDA, LISMO} vs {STAES, BACSU, GEOKA, CLOTE},
{ENTFA, LACDA, LISMO, STAES} vs {BACSU, GEOKA, CLOTE},
{ENTFA, LACDA, LISMO, STAES, BACSU} vs {GEOKA, CLOTE}.