
- e3qh2A1 и e3qh2D1 с границами 1-202 в цепях А и D соответственно;
- e3qh2B1 и e3qh2C1 с границами 0-200 в цепях В и С соответственно.
В CATH были обнаружены следующие домены:
- 3qh2A00 и 3qh2D00 с границами 1-202 в цепях А и D соответственно;
- 3qh2B00 и 3qh2C00 с границами 0-202 в цепях А и D соответственно.
Для работы был выбран белок TenI с PDB-кодом 3QH2, использовалась программа DSSP (выдача программы).
Таблица 1
| Вторичная структура | Границы в PDB | Границы в DSSP |
| Спираль | 38-52 | 38-53 |
| Спираль | 184-202 | 185-201 |
| Тяж | 59-62 | 59-62 |
| Тяж | 59-62 | 117-120 |
Из Таблицы 1 видно, что определенные в DSSP и в PDB-файле границы β-листов совпадают полностью. Одна из рассмотренных спиралей отличается на один остаток, вторая – на два остатка. Это тоже очень хорошее совпадение в определении границ.
C помощью SheeP была построена карта β-листа (Рис. 1).
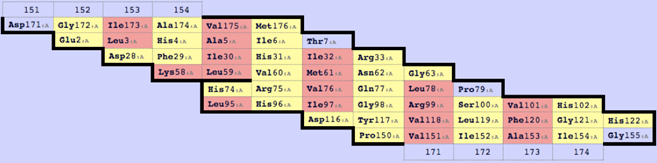
На Рис. 2, полученном также с помощью SheeP изображен рассматриваемый β-лист.
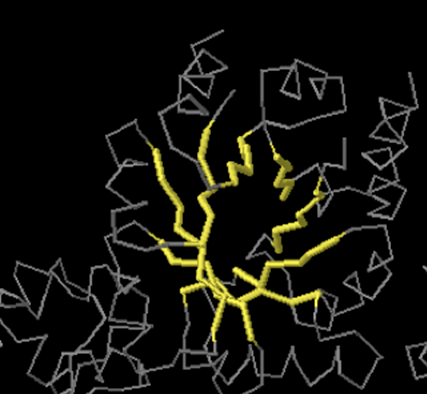
На Рис. 3 отражено соответствие одного столбца на карте и хребта в β-листе
На карте, которая представлена на Рис. 1, обращенные внутрь бочонка остатки обозначены желтым.
Используя Original Stride, для участка β-листа (взято 3 тяжа с несколькими хребтами) была получена карта водородных связей.

В нижнем правом углу видна нерегулярная структура, которая присутствует в выбранных тяжах.
С помощью PDBeFold были найдены структурные гомологи для цепи А из 3QH2(Рис. 2): 1xi3:A из Pyrococcus furiosus (Рис. 3), 1g4p:B из Bacillus subtilis (Рис. 4), 4bk9:C из Zymomonas mobilis (Рис. 5), 3iwp:G из Homo sapiens (Рис. 6). Выравнивание структур представлено на Рис.1.


 |
 |
 |
 |
Было получено еще одно выравнивание с помощью программы Muscle. Сравнение выравниваний (сравнивались большие буквы) показало, что структурное выравнивание в целом лучше, хотя отличий совсем немного (одно из них на Рис. 7).
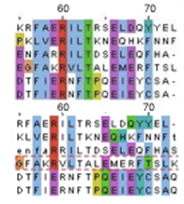
На Рис. 7 видно, что во множественном выравнивании некоторые остатки не выровнены, а в структурном – выровнены. Такими, например, являются Glu в 66 позиции на структурном выравнивании и Glu, находящиеся в выравнивании по последовательности в позициях 65-68. Также на структурном выравнивании Тht выровнены в позиции 63, а в выравнивании по последовательности они совсем не выровнены.
Поиск по сходству в PDBeFold
Для работы был выбран самый первый домен из списка – 1В0М А:203-315. Поиск осуществлялся в PDBeFold(Рис. 1). Результаты отсортированы по RMSD. Поиск осуществлялся при параметрах, заданных по умолчанию.

В результате поиска было получена выдача из 37 записей. Запрошенного домена среди них нет.
Для работы были выбраны опять же самые первые в списке константные домены Т-клеточного рецептора 1OGA из α-цепочки регион d:118-202 (Рис. 1) и из β-цепочки регион е:119-245 (Рис. 2).

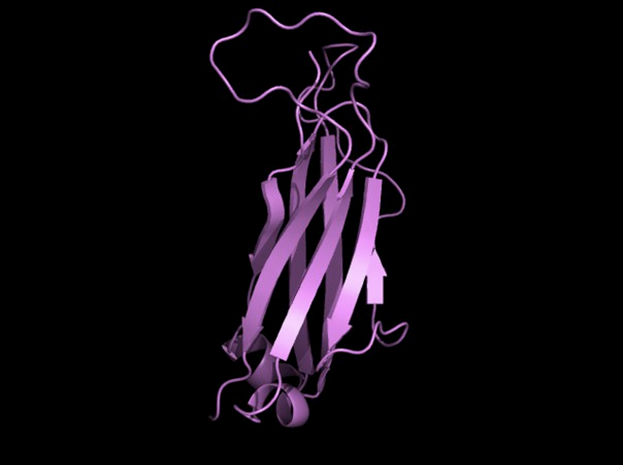
Карты β-листов для выбранных доменов были построены с помощью SheeP. Далее были выбраны соответствующие друг другу β-листы из α-цепочки (Рис. 3) и из β-цепочки (Рис. 4).


Предоставленные карты находятся в одной ориентации.
Дальнейшее выравнивание рассматриваемых β-листов основывается на консервативных остатках цистеина Cys134 из α-цепочки и Cys145 из β-цепочки и соседних с ними остатков. С помощью команды PyMol было построено совмещение (Рис. 5):
pair_fit t_alpha///133-135+122+175+155/CA, t_beta///144-146+127+192+172/CA

Анализируя Рис. 5 можно утверждать, что топологии неплохо совпадают. Отчетливо просматривается соответствие хода полипептидной цепи и β-листов обеих цепей.
С помощью ресурса Clud были найдены гидрофобные кластеры в структуре 3QH2 (Рис. 1).




При параметрах по умолчанию видны гидрофобные взаимодействия внутри белка. По мере увеличения строгости параметров гидрофобные кластеры меняют свои размеры, но все равно покрывают практически весь белок.
Каждый гидрофобный кластер включает в себя две субъединицы.
Для отображения гидрофобных кластеров на интерфейсе двух цепочек была выбрана структура 3Q8T(Рис. 5).
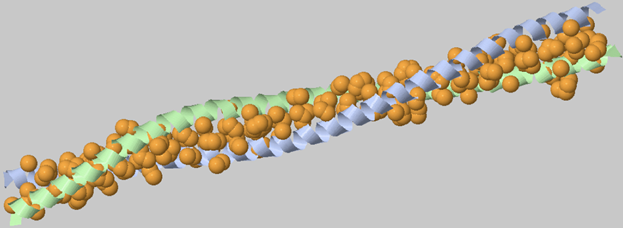
Гидрофобный кластер между двумя спиралями крайне необходим для контакта.
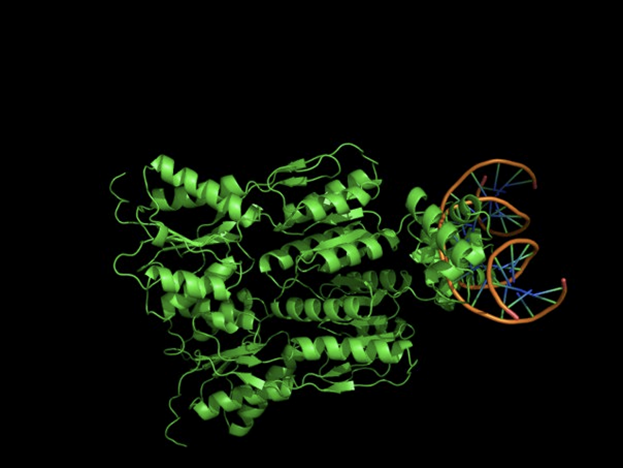
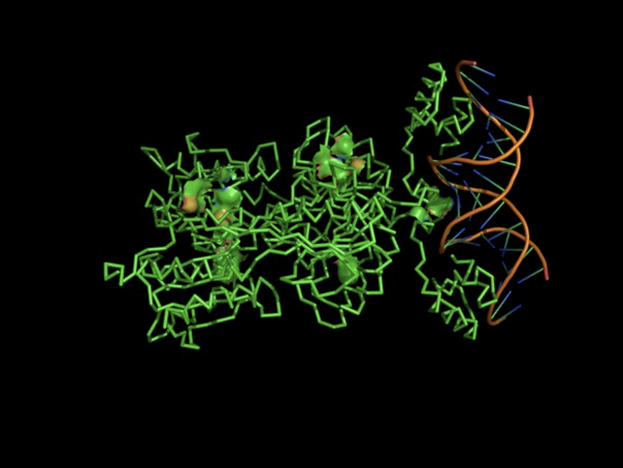
Для работы был выбран комплекс димера пуринового репрессора с ДНК 1BDH. Визуализация выполнена с помощью PyMol.



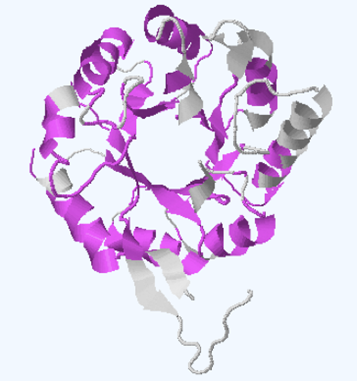
Для сравнения доменов различных классификаций использовалась структура 3QH2.
В Pfam для рассматриваемого белка был определен один домен(Рис. 1):

Границы домена в Pfam: 4-179.
Удивительно, но поиск в SCOP не дал результатов («This PDB entry is not classified in SCOPe 2.05»). В записи, относящейся к рассматриваемому белку, есть только данные записи PDB.
В ECOD были обнаружены следующие домены:
Из представленных данных видно, что домены ECOD и CATH полностью совпадают. Кроме того, они соответствуют делению субъединицам белка. Домен Pfam не совпадает с доменами из ECOD и CATH. Это, вероятно, связанно с тем, что Pfam ищет эволюционные, а не структурные домены.
На сайте были скачены все последовательности белков, определенные при помощи метода электронной микроскопии, одним файлом. Было найдено 924 последовательности. Для их нахождения использовано Advances search -> Experimental method -> ELECTRON MICROSCOPY. Причем из найденных структур экспериментальные данные имеются для 877 (файл с этими последовательностями).
На странице сайте PDB для структуры 3QH2 в разделе Structure Similarity структурные гомологи, полученные программой jFATCAT, не были найдены.
Поэтому для работы была использована структура 1YAD, с помощью которой решалась фазовая проблема при определении структуры 3QH2. В разделе Structure Similarity (ссылка - http://www.rcsb.org/pdb/explore/structureCluster.do?structureId=1YAD) .
Результат алгоритма jFATCAT выдается при идентичности 40% и P-value = 0.001. Обнаружено 515 гомологов.
В PDBeFold было найдено 207 записей, из которых только 8 совпадают с результатом поиска jFATCAT.