 |
Филогенетические деревья
|
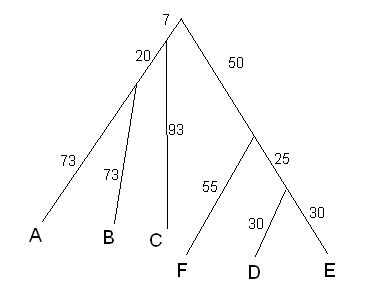
Исходное дерево: (((А:73,В:73):20,С:93):7,((D:30,Е:30):25,F:55):50);
В корненаходится ген dkgA длиной 828 нуклеотидов. Поэтому количество мутаций высчитывается
по следующей формуле:
кол-во мутаций=длина ветви*8,28
Ветви:
A B C D E F
* * . . . .
* * * . . .
* * * . . *
* . . . . .
. * . . . .
. . * . . .
. . . * . .
. . . . * .
. . . . . *
Мутанты гена dkgA были получены с помощью следующего скрипта:
msbar DKGA_gene2.fasta abc.fasta -point 4 -count 58 -auto
msbar abc.fasta ab.fasta -point 4 -count 166 -auto
msbar ab.fasta a.fasta -point 4 -count 604 -auto
msbar ab.fasta b.fasta -point 4 -count 604 -auto
msbar abc.fasta c.fasta -point 4 -count 770 -auto
msbar DKGA_gene2.fasta fde.fasta -point 4 -count 414 -auto
msbar fde.fasta f.fasta -point 4 -count 455 -auto
msbar fde.fasta de.fasta -point 4 -count 207 -auto
msbar de.fasta d.fasta -point 4 -count 248 -auto
msbar de.fasta e.fasta -point 4 -count 248 -auto
Потом по последовательностям мутантов на листьях дерева деревья были восстановлены 3-мя
алгоритмами:
- максимального правдоподобия
+-------------F
+---------------4
| | +------E
+--2 +----3
| | +-------D
| |
| +----------------B
|
1--------------------C
|
+--------------------A
- Neighbor-joining
+--------------------C
!
! +----------------B
! !
3---4 +-------D
! ! +----1
! +-----------------2 +-------E
! !
! +------------F
!
+---------------------A
- UPGMA
+----------------------------------------A
+-3
+----------4 +----------------------------------------B
! !
! +-----------------------------------------C
--5
! +--------------D
! +----------1
+--------------------------2 +--------------E
!
+-------------------------F
| A B C D E F |
Истинное дерево |
Максимальное правдоподобие |
Neighbor-joining |
UPGMA |
| * * . . . . |
+ |
- |
- |
+ |
| * . * . . . |
- |
+ |
+ |
- |
| * * * . . . |
+ |
+ |
+ |
+ |
| * * * . . * |
+ |
+ |
+ |
+ |
В деревьях, восстановленных разными методами, совпадают почти все ветви. Путаница возникает
только при разбиении группы А,В,С. Это может быть из-за того, что ветвь между С и АВ на
исходном дереве довольно короткая (20) по сравнению с длиной самих ветвей А,В,С (73, 73, 93).
Поэтому при получении мутантов исходной нуклеотидной последовательности все эти потомки
различаются примерно одинаково, а не С сильнее отличается от А и В, чем они между собой.
Из-за этого разные программы по-разному восстанавливают деревья.
С помощью программы fseqboot было получено 100 бутстреп-реплик выравнивания (т.е.
100 выравниваний, полученных из данного путем копирования случайно выбранной половины
позиций и удаления оставшейся половины). На основе этих выравниваний были построены
100 деревьев программой dnaml. После этого программа fconsense построила консенсусное
дерево для этих 100 деревьев.
Ветви консенсусного дерева:
A B C D E F
* . * . . . 75
* * * . . . 100
* * * . . * 95
Таким образом, видно, что по топологии реальное дерево и консенсусное не совпадают,
они отличаются одной внутренней ветвью. Около ветвей указаны их бутстреп-значения,
т.е. во скольких деревьях из 100 встретилась эта ветвь. Для реального дерева эти
значения таковы:
A B C D E F
* * . . . . 17
* * * . . . 100
* * * . . * 95
Если бы исходное дерево было неизвестно, то с помощью бутстреп-анализа было бы поддержано
дерево, не совпадающее с реальным. Так как 17 - очень маленькое число по сравнению
с 75, правильная ветка не была бы поддержана. Но все остальные ветви, поддержанные
бутстрепом, верные и значит этот метод хорошо подходит для проверки построенных деревьев.
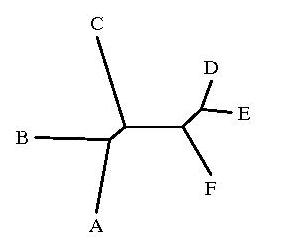
Дерево, созданное пргораммой fdrawtree на основе скобочной записи:
На страницу 4-го семестра
© Моросанова Мария


