 |
Сравнение разных способов оценки эволюционных расстояний между 2-мя генами
|
С помощью программы msbar я получила 5 мутантных последовательностей в таком порядке:
DKGA_gene2.fasta - mutant0.1 - mutant0.2 - mutant0.25 - mutant0.3 - mutant0.4 - mutant0.5.
Соответственно "истинные" расстояния между ними составляют 10,10,5,5,10,10 замен на 100 нуклеотидов. Мутанты я получила с помощью следующего скрипта:
msbar DKGA_gene2.fasta mutant0.1.fasta -point 4 -count 84 -auto
msbar mutant0.1.fasta mutant0.2.fasta -point 4 -count 84 -auto
msbar mutant0.2.fasta mutant0.25.fasta -point 4 -count 42 -auto
msbar mutant0.25.fasta mutant0.3.fasta -point 4 -count 42 -auto
msbar mutant0.3.fasta mutant0.4.fasta -point 4 -count 84 -auto
msbar mutant0.4.fasta mutant0.5.fasta -point 4 -count 84 -auto
cat DKGA_gene2.fasta > itog.fasta
cat mutant0.1.fasta >> itog.fasta
cat mutant0.2.fasta >> itog.fasta
cat mutant0.25.fasta >> itog.fasta
cat mutant0.3.fasta >> itog.fasta
cat mutant0.4.fasta >> itog.fasta
cat mutant0.5.fasta >> itog.fasta
Дальше с помощью программы distmat были определены попарные эволюционные расстояния между
последовательностями. Выбирая соответствующий метод оценки, я получила попарные расстояния
вычисленные без поправки и по формуле Джукса - Кантора. На основе этих данных были созданы таблицы,
которые находятся в этом файле.
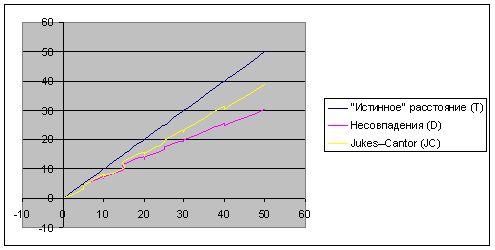
На приведенном графике показана зависимость 2-х разных оценок расстояния от "истинного" расстояния,
на которм видно, что оценка по формуле Джукса - Кантора точнее.
На страницу 4-го семестра
© Моросанова Мария


