Была выбрана бактерия Shigella flexneri, плазмида (). (gi 58044990 gb AY879342.1 Shigella flexneri plasmid pSF5, complete sequence)
Это грамм-отрицательная бактерия, вызывающая диарею. Инфекции обычно лечат антибиотиками, но некоторые штаммы становятся резистентными. Не угрожающие жизни случаи обычно не лечатся, чтобы штамм не стал устойчивым к антибиотику.)
Вот картиночка:

В NCBI gene по запросу Shigella flexneri plasmid pSF5 была выдана таблица в текстовом виде: gene_result.txt. Далее я зарегистрировалась на сайт RAST, создала новое задание(Jobs => Upload new job), получила аннотацию. Ссылка на отчет работы RASTa: http://rast.nmpdr.org/rast Всего найдено 217 генов. Результат аннотации доступен по ссылке: rast_sambr.xls
Далее для визуализации результата я воспользовалась SEED viewer.
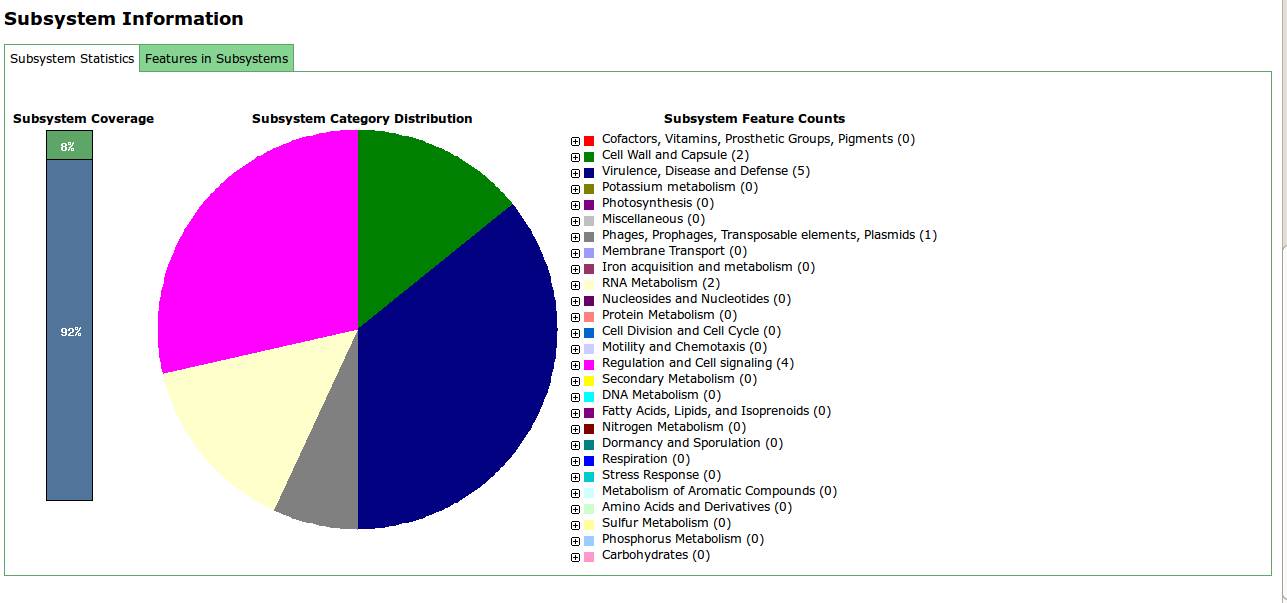
Диаграмма распределения различных генов по их функциональным характеристикам.
Более подробную информацию можно получить из Features in Subsystems.
Далее была создана сводная таблица с помощью excel. Результат доступен по ссылке: compare.xls
Чтобы найти количество контигов с совпадающими старт-кодонами, я использовала команды: cut -f 2 compare.csv | sort | uniq -c | sort -g > сount_uniq.txt (335 строчек) cut -f 2 compare.csv > сount_all.txt (383 строчки) Таким образом, по старт-кодону совпадают 48 генов.
cut -f 3 compare.csv | sort | uniq -c | sort -g > сount_uniq_stop.txt (304 строчки)
По стоп-кодону совпадают 79 генов. Далее с помощью команд: cut -f 2,3 compare.csv > сount_all_2.txt cut -f 2,3 compare.csv | sort | uniq -c | sort -g > сount_uniq_2.txt было выяснено, что совпадают и по старт и по стоп кодону 47 генов(здесь не учитывались случаи, когда старт и стоп были поменяны местами). В таблице, полученной из RAST, 217 генов, в таблице, полученной из Genbank - 167 генов.
Результат поиска первой последовательности показал, что белок скорее всего является гликозил трансферазой. Это достаточно точно соответствует аннотации RAST.Результат поиска первой последовательности показал, что наибольшее совпадение с различными hypothetical protein или c YacA plasmid stabilization protein [Shigella sp. MO17].
Результат поиска первой последовательности показал, что белок имеет функцию регуляции транскрибции. Под это описание подходит и аннотация genbank - Co-activator of prophage gene expression IbrB.
В целом, можно отметить, что RAST аннотирует последовательности с большой точностью, кроме того, он описывает функции продуктов, которые не описаны в gb.