*Вся работа проводилась в /nfs/srv/databases/ngs/margarita
- Был выполнен контроль качества чтений с помощью программы FastQC.
Команда: fastqc chr15.fastq
Результат выполнения доступен по данной ссылке: chr15_fastqc.html Далее был произведен анализ качества чтений на основании полученных данных и примеров, которые можно видеть, перейдя по ссылкам ниже:
Пример плохой последовательности:
http://www.bioinformatics.babraham.ac.uk/projects/fastqc/bad_sequence_fastqc.html
Пример хорошей последовательности:
http://www.bioinformatics.babraham.ac.uk/projects/fastqc/good_sequence_short_fastqc.html
Общие сведения:
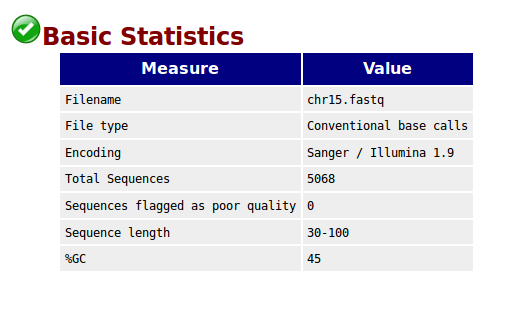
В графе basic statistics можно видеть, что число чтений 5068, GC% равен 45,распределение Per sequence GC content сильно отличается от кривой гаусса, что представляет большую проблему.
- Очистка чтений с помощью программы Trimmomatic
http://www.usadellab.org/cms/uploads/supplementary/Trimmomatic/TrimmomaticManual_V0.32.pdf
Команды:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 chr15.fastq chr15_trim.fastq TRAILING:20 MINLEN:50
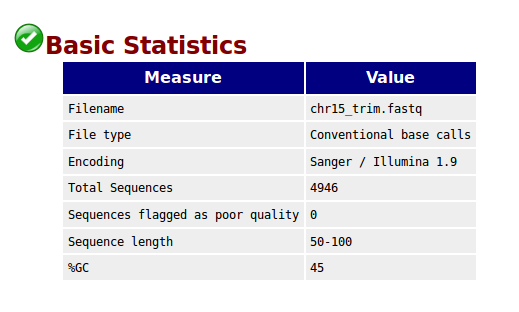
Можно сказать, что, во-первых, trimmotanic сработал, поскольку последовательностей стало меньше(4946) и они стали длины от 50 до 100(были от 300 до 100). Далее можно заметить, что BoxWhisker plot стал выгядеть намного лучше, в частности поднялось среднее(синяя линия) и усы перестали вылезать в красную область.
Также необходимо заметить наличие overrepresented sequences(шесть последовательностей повторяются по 5 раз), обусловленное отрезанием концов от последовательностей.
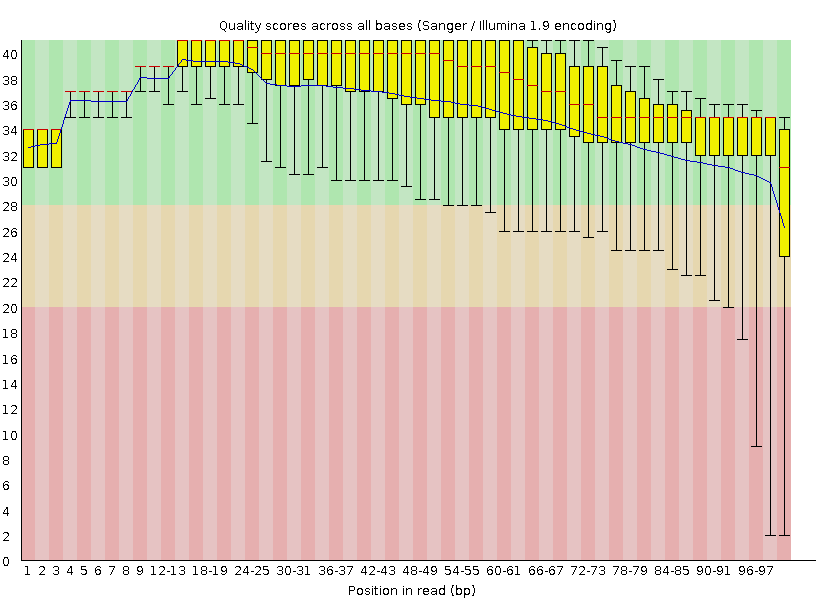
Pic 1: per base quality
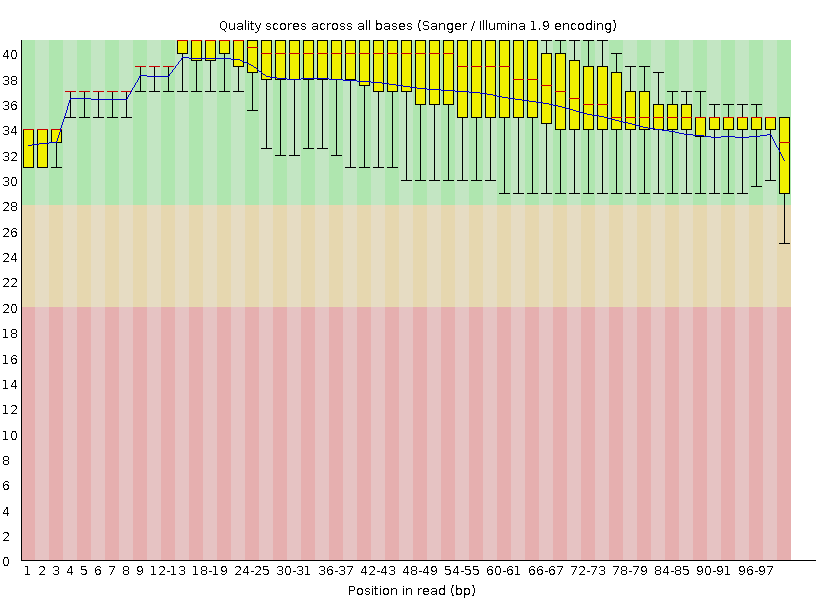
Pic 2: per base quality after trimmotanic
Расшифорвку всех графиков я взяла здесь:
http://www.bioinformatics.babraham.ac.uk/projects/fastqc/Help/3 Analysis Modules/
- Очищенные чтения были откартированы с помощью программы BWA.
Команды:
bwa index chr15.fasta bwa mem chr15.fasta chr15_trim.fastq align.sam
Далее был проведе анализ выравнивания:
samtools view -b -o samtools_align.bam align.sam
samtools sort -T /tmp/aln.sorted -o aln.sorted.bam samtools_align.bam
samtools index aln.sorted.bam
samtools idxstats aln.sorted.bam > idxstats_sorted.txt
chr15<------>102531392<------>4946<------>0
На данной табличке видно, что откартировалось 4946 чтений(все), длина 15 хромосомы 102531392.
Дополнительное задание: Найти один экзон и определите его среднее покрытие чтениями.
samtools depth aln.sorted.bam > depth.txt
Далее был выбран один из хорошо покрытых прочтениями нуклеотидов - нуклеотид 77756460 с покрытием 29.
В GenomeBrowser были определениы координаты участка, содержащего этот нуклеотид, после чего был перезапущен samtools depth,
с указанными границами экзона.
samtools depth aln.sorted.bam -r chr15:77712993-77777946 > depth_exon.txt
head -n 1 depth_exon.txt ; tail -n 1 depth_exon.txt
chr15<------> 77713424<------> 1
chr15<------> 77777946<------> 57
- Поиск SNP и инделей
Команды:
samtools mpileup -uf chr15.fasta aln.sorted.bam > aln.sorted.bcf
bcftools call -cv aln.sorted.bcf > compare_diff.vcf
Созданный файл содержит список отличий между референсом и чтениями. compare_diff.vcf
Всего найдено 92 SNP и 8 инделей. Описание трех полиморфизмов:
chr<------>coord. <------>ref <------>read <------>depth <------> quality
chr15<------> 58853793<------> G<------> T<------> 9.52546<------> DP=1
chr15<------> 89390006<------> A<------> G<------> 10.4247<------> DP=1
chr15<------> 89391160<------> C<------> A<------> 221.999<------> DP=24
DP - Raw read depth
- Анализ SNP С помощью программы annovar были проаннотированы только полученные snp.
Pipeline:
perl convert2annovar.pl -format vcf4 -outfile compare_diff.anv compare_diff.vcf
cat compare_diff.anv > compare_diff_1.anv
#В файле compare_diff_1.anv были вручную удалены индели.
Команды: perl /nfs/srv/databases/annovar/annotate_variation.pl -geneanno -dbtype refGene -buildver hg19 compare_diff_1.anv -outfile chr5_refgene /nfs/srv/databases/annovar/humandb/ perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype snp138 -buildver hg19 compare_diff_1.anv -outfile chr5_snp138 /nfs/srv/databases/annovar/humandb/ perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype 1000g2014oct_all -buildver hg19 compare_diff_1.anv -outfile chr5_1000g2014oct /nfs/srv/databases/annovar/humandb/ perl /nfs/srv/databases/annovar/annotate_variation.pl -regionanno -dbtype gwasCatalog -buildver hg19 compare_diff_1.anv -outfile chr5_gwas /nfs/srv/databases/annovar/humandb/ perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype clinvar_20150629 -buildver hg19 compare_diff_1.anv -outfile chr5_clinvar /nfs/srv/databases/annovar/humandb/ Rs имеют 76 полиморфизмов из 90. Были обнаружены 34 het замены и 53 hom. Встречаются следующие категории: intronic (интронные области) - 67, exonic (экзонные области) - 12, upstream - 3 , intergenic (между генов), ncRNA-intronic( non-coding RNA интронные) - 10, UTR3( 3' нетранслируемые регионы) - 1. Регионы, которые в принципе есть, но у меня нет: UTR5 (5' нетранслируемые регионы) downstream (регион в 1kb у начала транскрипции) splicing intergenic О частоте найденных snp: От 0.3 до 0.8 примерно, есть очень редкие гены с примерной частотой 0.009. SNP попали в гены: LIPC APQ9 HMG20A LOC101928694 ACAN Некоторые нуклеотидные замены привели к аминокислотным заменам. Полный список данных замен вы можете видеть в файле chr5_refgene.exonic_variant_function. Сводная таблица всех snp и их характеристик. Таблица Для spn попавших в экзоны (таких было 12) была сделана сводная таблица
Про покрытие можно сказать, что оно в основном больше 10. Про клиническую аннотацию: В chr5_gwas.hg19_gwasCatalog попало 5 snp связанных с холестеролом, диабетом и ростом. Для базы данных Clinvar файл chr5_clinvar.hg19_clinvar_20150629_dropped оказался пустым. В файле chr15_refgene.exonic_variant_function приведены синонимичные(8) и несинонимичные(4) замены, к которым привели SNP.