Отчет
Задание 1
C использованием инструментов пакета 3DNA построена A-, B- и Z-форма дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность GATC. Структуры дуплексов сохранены соответственно в файлах gatc-a.pdb, gatc-b.pdb и gatc-z.pdb с помощью следующих команд:
fiber -a gatc-a.pdb
fiber -b gatc-b.pdb
fiber -z gatc-z.pdb
Задание 2
Упражнение 1
С помощью программы Jmol выделим в А-структуре следующие атомы и химические группировки:
сахарофосфатный остов ДНК;
все нуклеотиды;
все аденины;
атом N7 во всех гуанинах;
атом N7 в первом по последовательности гуанине.
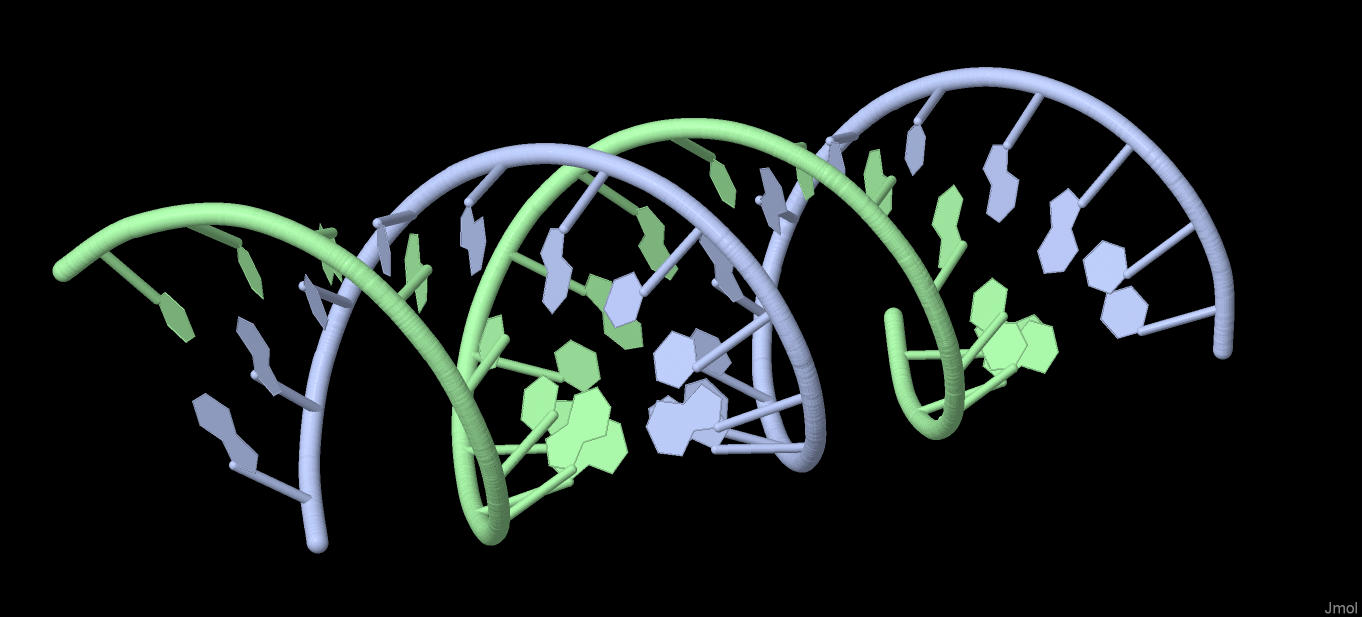
Рис. 1 Cахарофосфатный остов ДНК
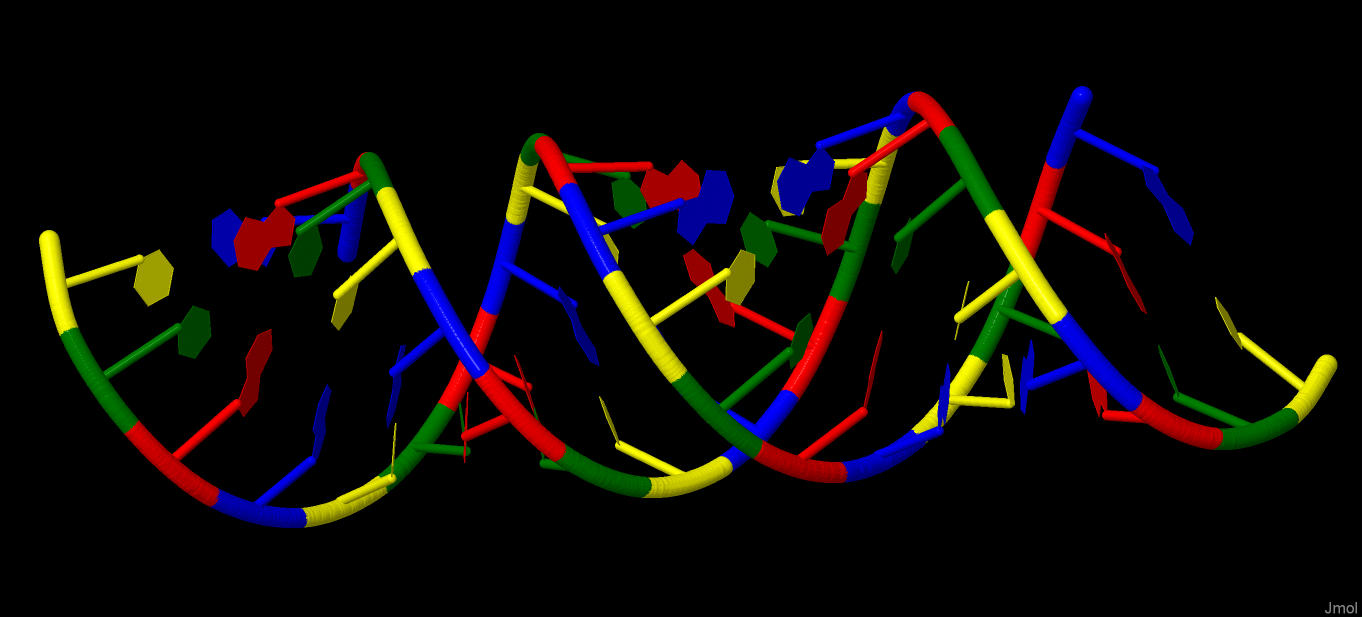
Рис. 2 Все нуклеотиды

Рис. 3 Все аденины
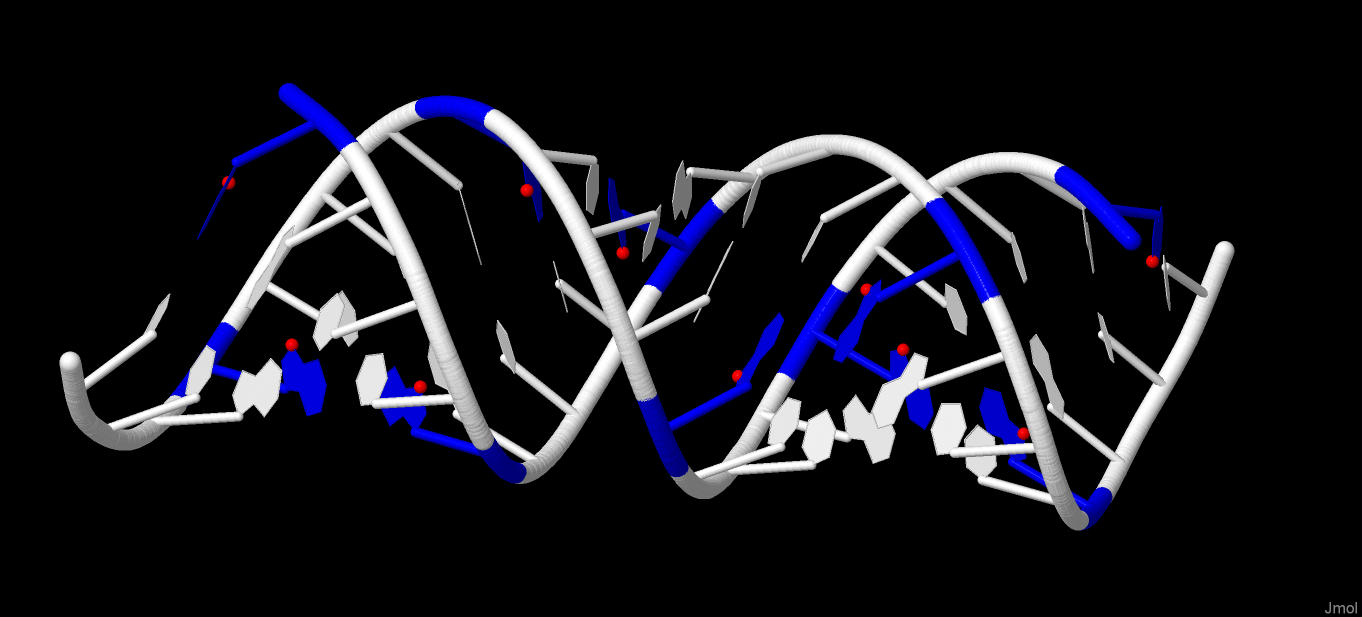
Рис. 4 Атом N7 во всех гуанинах
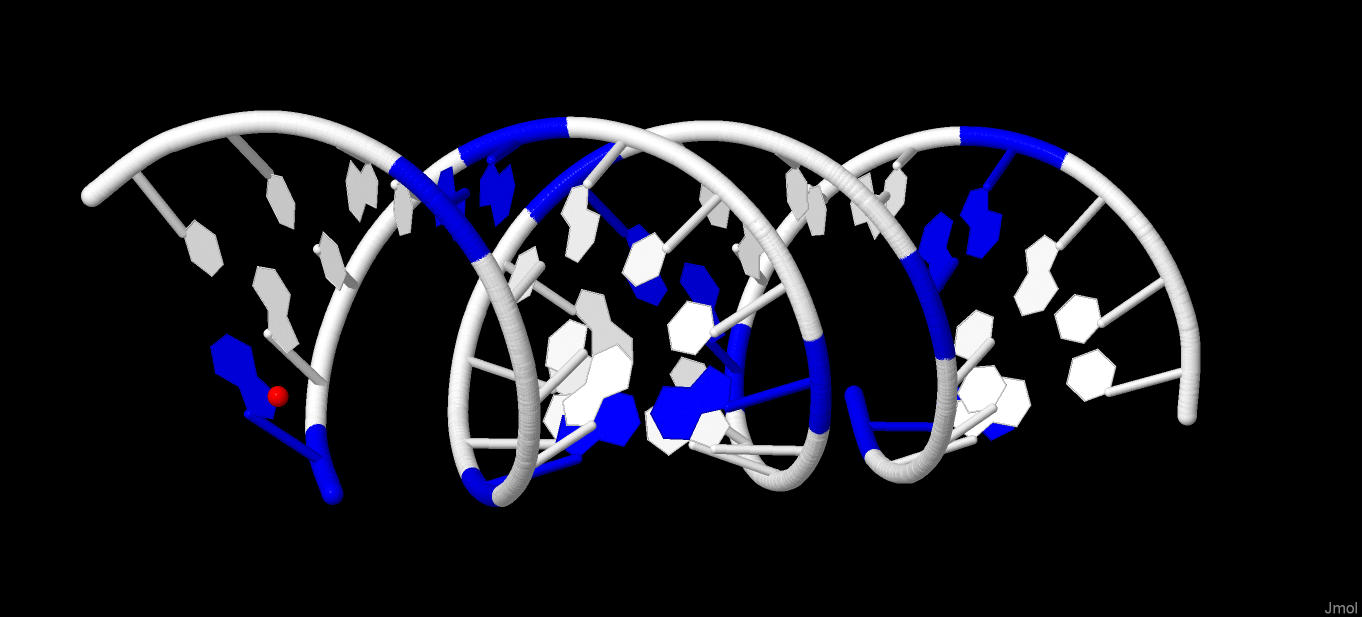
Рис. 5 Атом N7 в первом по последовательности гуанине
Упражнение 2
Получены файлы PDB: 1H4S - тРНК и 1MHD- ДНК.
Упражнение 3
Проверены заданные структуры ДНК и РНК на наличие разрывов.
Для этого структуры 1H4S и 1MHD были внимательно рассмотрены с помощью Jmol.Разрывов не было найдено.
Координаты атомов только ДНК и РНК были сохранены с помощью команды write RNA.pdb в консоли Jmol.
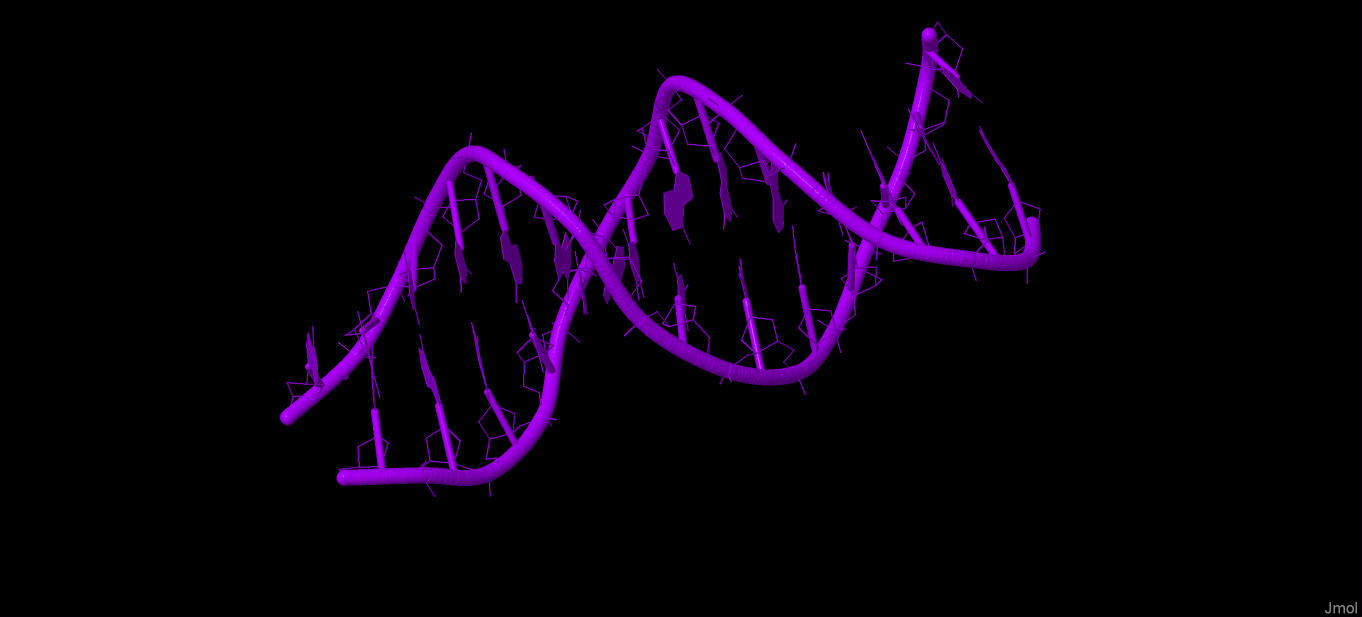
Рис. 6 Изображение ДНК
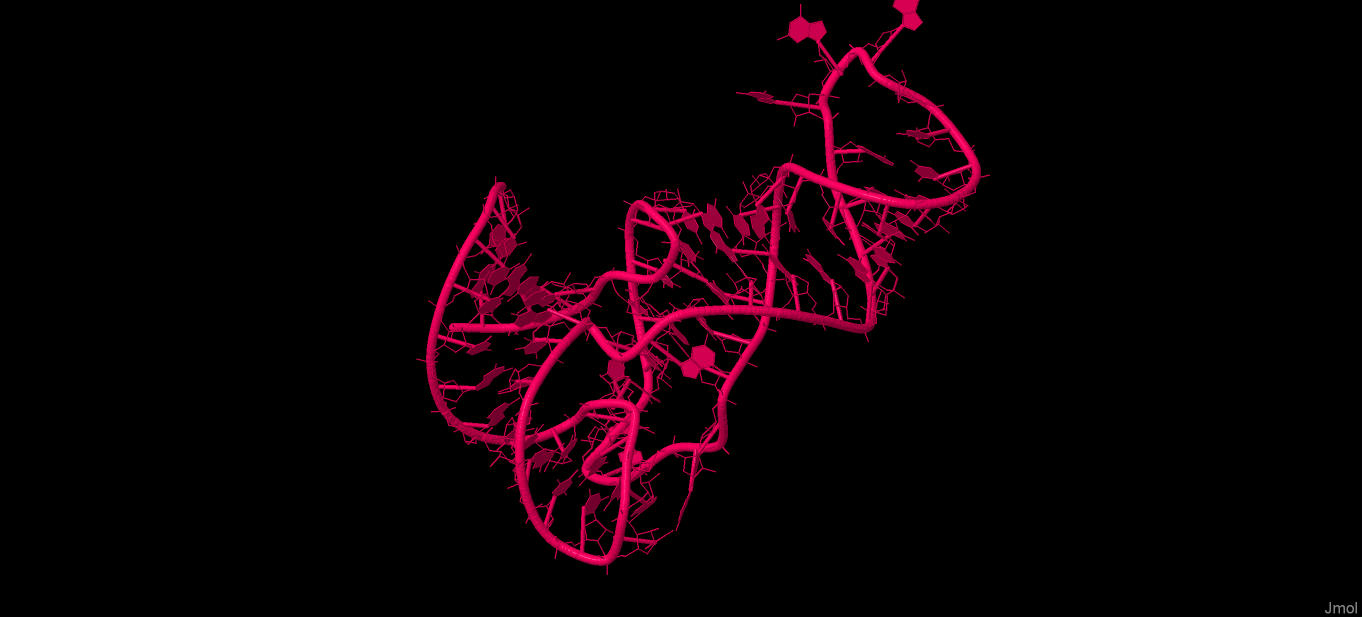
Рис. 7 Изображение РНК
Задание 3
Упражнение 1
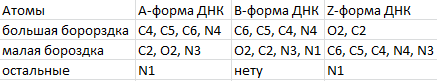
Для А, В и Z форм ДНК были найдены большие и малые бородки
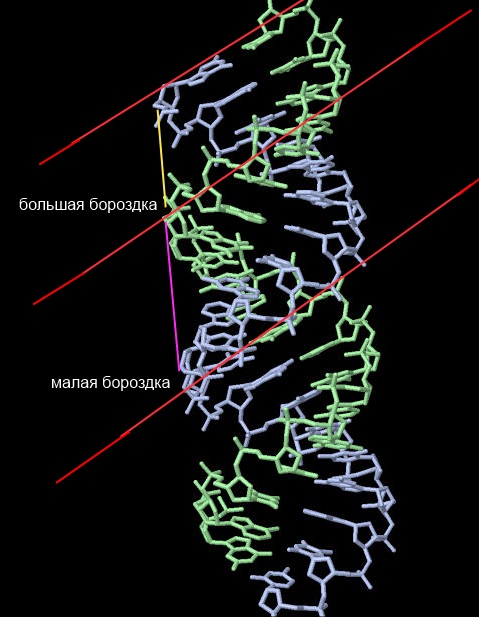
Рис. 8 Большая и малая бороздка А-ДНК
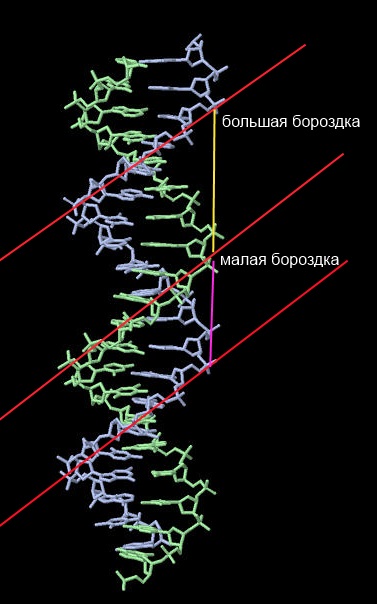
Рис. 9 Большая и малая бороздка В-ДНК
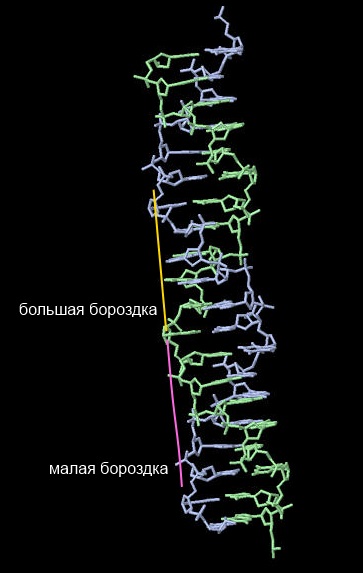
Рис. 10 Большая и малая бороздка Z-ДНК
Далее был выбран цитозин и для него было определено, какие атомы основания явно обращены в сторону большой бороздки, а какие в сторону малой.
Синим отмечены атомы, смотрящие в сторону малой бооздки, красным - атомы, смотрящие в сторону большой бороздки.
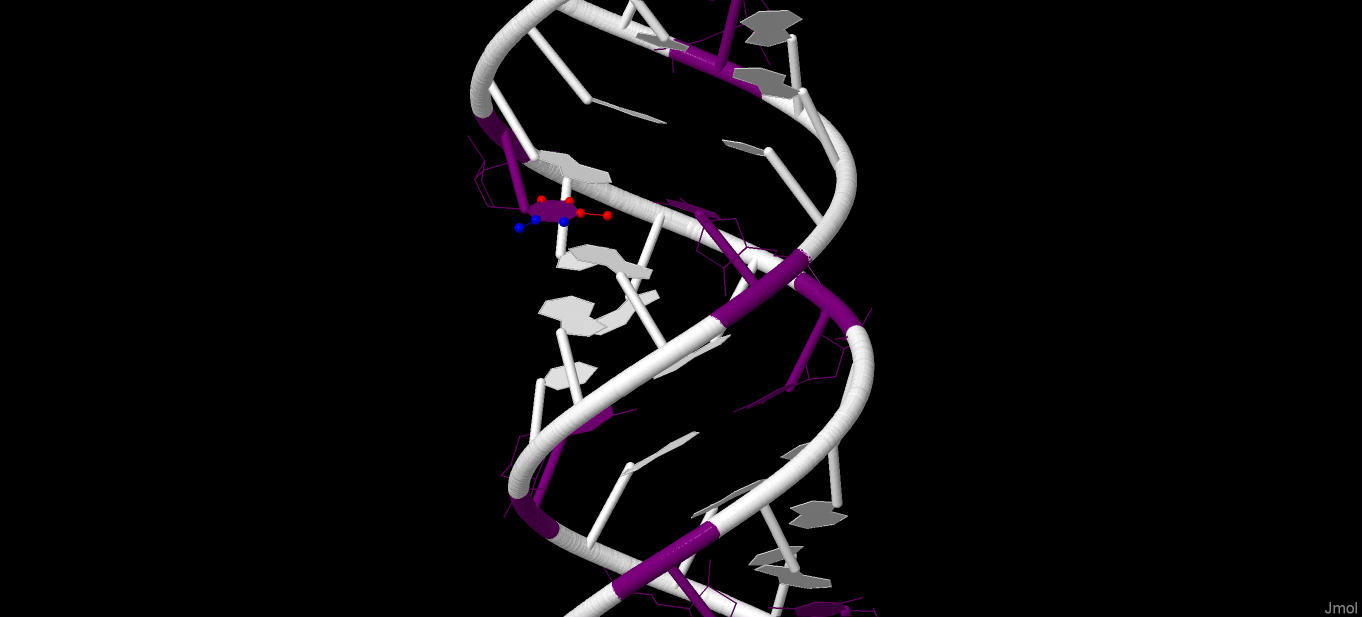
Рис. 11 Цитозин в A-ДНК
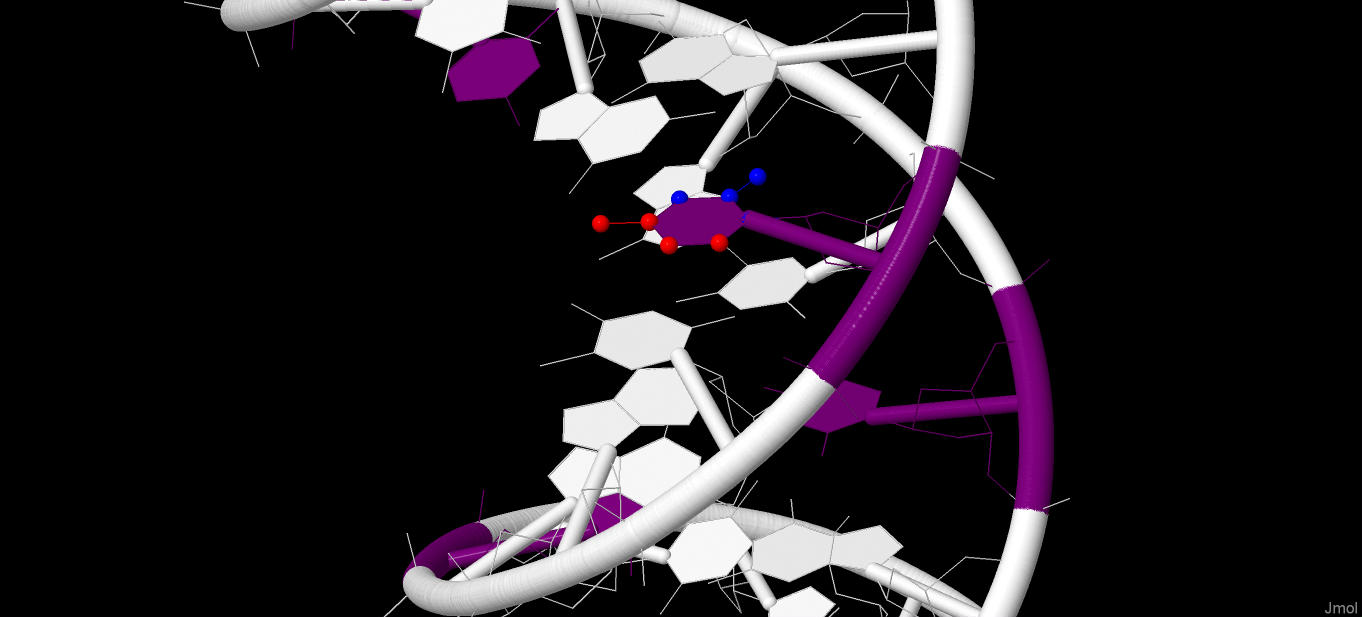
Рис. 12 Цитозин в В-ДНК
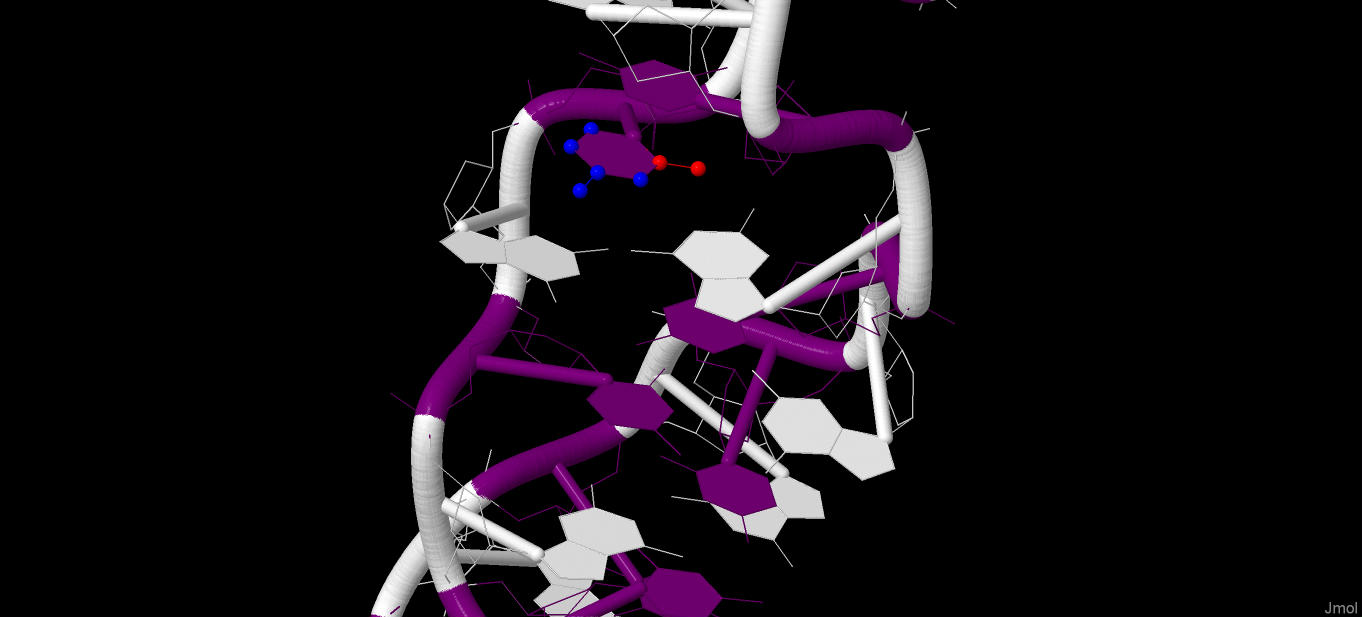
Рис. 13 Цитозин в Z-ДНК
Проект Chemsketch
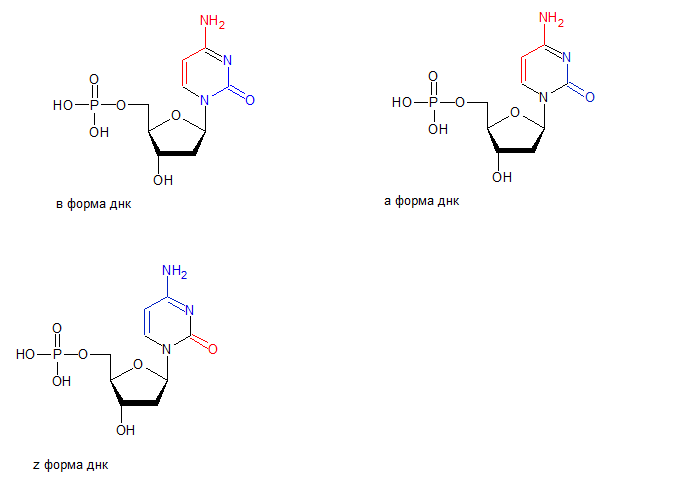
Таблица 1 - ориентация атомов цитозина в ДНК
Упражнение 2
Сравнены основные спиральные параметры разных форм ДНК.
Таблица 2 - основные спиральные параметры разных форм ДНК
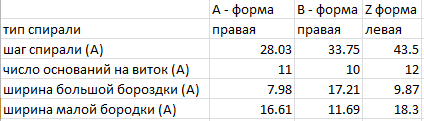
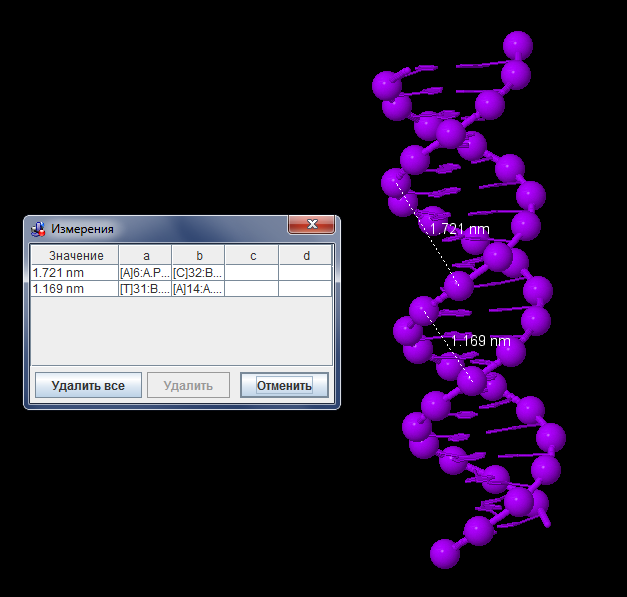
Рис. 14 Проведение измерений ширины бороздок
Упражнение 3
Торсионные углы были померяны в соответствие с этой схемой:

Рис. 15 Проведение измерений торсионных углов
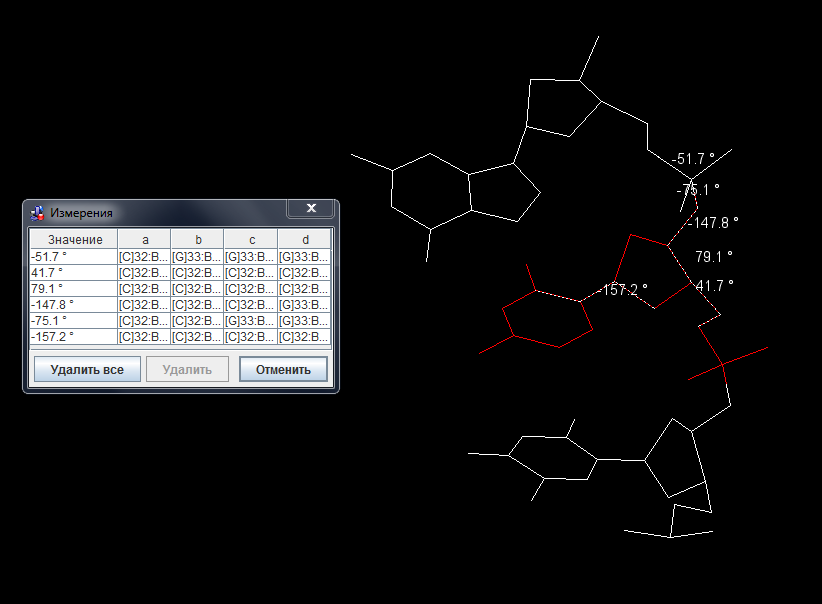
Результаты:
Таблица 3 - торсионные углы
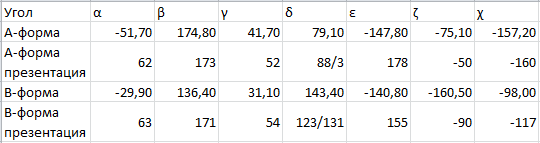
Как видно из таблицы, померянные значеня торсионных углов сильно отличаются от теоретических значеный. Это может быть обьяснено тем, что в презентации даны усредненные значения измерений большого количества нуклеотидов.
Задание 4
Упражнение 1
Для каждого из файлов gatc-a.pdb, gatc-b.pdb и gatc-z.pdb были выполнены следующие команды:
remediator --pdb --old XXXX.pdb > XXXX_old.pdb
find_pair -t XXXX_old.pdb
find_pair -t XXXX_old.pdb stdout | analyze
В результате для каждой структуры создан ряд файлов с описанием разных её параметров(gatc-a.out, gatc-b.out и gatc-z.out). В файлах gatc-a.out, gatc-b.out и gatc-z.out можно найти значения торсионных углов.
Для файла 1MDH.pdb были проделаны команды:
remediator --pdb --old 1MDH.pdb > 1MDH_old.pdb find_pair -t 1MDH_old.pdb stdout | analyze
В файле 1MDH_old.out можно найти информацию о торсионных углах.
Таблица торсионных углов была обработана и представлена в формате csv.
Таблица 1 Таблица 2При сравнении значений соответствующих торсионных углов в структурах A-, B- и Z-форм ДНК было установлено, что в наибольшей степени различаются значения следующих углов: у A- и B-форм - δ и χ; у A- и Z-форм - α; у A- и Z-форм - α, ξ и χ.
Для файла 1H4S.pdb были проделаны команды:
remediator --pdb --old 1H4S.pdb > 1H4S_old.pdb
find_pair -t 1H4S_old.pdb stdout | analyze
В файле 1H4S_old.out содержится информация о торсионных углах.
Таблица торсионных углов была обработана и представлена в формате csv.
Strand 1 1H4S Base alpha beta gamma delta epsilon zeta chi 1 G --- 146.00 56.00 87.30 -171.70 -60.30 -153.60 2 G 141.60 -174.70 -178.40 80.60 -135.90 -65.90 -172.60 3 A -54.10 163.80 53.20 75.60 -166.30 -97.00 -168.10 4 G 156.50 -150.00 -172.90 128.70 --- --- -159.40 5 G --- 146.70 51.60 84.10 -134.30 -73.10 177.10 6 C -65.60 165.80 53.80 81.90 -154.70 -73.90 -171.90 7 U -66.30 177.60 55.70 83.30 -157.00 -13.00 -153.80 8 G -137.70 79.10 173.70 88.70 -128.90 -83.00 175.60 9 G 140.90 -141.80 176.10 81.00 -129.10 -79.40 174.00 10 u -62.10 176.50 45.70 78.80 -128.10 -73.10 -162.60 11 P -52.90 163.70 50.70 78.60 --- --- -149.00 12 A --- -103.90 179.00 88.80 -147.20 -70.90 -170.60 13 C -67.20 178.10 47.80 80.00 -160.60 -75.00 -152.90 14 G -66.80 174.40 51.10 82.10 -154.40 -76.40 -156.90 15 A -63.60 166.50 50.40 81.70 -152.60 -72.50 -159.00 16 G -51.30 165.90 52.40 79.10 -162.90 -57.60 -167.90 17 G 160.40 -172.10 179.80 83.80 -142.20 -79.00 179.10 18 G -63.60 170.80 46.70 78.50 --- --- -163.00 19 G --- -128.90 71.00 91.60 -153.10 -68.40 -171.90 20 C -70.20 -179.40 46.60 82.00 -155.00 -69.30 -160.60 21 G -59.00 178.10 43.90 78.60 -159.50 -73.40 -154.60 22 C 148.90 -155.60 -174.50 85.00 -141.50 -77.50 -174.10 23 A -71.40 167.80 64.30 75.70 -152.60 -61.50 -174.00 24 G -59.70 177.20 54.10 87.70 --- --- -150.90 25 G --- -158.50 46.50 126.70 --- --- -71.50 average -13.16 54.16 44.43 84.17 -148.21 -70.52 -103.83 Strand 2 1H4S 1 C -63.80 170.50 54.80 81.40 --- --- -154.80 2 C -68.20 179.30 46.30 80.10 -151.70 -73.50 -156.10 3 U -64.60 -170.00 50.00 84.80 -166.20 -73.70 -158.30 4 C -62.10 177.20 51.60 80.30 -157.60 -61.10 -171.40 5 U -74.00 171.30 59.20 82.30 -152.70 -79.90 -168.30 6 G -62.50 160.50 63.50 79.20 -156.20 -76.50 -168.60 7 A -64.50 177.50 52.10 80.40 -146.10 -72.50 -163.50 8 C -65.50 -178.80 49.90 83.30 -151.40 -72.90 -164.40 9 C "--- " -136.80 51.90 81.70 -156.20 -73.60 -166.80 10 G --- -146.40 56.50 139.20 --- --- -66.70 11 G --- 146.10 145.40 132.30 --- --- -135.10 12 U -71.70 171.90 61.30 79.50 --- --- -157.20 13 G -72.70 164.20 63.80 80.70 -159.60 -67.80 -161.10 14 C -70.50 177.30 51.70 80.20 -153.30 -71.70 -152.70 15 U -64.30 -174.80 48.90 80.80 -152.40 -69.80 -170.10 16 C -70.50 -168.80 47.70 82.90 -154.10 -68.80 -166.40 17 C -61.40 176.60 46.20 81.70 -154.10 -73.20 -161.00 18 A -59.90 173.80 49.70 84.80 -142.60 -68.90 -162.90 19 C -67.10 171.60 52.10 80.90 -145.30 -66.90 -162.20 20 G -70.50 177.80 51.60 82.50 -142.30 -72.10 -166.60 21 C -75.40 178.30 53.80 79.20 -158.50 -76.30 -160.10 22 G --- 159.30 57.30 82.10 -160.80 -75.60 -164.80 23 U --- -144.80 65.80 83.90 --- --- -167.80 24 C --- 134.30 51.00 129.40 --- --- -138.50 25 C --- -167.80 59.70 85.30 --- --- -162.60 average -67.38 68.55 57.71 88.36 -153.39 -71.93 -156.98Таблица 3 Таблица 4 Структура РНК больше всего похожа на A-форму ДНК, если рассматривать значения торсионных углов.
Упражнение 2
Рассмотрим каноническую структуру trna.
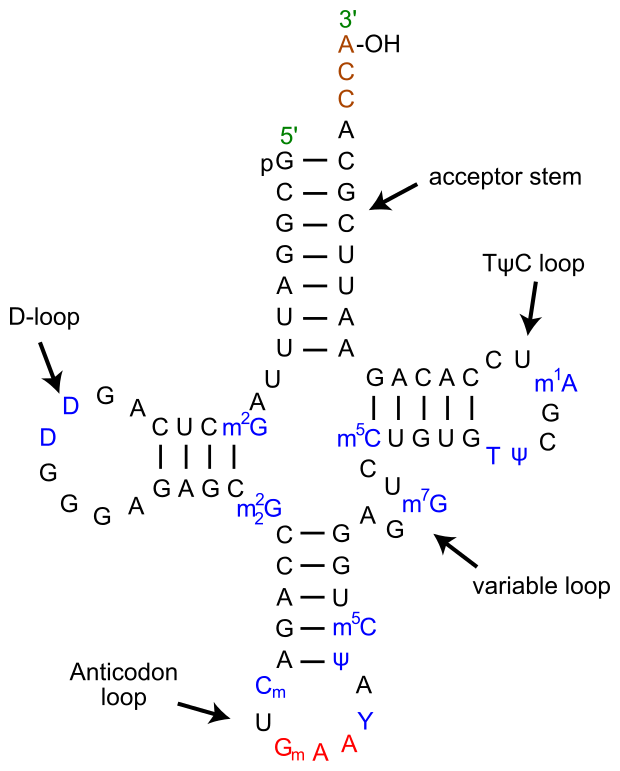
Рис 16 Каноническая структура t rna
Номера нуклеотидов, образующих стебли во вторичной структуре тРНК показаны зеленым
На рис. 17 сверху вниз в зеленых рамках идут - акцепторный стебель, T стебель, антикодоновый стебель, D стебель.
В тРНК образуется много неканонических пар оснований.
Неканонические пары оснований в структуре тРНК показаны розовым
Дополнительная водородная связь в тРНК, стабилизирующая ее третичную структуру - псевдоузел - показана желтым
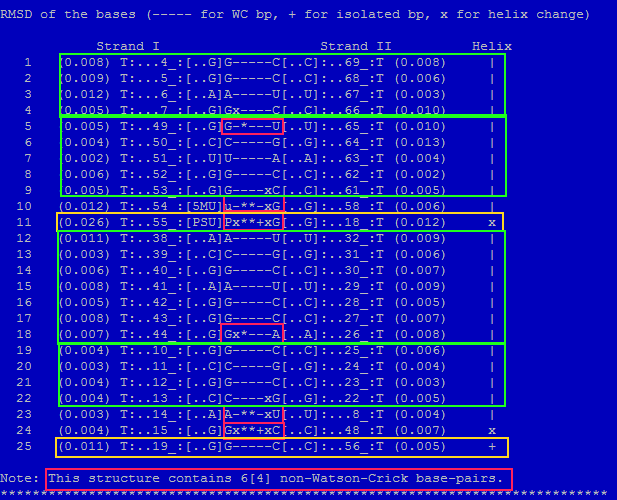
Рис. 17 Структура РНК
Упражнение 3
Далее были рассмотрены возможные стекинг-взаимодействия.
Для этого в файле RNA.out были найдены данные о площади перекрывания 2х пар азотистых оснований.
В рамочках выделены минимальная и максимальная площади перекрывания.
Некоторые пары вообще не перекрываются из-за того, что они принадлежат различным областям t rna.
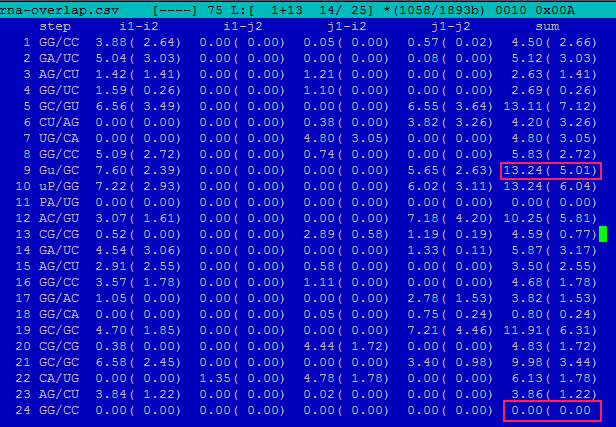
Рис. 18 Перекрывание пар азотистых оснований
Отмеченные пары были выбраны для дальнейшего анализа.
Для данных пар из step 10 и step 24 были выполнены следующие команды:
ex_str -10 stacking.pdb step10.pdb
stack2img -cdolt step10.pdb step10.ps
convert -scale 30% step10.ps step10.png
Далее представлены изображения, полученные с помощью stack2img и Jmol.
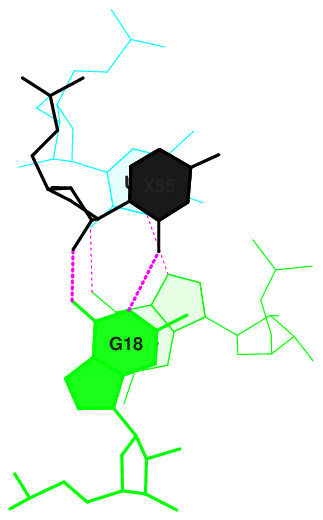
Рис. 19 Максимальное перекрывание
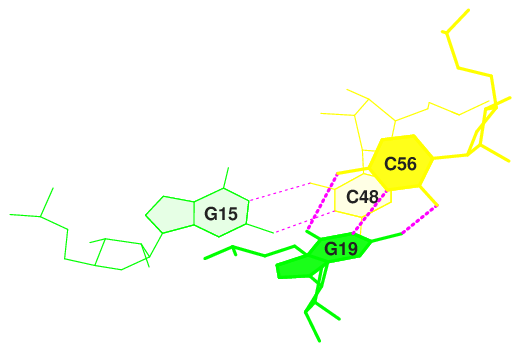
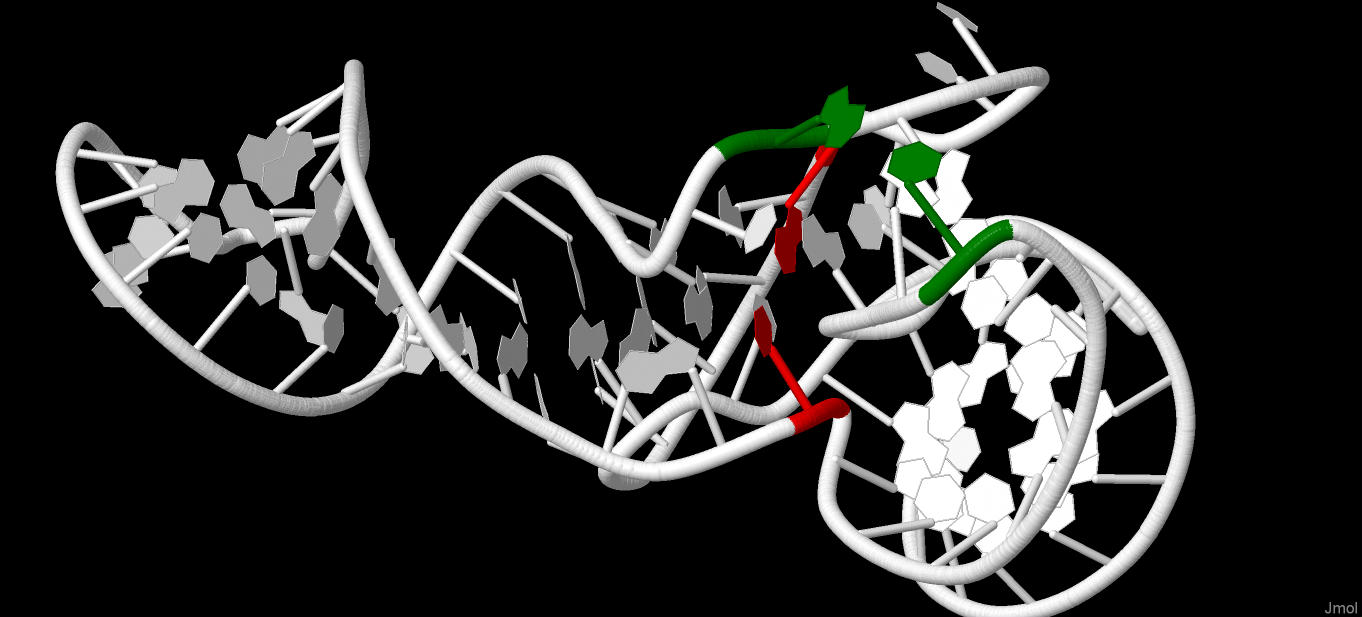
Рис. 20 Минимальное перекрывание