1. Blastn - установление таксономии и гена организма
Для анализа была взята последовательность forward_clean.fasta
Ген принадлежит организму Ophiopholis aculeata.

Рис 1 Внешний вид Ophiopholis aculeata
Был установлен таксон организма - Ophiopholis aculeata
Ophiopholis aculeata - морская звезда, по-другому называемая mottled brittle star.
Небольшая по размерам морская звезда, она любит жить под камнями и быстро двигается на своих тонких щупальцах в случае опасности.
Полная таксономия представлена ниже:
Eukaryota; Metazoa; Echinodermata; Eleutherozoa; Asterozoa;
Ophiuroidea; Ophiuridea; Ophiurida; Ophiurina; Gnathophiurina;
Gnathophiuridea; Ophiactidae; Ophiopholis.
По результатам поиска в blast можно заключить, что ген кодирует субьединицу цитохром оксидазы
Лучшие находки:
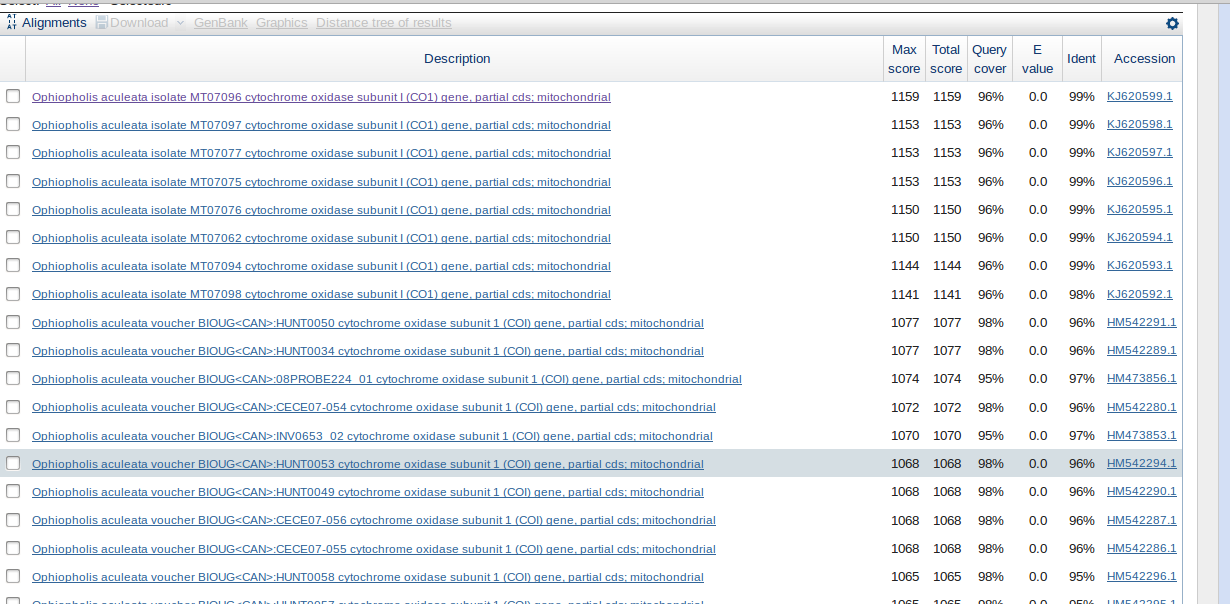
Рис 2 Результат поиска с blast
Найбольшее совпадание обнаружилось с Ophiopholis aculeata isolate MT07096 cytochrome oxidase subunit I (CO1) gene, partial cds; mitochondrial
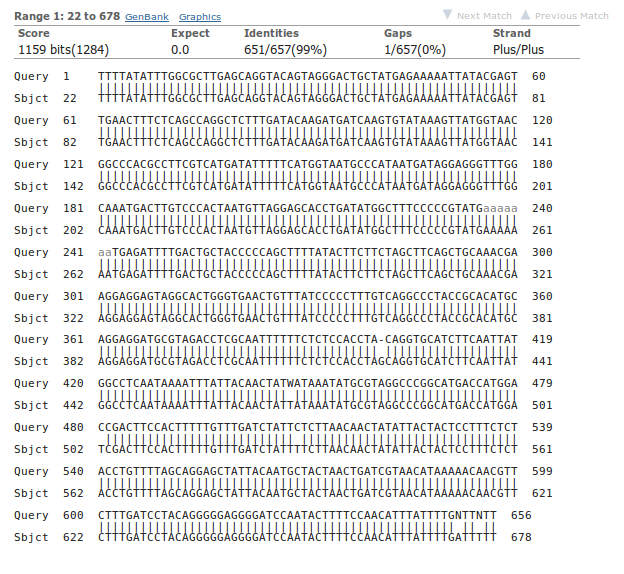
Рис 3 Лучшее выравнивание
2. Сравните списки находок нуклеотидной последовательности 3-я разными алгоритмами blast
Последовательность - из практикума 7
Область поиска ограничена таксоном Amphipholis squamatа. Поиск по базе даных refseq.
blastn:
Дает 16 находок, виден четкий порог между хорошими находками с E value 3e-118 и плохими находками с E value 1.8. Находки заметно отличаются по Query cover и E value.
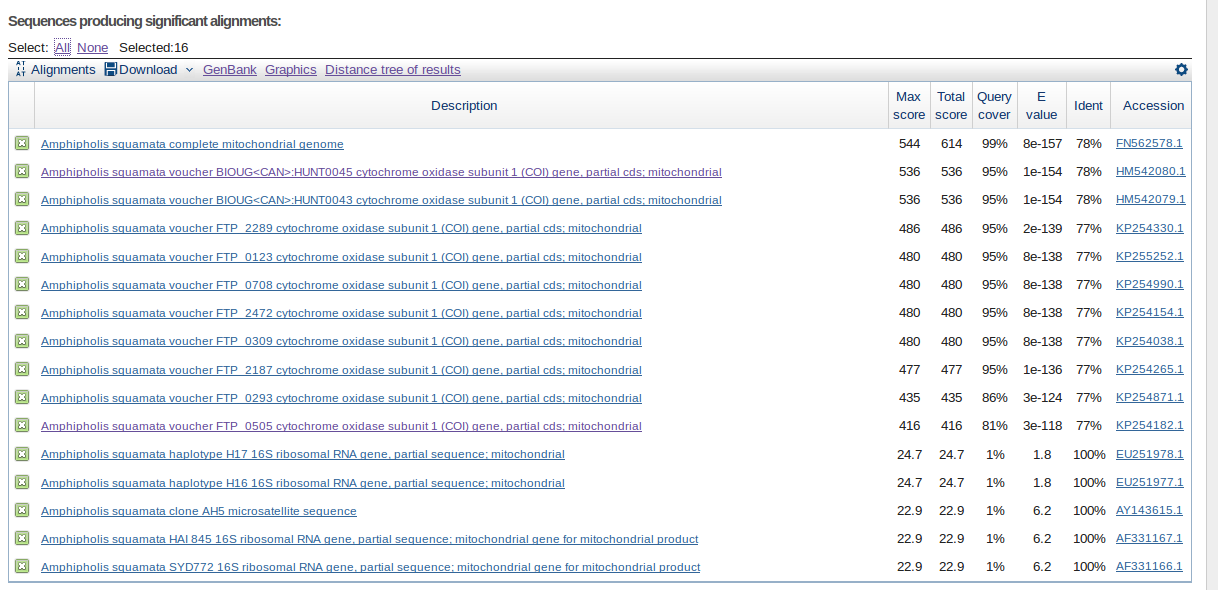
Рис 4 Поиск с помощью blastn
discontiguous megablast:
Дает 11 находок, все из них есть в находках nblast, Query cover и E value различаются не сильно. Заметим, что в discontiguous megablast параметр Word size равен 11.
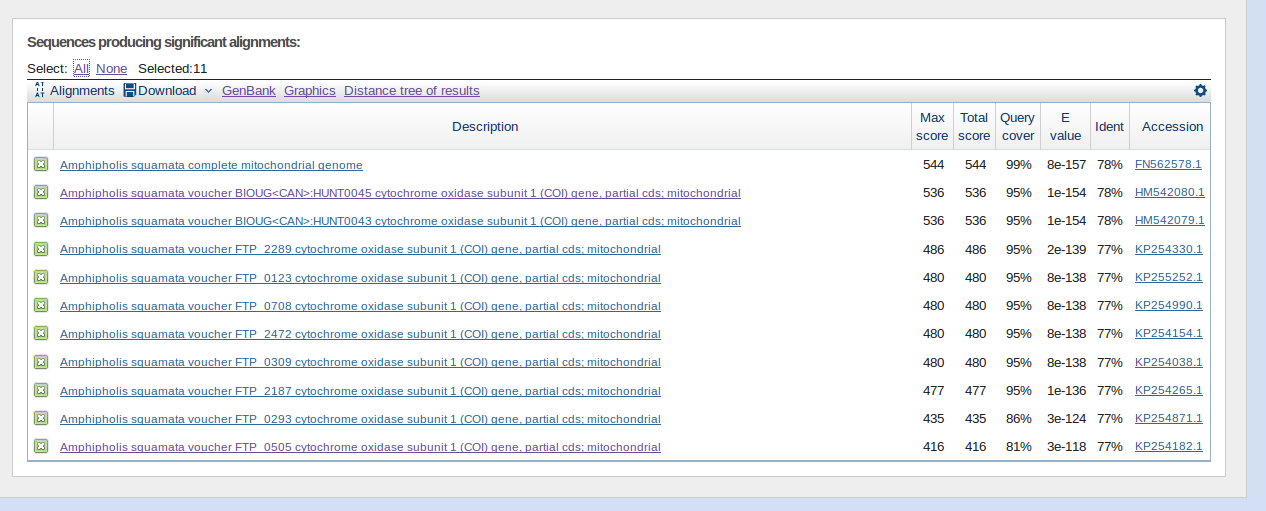
Рис 5 Поиск с discontiguous megablast
megablast:
Дает 8 находок, все из них есть в находках discontiguous megablast, параметр Word size равен 28, поэтому не были найдены 3 последовательности, которые нашел discontiguous megablast.
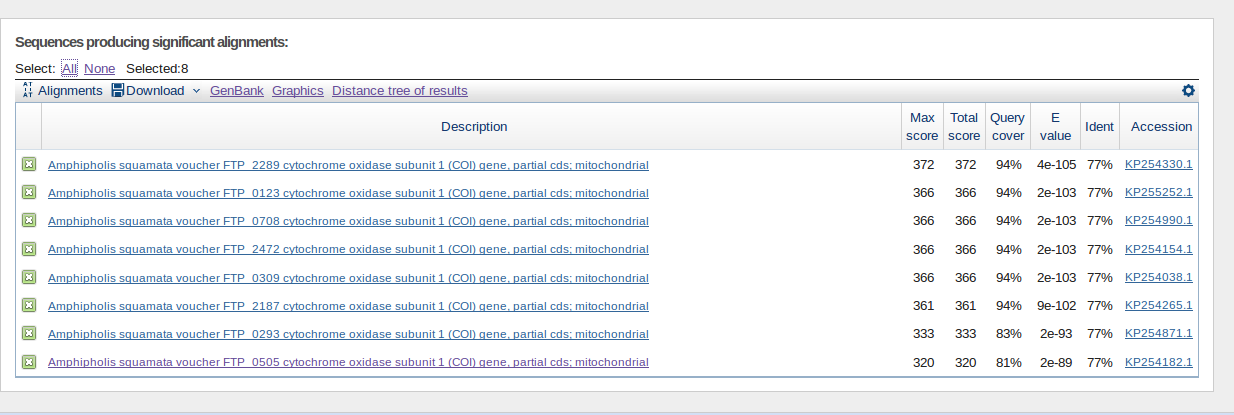
Рис 5 Поиск с megablast
Если в megablast подставить параметры Word size 16 и match/mismatch scores 2,-3, то выдаются 11 результатов из discontiguous megablast c меньших max score и большим E value.
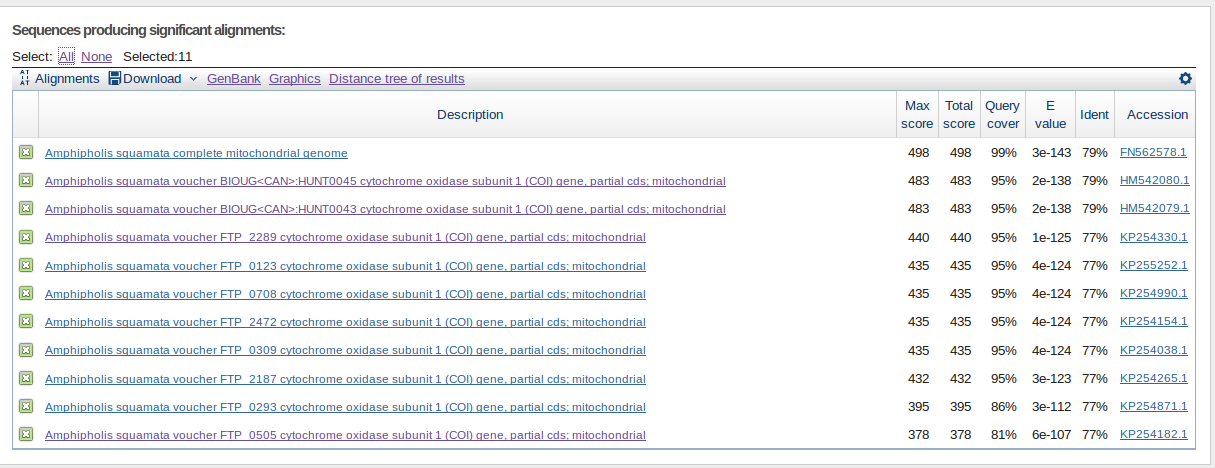
Рис 6 Поиск с megablast с измененными параметрами
3. Проверьте наличие гомологов пяти белков в геноме одного из организмов
Везде брались файлы Isoform 1.
Белок HSP7C_HUMAN: Шаперон - белок теплового шока, компонент PRP19-CDC5L комплекса который формирует часть сплайсосомы и необходим для активации пре-мРНК сплайсинга. Подробнее[(здесь]http://www.uniprot.org/uniprot/P11142#sequences)
Был произведен поиск tblastn, база данных refsec nucleotide, организм Caenorhabditis elegans.
Хороших находок в геноме 6(хромосомы 1-5 и Х).
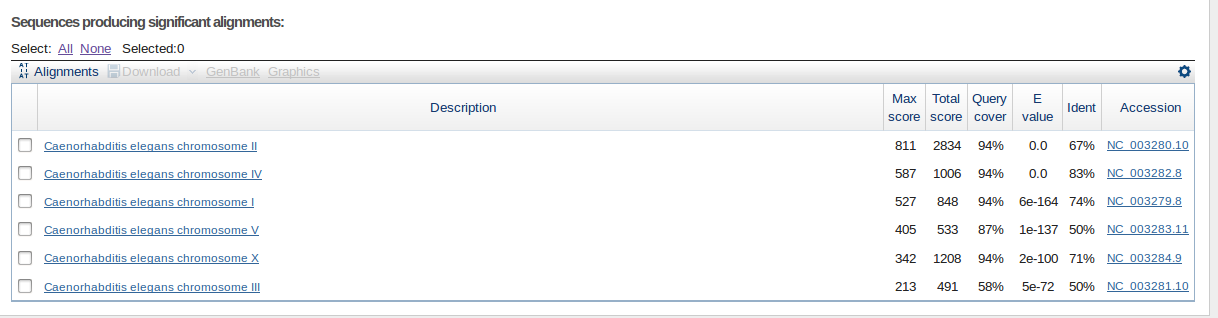
Рис 7 Поиск с помощью tblastn
Если посмотреть на Е value и Query cover, то можно сказать что лучшие находки это первая и вторая, хотя остальные тоже очень хороши по Е value.
Это обьясняется тем, что белки теплового шока жизненно необходимы клетке.
Белок TERT_HUMAN:
Каталитическая субьединица теломеразы (митохондриальная). Подробнее здесь
Blast не нашел этого белка в данном организме. Возможно черви используют какой-то другой белок.
Белок CISY_HUMAN:
Цитрат синтетаза. Катализирует реакцию:
Acetyl-CoA + H2O + oxaloacetate = citrate + CoA Подробнее здесь:
Найдено одно достаточно хорошее совпадение:
Caenorhabditis elegans chromosome III
Рис 8 Хромосома 3 Caenorhabditis elegans
Белок RPB1_HUMAN:
Субьединица ДНК-зависимой РНК-полимеразы. Подробнее здесь:
Найдено 2 хороших совпадения: хромосома 2 и 4.
Рис 9 Хромосомы 2 и 4 Caenorhabditis elegans
Белок PABP2_HUMAN:
Полиаденилат-связивающий белок. Стимулирует поли(А)полимерзу (PAPOLA) пришивать поли(А) конец.
Подробнее здесь:
Найдено 3 совпадения: 1, 2 и 4 хромсомы. Совпадения не очень хорошие по Query cover. Рис 10 Хромосомы 1, 2 и 4 Caenorhabditis elegans
Поскольку у нас лучшая находка в каждом случае является целой хромосомой, то конечно они все аннотированы.
4. Классификация геномов родственных вирусов по сходству последовательностей
Были выбраны 5 геномов:1) Tomato leaf curl Barka virus KF260965.12) Tomato enation leaf curl virus KP195260.13) Tomato yellow leaf curl Axarquia virus KM215610.14) Tomato yellow leaf curl Mali virus LM651400.15) Tomato leaf curl Comoros virus AJ865341.1
Далее на сервере кодомо с помощью blast+,а таакже с помощью программы revise_blast_7.py были выполнены следующие команды:
seqret @virus_seq.txtvirus_seq.fasta
# В файл virus_seq.fasta записаны последовательности с идентификаторами из файла @virus_seq.txt.
makeblastdb -in virus_seq.fasta -dbtype prot
# Создание базы данных из 5 геномов
tblastx -query virus_seq.fasta -db virus_seq.fasta -outfmt 7 > out_all.txt
# Выравнивание всех против всех
python revise_blast_7.py -i out_all.txt -o out_all.tsv
# Обработка таблицы
sort -k 11 -g -t$'\t' < out_all.tsv > e_value_all.tsv
# Сортировка таблицы по e_value
sort -k 12 -r -g -t$'\t' < out_all.tsv > bit_all.tsv
# Сортировка таблицы по bit_score
Далее были сравнены файлы e_value_all.tsv и bit_all.tsv.
Сортированная по bit_score таблица имеет соответствующие значения e_value.
( простите просто слова корреляция мы уже боимся )
Поэтому для дальнейшего анализа была взята она.
Я решила характеризовать сходство последовательностей по суммарному числу bit выравнивания.
Имеется в виду, что суммируется число bit для каждой находки для двух геномов.
Для этого я написала скрипт summ_bit.py, который выдает таблицу table.txt суммарных bit_score.
Из таблицы можно сделать вывод, что наибольший суммарный bit_score 13038.9
между LM651400(yellow leaf curl Mali virus) и KM215610(yellow leaf curl Axarquia virus).
Также если запустить tblastx c сайта, то можно увидеть вот такую карту локального выравнивания.
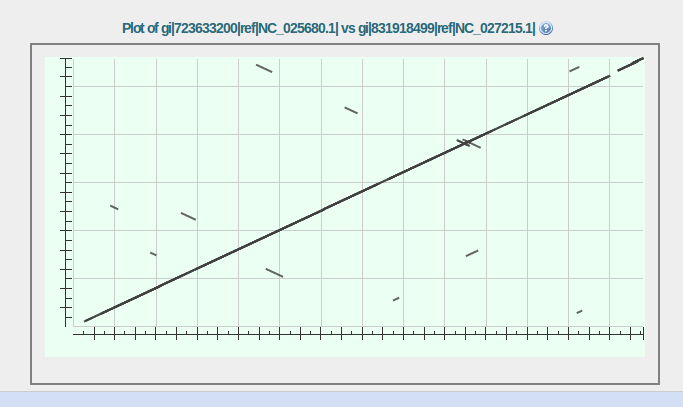
Рис 11 Карта локального выравнивания
На данной карте много инверсий (маленькие черточки перпендикулярные основной линии).
Также LM651400 проявляет почти такое же сходство с KF260965 и AJ865341.
Диагональные элементы по идее должны были быть нулевыми вследствие оработки скриптом revise_blast_7.py
и все-таки не равны нулю из-за присутствия repeat последовательностей, которые не затираются скриптом.