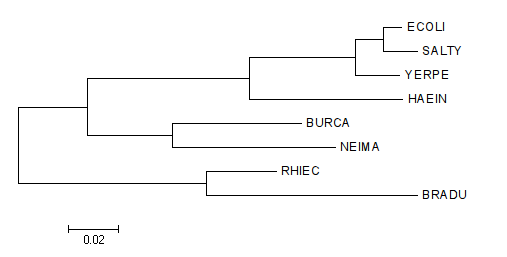
Цель - построить филогенетическое дерево тех же бактерий, что в предыдущих заданиях, используя последовательности РНК малой субъединицы рибосомы (16S rRNA).
В базе полных геномов NCBI были взяты следующие геномы:
Burkholderia_cenocepacia_AU_1054_uid58371
Rhizobium_etli_CFN_42_uid58377
Neisseria_meningitidis_053442_uid58587
Escherichia_coli_042_uid161985
Salmonella_enterica_serovar_Typhimurium_14028S_uid86059
Yersinia_pestis_A1122_uid158119
Haemophilus_influenzae_10810_uid86647
Bradyrhizobium_BTAi1_uid58505
Последовательности 16S РНК находятся в файле с расширением frn. Все последовательности собрала в единый файл в fasta-формате и выровняла с пом. Muscle в MEGA. Дерево построила методом “maximum likelyhood”.
Полученное дерево топологически не отличается от построенного по белкам.

Цель - найти в бактериях достоверные гомологи белка CLPX_ECOLI, посторить дерево и найти ортологи и паралоги.
Чтобы найти гомологов в заданных организмах, я создала файл clpx_ecoli.fasta и сделала базу данных для blastp из восьми “моих” белков. Файлы с протеомами я взяла из P:\y14\term4\Proteomes.
Сначала я собрала восемь протеомов “моих” бактерий в один файл:
cat name.fasta >> my_bacteria.fasta
Потом сделала базу данныхдля blastp:
makeblastdb -in Proteomes/my_bacteria.fasta -dbtype prot
Провела blast:
blastp -evalue 0.001 -query clpx_ecoli.fasta -db Proteomes/my_bacteria.fasta > output.txt
В файле output.txt содержится протокол по бласту, и есть табличка с параметрами находок:
Sequences producing significant alignments: Score E Value
sp|P0A6H1|CLPX_ECOLI ATP-dependent Clp protease ATP-binding sub… 860 0.0
sp|Q8ZRC0|CLPX_SALTY ATP-dependent Clp protease ATP-binding sub… 843 0.0
sp|Q8ZC66|CLPX_YERPE ATP-dependent Clp protease ATP-binding sub… 805 0.0
sp|Q1BH84|CLPX_BURCA ATP-dependent Clp protease ATP-binding sub… 621 0.0
sp|Q2K9U6|CLPX_RHIEC ATP-dependent Clp protease ATP-binding sub… 598 0.0
sp|Q89KG2|CLPX_BRADU ATP-dependent Clp protease ATP-binding sub… 598 0.0
sp|P44838|CLPX_HAEIN ATP-dependent Clp protease ATP-binding sub… 587 0.0
sp|Q9JTX8|CLPX_NEIMA ATP-dependent Clp protease ATP-binding sub… 557 0.0
sp|Q8ZJJ5|HSLU_YERPE ATP-dependent protease ATPase subunit HslU… 95.1 3e-21
sp|Q2KE54|HSLU_RHIEC ATP-dependent protease ATPase subunit HslU… 93.6 9e-21
sp|P0A6H5|HSLU_ECOLI ATP-dependent protease ATPase subunit HslU… 93.6 9e-21
sp|P43773|HSLU_HAEIN ATP-dependent protease ATPase subunit HslU… 92.0 4e-20
sp|Q89WN2|HSLU_BRADU ATP-dependent protease ATPase subunit HslU… 91.7 4e-20
sp|O30911|HSLU_SALTY ATP-dependent protease ATPase subunit HslU… 91.7 4e-20
sp|Q1BSM8|HSLU_BURCA ATP-dependent protease ATPase subunit HslU… 82.4 6e-17
+++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++
sp|P0AAI3|FTSH_ECOLI ATP-dependent zinc metalloprotease FtsH OS… 46.2 2e-05
tr|Q74RB9|Q74RB9_YERPE Putative magnesium chelatase family prot… 46.2 3e-05
sp|P63343|FTSH_SALTY ATP-dependent zinc metalloprotease FtsH OS… 46.2 3e-05
sp|Q89U80|RUVB_BRADU Holliday junction ATP-dependent DNA helica… 45.4 3e-05
tr|Q0WBE7|Q0WBE7_YERPE Cell division protein OS=Yersinia pestis… 45.8 3e-05
tr|Q2K4M2|Q2K4M2_RHIEC ATP-dependent zinc metalloprotease FtsH … 45.4 4e-05
tr|Q8KKT3|Q8KKT3_RHIEC Probable ATPase (ATP-binding) protein OS… 44.3 8e-05
tr|A0A0H2XMS5|A0A0H2XMS5_BURCA ATP-dependent zinc metalloprotea… 43.9 1e-04
tr|H7C810|H7C810_BRADU ATP-dependent zinc metalloprotease FtsH … 43.5 2e-04
sp|Q9JUB0|RUVB_NEIMA Holliday junction ATP-dependent DNA helica… 42.7 2e-04
sp|P71377|FTSH_HAEIN ATP-dependent zinc metalloprotease FtsH OS… 42.7 3e-04
sp|P57015|YIFB_SALTY Uncharacterized protein YifB OS=Salmonella… 42.4 3e-04
Были взяты находки, которые в таблице находятся до “плюсиков”(поскольку там наблюдается резкий скачок в e-value).
Эти находки были сложены в сводный файл my_bacteria_hom.fasta.
Далее последовательности были выровнены в Muscle и построено дерево методом minimum evolution(слева) и maximum likelyhood(справа) с бутстрэпом(100 реплик).
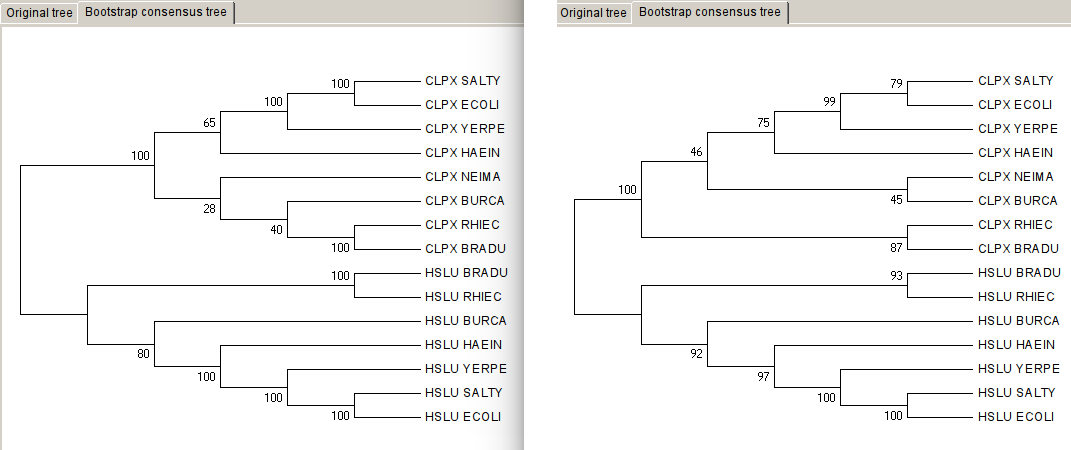

На данных деревьях ортологами друг друга являются все белки CLPX(и все белки HSLU тоже).
Паралогами же будут являтся пары вида CLPX-ECOLI - HSLU-ECOLI.
Как можно заметить, поддержка ветви, разделяющей паралоги, 100%
Видно, что CLPX и HSLU являются предками одного белка, и когда-то давно произошла дупликация, которая привела к их образованию.