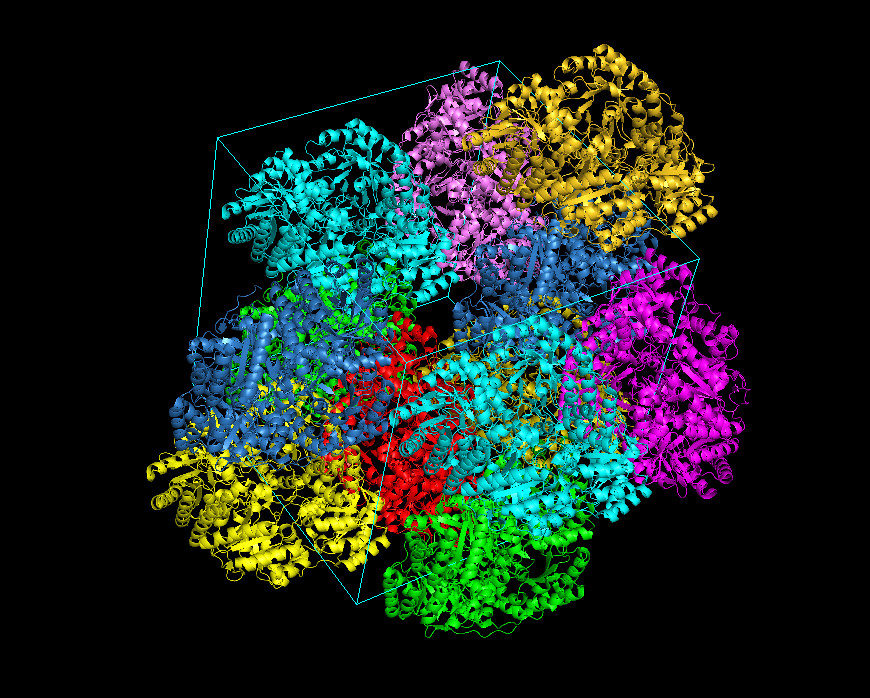
HEADER TRANSFERASE 21-AUG-08 3E9A TITLE CRYSTAL STRUCTURE OF 2-DEHYDRO-3-DEOXYPHOSPHOOCTONATE ALDOLASE FROM TITLE 2 VIBRIO CHOLERAE O1 BIOVAR ELTOR STR. N16961 . . . CRYST1 117.187 117.187 117.187 90.00 90.00 90.00 I 2 3 24 ORIGX1 1.000000 0.000000 0.000000 0.00000 ORIGX2 0.000000 1.000000 0.000000 0.00000 ORIGX3 0.000000 0.000000 1.000000 0.00000 SCALE1 0.008533 0.000000 0.000000 0.00000 SCALE2 0.000000 0.008533 0.000000 0.00000 SCALE3 0.000000 0.000000 0.008533 0.00000В программе Pymol можно нарисовать элементарную ячейку кристалла (команда 'show cell') и создавать симметричные дупликаты белка, согласно известной группе симметрии (команда 'symexp sym, 3e9a, 3e9a, [distance]'). Оказалось, что данный белок упакован в виде тетрамеров, которые могут быть расположены в трех перпендикулярных направлениях, см. Рис. 2.

Рис. 1. Упаковка структур 3E9A в кубической ячейке кристалла. Тетрамеры раскрашены разными цветами. Для наглядности, показаны не все тетрамеры, имеющие пересечение с кристаллографической ячейкой.
Каждый тетрамер имеет вид квадратного параллелепипеда, он имеет симметрии поворота на 180 градусов вокруг всех трех осей (но не имеет поворотной симметрии 90 градусов). По отношению к кубической ячейке тетрамеры расположены двумя способами:Следует отметить, что разделение тетрамеров на эти два вида условно, потому что можно определить другое разбиение кристалла на ячейки, в котором вершины кубов находятся в центрах кубов исходных ячеек. По отношению к этим новым ячейкам тетрамеры первого вида становятся лежащими на ребрах, а тетрамеры второго вида - погружены в грани. Других отличий нет, эти две системы ячеек эквивалентны.
Данные тетрамеры структуры 3E9A соответствуют наблюдаемой четверичной структуре этого белка в клетке. На сайте RCSB PDB приводятся три вида биологических единиц (Biological Assebly) для этого белка: тетрамер, и два разных димера, см. 3E9A.
Рассмотрим один тетрамер, а в нем одну из его цепей, цепь A1, см. Рис. 2.

Рис. 2. Тетрамер A1-A4, цепь A1 выделена розовым цветом.
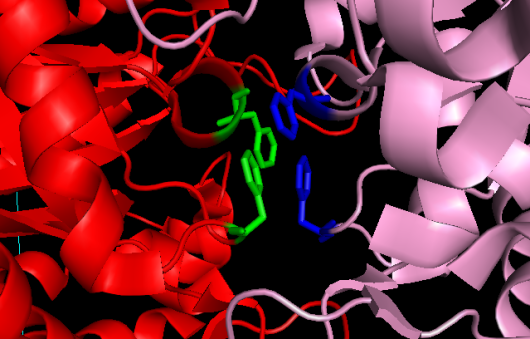
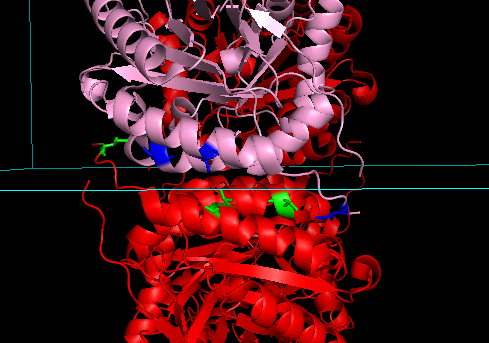
select inside, A1 and resn Leu+Ile+Val+Phe+Trp+Met select outside, (A* and not A1) and resn Leu+Ile+Val+Phe+Trp+Met # остатки пересечения окрестности одного множества с друним, и наоборот select inside2, byres (inside and (outside around 3.5)) select outside2, byres (outside and (inside around 3.5)) # отрисовка и расцветка найденных остатков show stick, inside2 show stick, outside2 color blue, inside2 color green, outside2Оказалось, что цепь A1 имеет одну общую гидрофобную область с цепью A2, образованную четырьмя фенилаланинами (см. Рис. 3а), и три потенциальных гидрофобных контакта с цепью A4, образованых изолейцинами, валинами, и лейцинами (см. Рис. 3б):
A1.PHE117 - A2.PHE142 A1.PHE142 - A2.PHE117 A1.ILE282 - A4.VAL222 A1.LEU270 - A4.LEU270 A1.VAL222 - A4.ILE282
 |  |
| а | б |
Рис. 3. Близкие гидрофобные остатки цепи A1 и соседних цепей тетрамера: (а) область контакта цепи A2 (красная, слева) с цепью A1 (розовая, справа); (б) три пары гидрофобных остатков цепи A1 (розовая, сверху) и цепи A4 (красная, снизу).
Аналогичный поиск гидрофобных контактов между цепями разных тетрамеров не дал результатов.select inside, A1 and elem O+N select outside, (A* and not A1) and elem O+N # остатки пересечения окрестности одного множества с друним, и наоборот select inside2, byres (inside and (outside around 3.5)) select outside2, byres (outside and (inside around 3.5)) # отрисовка и расцветка атомов найденных остатков select neighb, inside2 or outside2 show stick, neighb color white, neighb and elem C color yellow, neighb and elem S color blue, neighb and elem N color red, neighb and elem O # отображение близких дистанций dist hbonds, inside, outside, 3.5
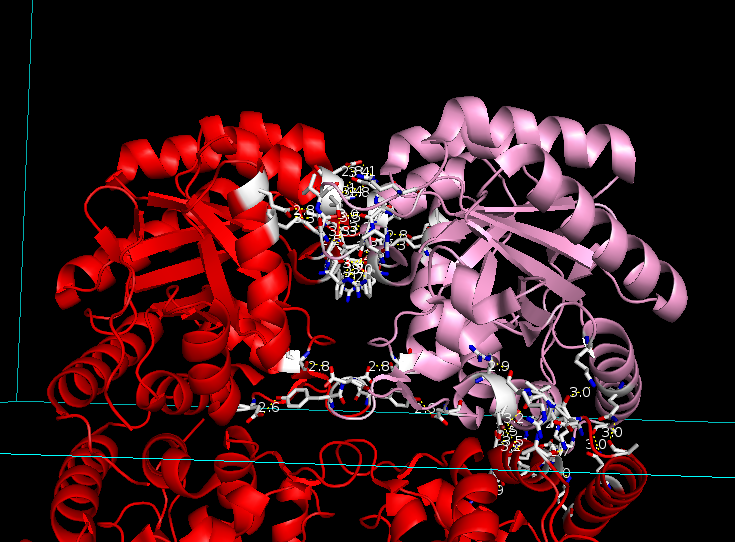
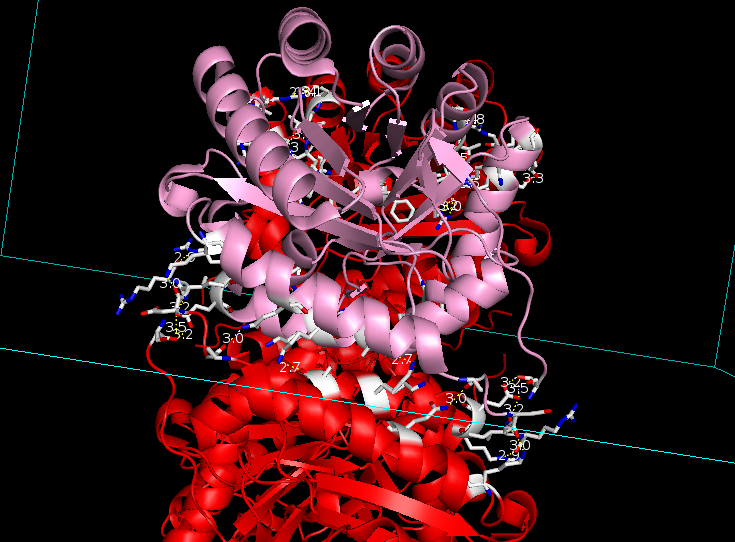
select inside, A1 and elem O+N select outside, (all AND not A*) and elem O+N # остатки пересечения окрестности одного множества с друним, и наоборот select inside2, byres (inside and (outside around 3.5)) select outside2, byres (outside and (inside around 3.5)) # отрисовка и расцветка атомов найденных остатков select neighb, inside2 or outside2 show stick, neighb color white, neighb and elem C color yellow, neighb and elem S color blue, neighb and elem N color red, neighb and elem O # отображение близких дистанций dist hbonds, inside, outside, 3.5 # отображение соседних остатков (чтобы их цвет показал цепь) select outside3, (byres (outside and (outside2 a. 2))) and not outside2 show stick, outside3Как видно на Рис. 4, внутри тетрамера есть много водородных связей между цепями A1 и цепями A2 и A4 (но не A3).
 |  |
| а | б |
Рис. 4. Водородные связи внутри тетрамера A: (а) область контактов цепи A2 с цепью A1 — вверху и в центре; (б) область контактов цепи A1 и цепи A4 — между петлями справа, слева, и посередине, между спиралями.
Есть также около десяти возможных водородных связей цепи A1 с цепями пяти других тетрамеров, см. Рис 5. Как видно из Рис. 1, цепь A1 красного тетрамера имеет 6 непосредственно близких тетрамеров, и потенциальные водородные связи были найдены для пяти из них. Предположительно, эти связи достаточно прочные, чтобы фиксировать форму кристалла, но энергии их связи недостаточно, чтобы в клеточной среде образовывались более крупные олигомеры.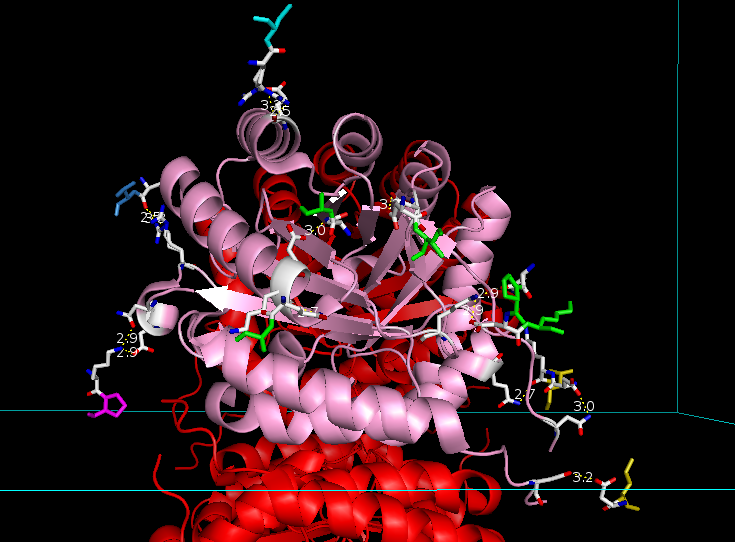
Рис. 5. Водородные связи цепи A1 с цепями других тетрамеров. Разными цветами показаны соседние остатки цепей других тетрамеров, цвета показывают, что контакты есть с пятью другими тетрамерами. С одним из тетрамеров (зеленым) есть четыре контакта.
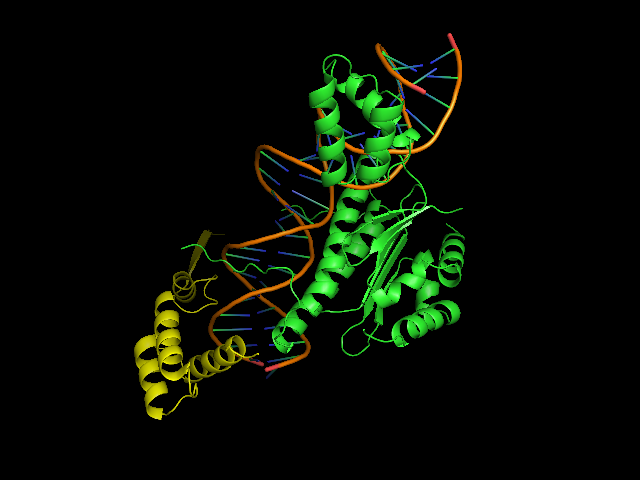
Рис. 6. Структура 1MNM. Цепь D, выделена желтым цветом, не имеет видимой связи с цепями ДНК.
Если же дополнить структуру содержимым соседней ячейки (команда Pymol 'symexp'), то видно, что контакт все же есть, см. Рис. 7.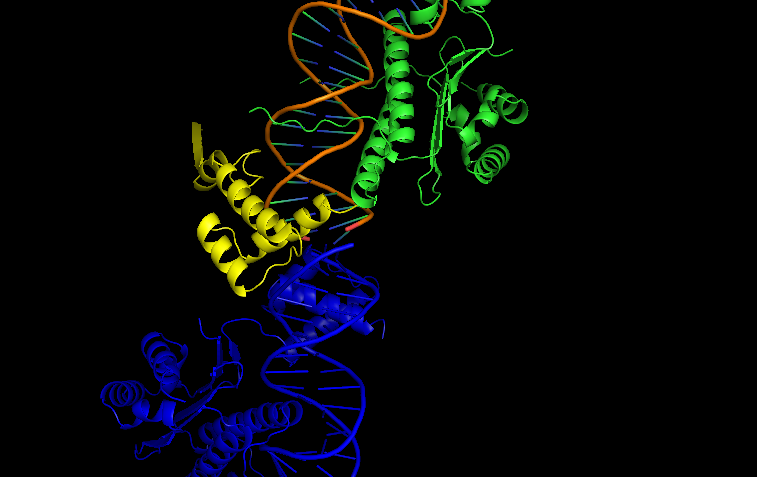
Рис. 7. Структура 1MNM. Изображение соседней кристаллографической ячейки показывает продолжение молекулы ДНК, которое имеет контакт с цепью D.
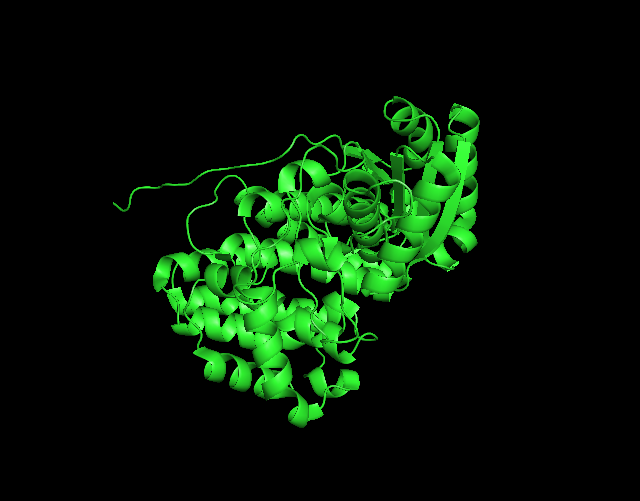
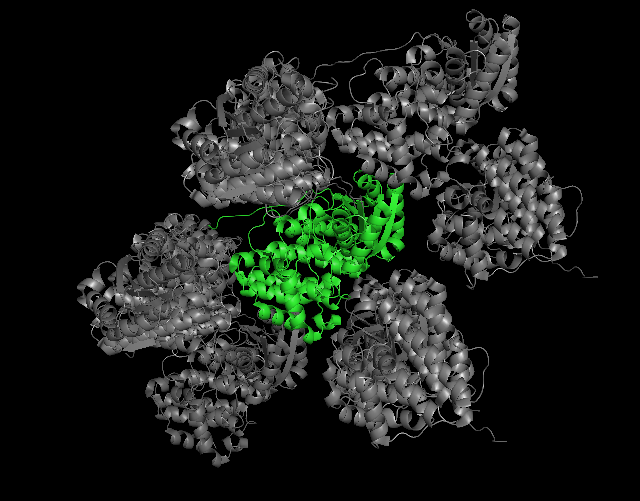
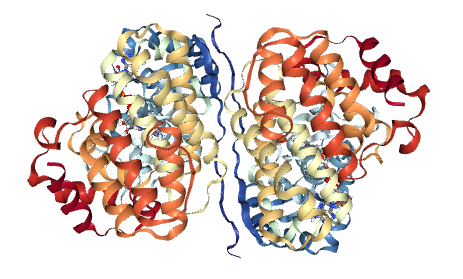
Например, у структуры 5YVR, см. файл 5yvr.pdb, асимметрическая единица состоит из одной цепи, см. Рис. 8а и 8б, хотя биологически активная форма белка образуется димером, см Рис. 8в.
 |  |  |
| а | б | в |
Рис. 8. Структура 5YVR: (а) асимметрическая единица, 1 цепь, (б) ячейки кристалла, (в) активная форма белка (3d viewer на сайте RCSB PDB).
И наоборот, у структуры 6AVY, см. файл 6avy.pdb, асимметрическая единица состоит из двух цепей, см. Рис. 9, хотя биологически активная форма белка — мономер.
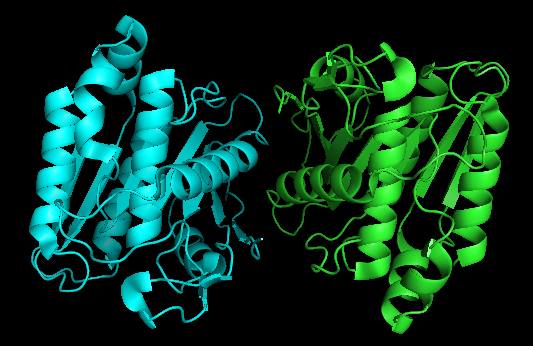
Рис. 9. Асимметрическая единица структуры 6AVY.
См. также страницу руководства сайта RCSB PDB Introduction to Biological Assemblies and the PDB Archive, где описаны причины и последствия таких несоответствий, а также приведены примеры таких структур.