Эволюционные домены. Pfam
Используемый домен в задании по БД Pfam: PF02540 (NAD synthase)
Из 15 доменных организаций выбрала 2: GATase и GATase + GMP_synt_C
| Археи |
Бактерии |
Эукариоты |
Вирусы |
| 222 |
3835 |
375 |
1 |
Было выбрано 20 последовательностей белков двух доменных архитектур.
Для этого была создана сводная таблица последовательностей с указанием длины каждой, доменной архитектуры,
принадлежности к определенному виду, царству. (Для отбора нужных последовательностей использовался скрипт
swisspfam_to_xls.py:
python swisspfam_to_xls.py -i /srv/databases/pfam/swisspfam.gz -o A2.xls -z -p PF02540
Excel-файл с выбранными последовательностями
Было проведено выравнивание двух групп последовательностей в пределах домена.
Для того, чтобы оставить в выравнивании Full из БД Pfam только нужные последовательности из 2 групп,
был использован скрипт filter_alignment.py.
Было построено филогенетическое дерево (fprotdist, fneighbor). Вид дерева в программе MEGA:
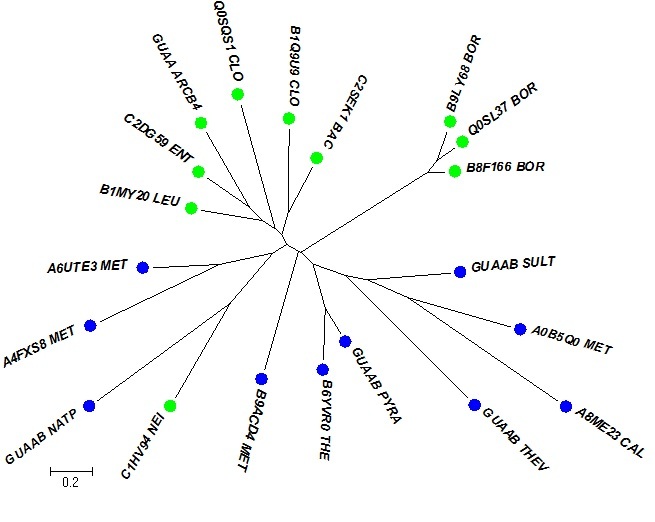
Синий - 1 группа
Зелёный - 2 группа
Скобочная форма: (GUAAB_NATP:0.78540,((A4FXS8_MET:0.63433,A6UTE3_MET:0.31661):0.26143,
((((B1MY20_LEU:0.29375,(C2DG59_ENT:0.24499,GUAA_ARCB4:0.41270):0.08785):0.07398,
Q0SQS1_CLO:0.61498):0.04249,(B1Q9U9_CLO:0.40322,C2SEK1_BAC:0.24897):0.10429):0.05282,
((((B9LY68_BOR:0.14558,Q0SL37_BOR:0.10159):0.07178,B8F166_BOR:0.08404):0.71726,
(((GUAAB_SULT:0.39997,(A0B5Q0_MET:0.51346,A8ME23_CAL:0.87369):0.21157):0.10665,
GUAAB_THEV:0.82966):0.16505,(GUAAB_PYRA:0.14093,B6YVR0_THE:0.24891):0.21845):0.08057):0.01328,
B9ACD4_MET:0.58927):0.06438):0.07768):0.31563,C1HV94_NEI:0.52074);
Полученное дерево видимо разделяется на 2 конкретные ветви (с одним и двумя доменами, соответственно).
Вполне возможно, что слияние доменов произошло единожды и передавалось по наследству. Правда один белок, у которого должно быть 2 домена, попал в группу белков с одним доменом
(возможно произошло ошибочное выравнивание).
© Dzama Margarita,2010-2011
