Реконструкция филогении по нуклеотидным последовательнсотям. Анализ деревьев, содержащих паралоги.
Построение дерева по нуклеотидным последовательностям
При помощи ряда программ (entret, seqret, muscle и другие) были получены последовательности ДНК, кодирующие 16S рРНК, и построено их выравнивание.Данный файл был подан на вход программе fdnapars.
+--GEOKA
+----3
| | +-BACSU
| +--6
| | +--BACAN
| +--5
| | +-ENTFA
| +--4
| +-----LACAC
|
| +-CLOTE
1---2
| +-CLOB1
|
+------FINM2
Скобочная форма: ((GEOKA:0.05020,(BACSU:0.03176,(BACAN:0.04626,(ENTFA:0.04280, LACAC:0.11065):0.03616):0.01931):0.02491):0.07847,(CLOTE:0.04423, CLOB1:0.04035):0.06160,FINM2:0.12311);
Правильные ветви:
(LACAC, ENTFA)
(CLOTE, CLOB1)
((CLOTE, CLOB1), FINM2)
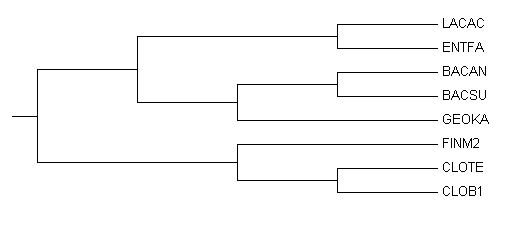
Верное дерево:
Построение и анализ дерева, содержащего паралоги
+--HSLU_GEOKA
+-11
+-10 +--CLPY_BACSU
! !
+-----------------9 +-----HSLU_BACAN
! !
! +--------HSLU_ENTFA
+--8
! ! +--------------CLPX_ENTFA
! ! !
! ! ! +--CLPX_BACAN
! +-----------4 +--7
! ! +-----6 +--CLPX_BACSU
+--2 ! ! !
! ! +--5 +-----CLPX_GEOKA
! ! !
! ! ! +--CLPX_CLOTE
1 ! +--------3
! ! +--CLPX_CLOB1
! !
! +-----------------------------CLPE_BACSU
!
+--------------------------------CLPC_BACSU
Ортологи:
CLPX_BACAN, CLPX_BACSU
CLPX_CLOTE, CLPX_CLOB1
Паралоги:
CLPY_BACSU, CLPX_BACSU
CLPE_BACSU, CLPC_BACSU
Доделки
1) Отоборанные бактерии| Мнемоника | AC записи EMBL | Координаты FT, в которой описано 16S рРНК |
| BACAN | AE016879 | 9335:10841 |
| BACSU | CP002468 | 2306291..2307843 |
| CLOB1 | FR773526 | 9246..10747 |
| CLOTE | AE015927 | complement(8715..10223) |
| ENTFA | CP002491 | complement(374851..376399) |
| FINM2 | AP008971 | 197837..199361 |
| GEOKA | BA000043 | 10421..11973 |
| LACAC | CP000033 | 59255..60826 |
Команда, которой создавали файл с последовательностью рРНК данной бактерии:
seqret "BACAN.enret[9335:10841]" stdout >> rna.fasta (для всех остальных аналогично)
seqret CLOTE.entret -sask stdout >>rna.fasta (комплементарная цепь)
2) Команда, с помощью которой получено выравнивание последовательностей 16S рРНК бактерий:
muscle -in newrna.fasta -out rna.fasta
3) Для построения деревьев была использована команда fdnapars. На вход подавался файл с последовательностями rna.fasta. Полученный файл.
Правильные ветви в полученном дереве:
(LACAC, ENTFA)
(CLOTE, CLOB1)
((CLOTE, CLOB1), FINM2)
4)Реконструкция по белкам, сделанная в предыдущих заданиях, оказалась менее точной, чем реконструкция по РНК:
fprotpars - 2 верных ветви
алгоритм Neighbor-Joining - топология дерева неправильная [нет (LACAC, ENTFA)]
алгоритм UPGMA - топология неверная [нет (LACAC, ENTFA)]
TreeTop - топология дерева неправильная [совпадает только ((CLOTE, CLOB1), FINM2) и (CLOTE,CLOB1)]
Во всех этих реконструкциях встретились ветви
(LACAC,ENTFA,BACAN,BACSU,GEOKA) против (FINM2,CLOTE,CLOB1)
(CLOTE,CLOB1) против (FINM2,LACAC,ENTFA,GEOKA,BACAN,BACSU)
© Dzama Margarita, 2011