Выравнивание последовательностей.
.png)
Задание №1.
Из семейства HSP70 было выбрано 6 белков:P9WMJ9, P36415, Q8TQR2, Q9I8F9, Q18GZ4, Q57AD7. Два из этих белков принадлежат археям, два - бактериям и соотвественно два - эукариотам. Затем, с помощью программы JalView мы построили выравнивание этих последовательностей, часть которого можно увидеть на картинке.(Рис. 1) Мы использовали вырванивание Clustal(with defaults) с раскраской ClustalX(Above Identity Threshold). Благодаря этому мы смогли выделить абсолютно консервативные позиции и отметили эти колонки буквой "C". Колонки, содержащие гэпы, было видно сразу после выравнивания программой, и их мы пометили буквой "G". Для выявления абсолютно функционально консервативных позиций были применены различные окраски(по разным свойствам), а также подсказки из словаря терминов на странице kodomo.fbb.msu.ru. Эти колонки были отмечены сиволом "F". Проект с полным выравниваем вы можете скачать по ссылке, приложенной ниже.
Проект выравнивания.В Таблицы 1 приведена информация о консервативности белков. Длина выравнивания - 683
| Название последовательности | Длина последовательности | Длина выравниванииz | Число абсолютно консервативных | Процент абсолютно консервативных | Число абсолютно функционально консервативных | Процент абсолютно функционально консерватинвых | Число позиций с гэпами | Процент позиций с гэпами |
| DNAK_BRUAB | 637 | 683 | 197 | 28,8 | 301 | 44,1 | 44 | 6,4 |
| DNAK_HALWD | 641 | 683 | 197 | 28,8 | 301 | 44,1 | 42 | 6,15 |
| HSP71_ORYLA | 639 | 683 | 197 | 28,8 | 301 | 44,1 | 40 | 5,9 |
| DNAK_METAC | 617 | 683 | 197 | 28,8 | 301 | 44,1 | 63 | 9,2 |
| HS7C1_DICDI | 640 | 683 | 197 | 28,8 | 301 | 44,1 | 39 | 5,7 |
| DNAK_MYCTU | 625 | 683 | 197 | 28,8 | 301 | 44,1 | 56 | 8,2 |
Задание №2.
Произошедшие мутации:(основываясь на выравнивании, представленном на Рис 3.)
1. На позиции 1 вставка аминокислоты А(аланин) от p1 к p2
2. Вставка аминокисоты L(лейцин) на позиции 15 от p2 к p3
3. Замена аминокислоты L(лейцин) на M(метионин) на позиции 15 от p4 к p5
4. Замена аминокислоты N(аспарагин) на K(лизин) на позиции 16 от исходной последовательности к p1
5. На позиции 17 замена аминокислоты S(серин) на N(аспарагин) от исходной последовательности к p1
6. Замена на позиции 17 аминокислоты N(аспарагин) на S(серин) от p5 к p6
7. На позиции 18 замена аминокислоты E(глутаминовая кислота) на S(серин) от исходной последовтельности к p1
8. Замена на позиции 18 аминокислоты S(серин) на E(глутаминовая кислота) от p5 к p6
9. Выпадение аминокислоты E(глутаминовой кислоты) на позиции 21 от p1 к p2
10. Вставка аминокислоты E(глутаминовая кислота) на позиции 27 от исходной последовательности белка к p1
11. Выпадение аминокислоты E(глутаминовая кислота) на позиции 27 от p5 к p6
12. Вставка аминокислоты I(изолейцин) на позиции 29 от p3 к p4
13. Вставка аминокислоты A(аланин) на позиции 34 от p3 к p4
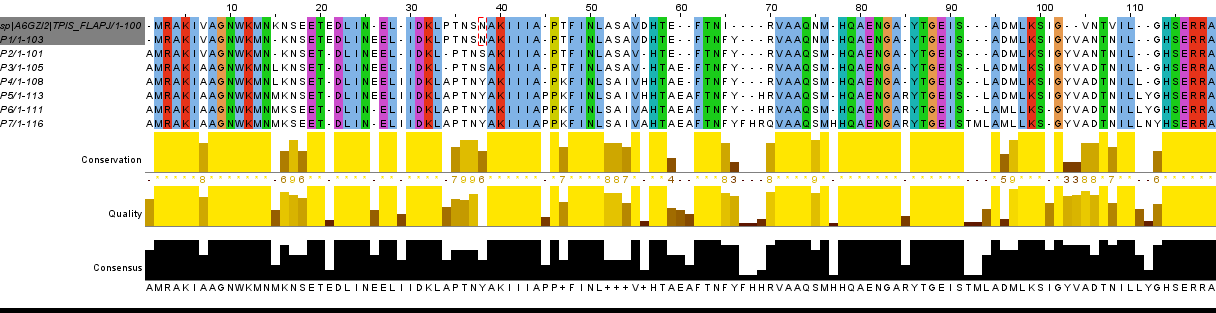
Ручные исправления:(Рис 3.)
1. Поменяли местами аминокислоты E и T в позициях 19 и 20 соответсвенно в p1, так как очень мало вероятны
две мутации подряд, тем более с последующим возвращением к исходному нуклеотиду.
2. На позициях 27-28-29 также были произведены изменения. С 29 позиции в p6, p7 были удалены гэпы и поставлены
на позицию 27 соотвествующих поколений. Таким образом, образовалась абсолютно консервативная колонка на позиции 28,
а также вероятность таких мутаций возросла.
Полную информацию о проделанной работе можно найти в проекте JalView: Проект задания №2
Скрипт, с помощью которого были полученны 7 поколений со случайными мутациями: Скрипт задания №2
