Поиск по сходству.
.png)
Задание №1.
Ссылка на проект с выравниванием
Наш белок: A6GZI2 (TPIS_FLAPJ) Triosephosphate isomerase
| ID/AC | Название белка | Coverage | Identity % | E-value | Гомологичен исходному белку или нет |
| Q10657 (TPIS_CAEEL) | Triosephosphate isomerase | 98 | 45 | 4e-65 | да |
| P48499 (TPIS_LEIME) | Triosephosphate isomerase | 96 | 42 | 1e-62 | да |
| Q9C401 (TPIS_ZYGBA) | Triosephosphate isomerase | 98 | 41 | 6e-57 | да |
| P55275 (TPIS_HELVI) | Triosephosphate isomerase | 87 | 45 | 2e-54 | да |
| Q9P940 (TPIS_CANAL) | Triosephosphate isomerase | 98 | 40 | 8e-53 | да |
| P48492 (TPIS_GRAGA) | Triosephosphate isomerase | 96 | 37 | 4e-49 | да |
| A8MCN3 (TPIS_CALMQ) | Triosephosphate isomerase | 87 | 27 | 7e-06 | нет |
| Q8ZX28 (TPIS_PYRAE) | Triosephosphate isomerase | 79 | 26 | 0.005 | нет |
| Q12UK2 (TPIS_METBU) | Triosephosphate isomerase | 34 | 28 | 1.1 | нет |
.png)
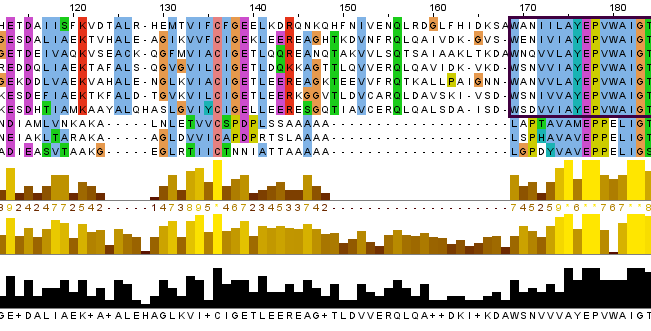
Комментарий:Учитвая все пункты, для оценки гомологичности белков, мы пришли к выводу: последние три белка в списке не являются
гомологичными к нашему.
1.E-value достаточно большой.
2.Процент схожести белков слишком маленький(identity составляет всего 26-28%)
3.Из множественного выравнивания мы видим, что эти последователности содержат очень много гэпов.\
Мы нашли блоки достоверного выравнивания и выделили их синим цветом. Как видно из рисунков, приведенных выше, последние
три последовательности не входят в них.
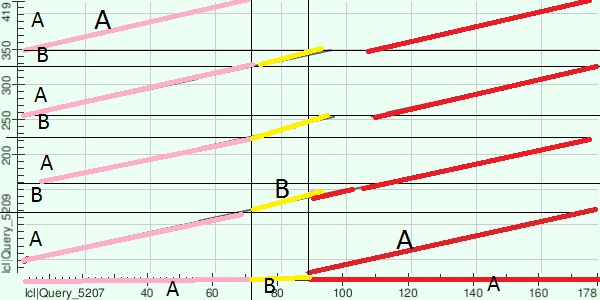

- Query ID: Query_5207
Description: V3ZJ66|V3ZJ66_LOTGI Uncharacterized protein OS=Lottia gigantea
Query Length: 179 - Subject ID: Query_5209
Description: A0A0N8DC56|A0A0N8DC56_9CRUS Cysteine and glycine-rich protein OS=Daphnia magna
Query Length: 179
Комментарий:Делеции выделены красным цветом на первом рисунке. На втором рисунке разными цветами показаны
дуплицированные участки.
Таким образом, как показано на рисунке выше, участки последоватнльностей с помощью однобуквенного кода можно записать так:
V3ZJ66: ABA
A0A0N8DC56: ABABABA