Практикум 3. Построение и анализ дерева, содержащего паралоги
В таблице ниже представлены выбранные протеобактерии:
| Сокращение | Название |
| PM | Pasteurella multocida |
| HI | Haemophilus influenzae |
| ST | Salmonella typhimurium |
| PRM | Proteus mirabilis |
| VC | Vibrio cholerae |
| PA | Pseudomonas aeruginosa |
| RS | Rhodobacter sphaeroides |
Для этих бактерий нашли достоверные гомологи белка CLPX_ECOLI и реконструировали дерево этих гомологов.
HSLU-HSLV ---> HslU--HslV peptidase ATPase
zinc metalloprotease ---> ATP-dependent zinc metalloprotease
Изображение дерева, построенное с помощью метода Neighbor-Joining:
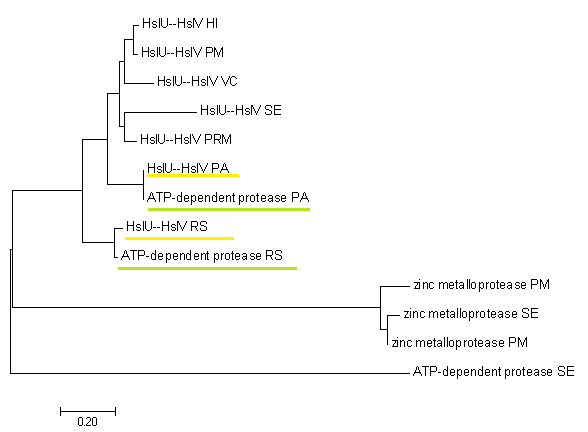
Можно выделить две ортологичные группы - zinc metalloprotease и HslU--HslV. Они образовались в результате видообразования. Желтым и зеленым подчеркнуты две пары паралогов, образовавшиеся в результате дупликации генов.
© Гурылева Мария Вячеславовна 2016