Практикум 6. Мотивы в белках. PSSM и паттерны. PSI-BLAST. Банк Prosite
Был выбран случайный идентификатор: C4Z088
На странице белкового BLAST в NCBI выберали PSI-BLAST и выполнили поиск по банку Swiss-Prot.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 165 | Q5F5V4.1 | 0.005 | A4WBG0.1 | 0.006 |
| 2 | 140 | Q4US07.1 | 7e-04 | Q9JX17.1 | 0.007 |
| 3 | 142 | A6X374.1 | 6e-09 | P57845.1 | 0.017 |
На третьей итерации разница между E-value худшей "правильной" находкой и "лучшей" неправильной уже стала достаточно большая. Поэтоиу можно предположить, что этот набор является семейством гомологичных белков
В банке Prosite был найден паттерн, описывающий выбранно в практикуме 2 семейство белков.
Для этого была просканирована последовательность RL1_PASMU. Название семейства в Prosite:
PS01199 RIBOSOMAL_L1.
Был найден только один паттерн: MrvVgq.LGqiLGPRGlMPN. Он расположен на позициях: 121 - 139.
Консенсусный паттерн: [IMGV]-x(2)-[LIVA]-x(2,3)-[LIVMY]-[GAS]-x(2)-[LMSF]-[GSNH]-[PTKR]-[KRAVG]-[GN]-x-[LIMF]-P-[DENSTKQPRAGVI]
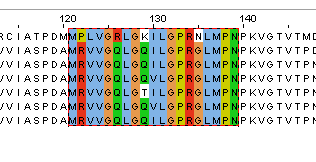
Более строгий паттерн: M-[PR]-[LV]-V-G-[RQ]-L-G-[KQT]-[IV]-L-G-P-R-[NG]-L-M-P-N-P-K-V-G-T-V-T

Затем был выполнен поиск в банке Prosite по более строгому паттерну. 146 соотвествий было найдено. С помощью команды MATCHLIST находки были занесены в список: MATCHLIST.txt
Теперь нужно сравнить список находок со списком всех представителей данного семейства белков из Proteobacteria, имеющихся в Swiss-Prot. "Правильный" список был получен на сайте Uniprot: TRUE.list
Для сравнения этих двух список в Python был написан скрипт: script.ipynb
Число истинных находок: 146(весь список MATCHLIST.txt)
Число ложных находок: 0
Число ненайденных: 282
© Гурылева Мария Вячеславовна 2016