Для сборки контигов использовался очищенный в предыдущем практикуме файл с чтениями - trimm.fastq.
Из чтений первоначально были выделены k-меры длиной 31 с помощью пакета velvet.
Использовалась следующая программа:
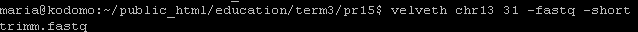
В результате был получен файл contigs.fa с контигами.
Всего в графе 628 узлов (т.е. собрано 628 контигов), n50 = 223, максимальная длина контига 1010, общая длина 45561. (команда: velvetg vel)

Задание 2.
Сравнение полученных контигов с последовательностью хромосомы было проведено с помощью программы blastn (алгоритм megablast).
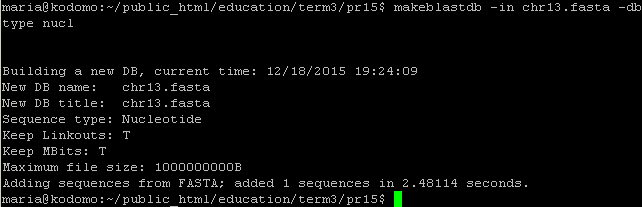
Для создания базы blast из последовательности хромосомы использовалась следующая программа:
makeblastdb -in chr13.fasta -dbtype nucl
Записк blastn был осуществлён с помощью программы:
blastn -db chr13.fasta -query vel/contigs.fa -outfmt 6 -out chr13.fasta

Результат в виде таблицы
В исходной таблице присутствуют контиги, картировавшиеся на одно место и на несколько мест.
Всего однозначно картируется 317 контигов, что составляет 1,753%.
СПАСИБО ЗА ПРОСМОТР
© Мария Медведева