В качестве организма была выбрана Equus przewalskii (Лошадь Пржевальского). Лошадь Пржевальского — единственный вид диких лошадей, сохранившийся на нашей планете. Для науки этот вид был открыт лишь в 1879 г. и получил свое название в честь имени первооткрывателя — путешественника Николая Пржевальского.

Исходная таблица

описание образца SAMN02808398 (BioSample) с сайта NCBI (сайт)
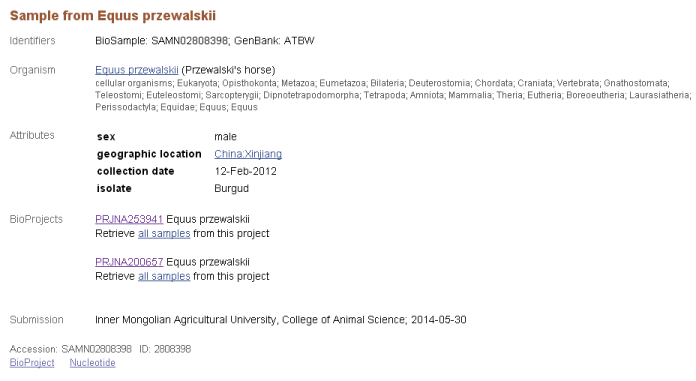
В описании указано следующее:
пол - самец
географическое положение - Синьцзян-Уйгурский автономный район (Китай)
дата сбора - 12/02/12
Изолят (субпопуляция) - Burgud
описание проекта (BIOPROJECT ID - PRJNA200657)
Ссылка на страницу BioProject PRJNA200657
На странице приведена аннотация к проекту:
Перевод аннотации: Currently, Przewalski’s horse (Equus przewalskii) is the only wild horse species survived in the world. The total number is around 2000 and distribute in wild and zoos. Przewalski’s horse is about 132cm height, , shorter than the domestic horse. Like other wild equids, it has black dorsal stripe, black tail and black erect mane without forelock. The most distinctive feature is the color of its coat: sandy. All of przewalski’s horse are come from 12 ancestors. It has 66 chromosomes which is different with the domestic horse (2n=64). Having its genome sequence could help to understand the genetic relationship between przewalski and domestic horse and is conducive for the protection of przewalski’s horse.
Также на странице проекта приведен ряд его параметров:

| контиг | Accession | Name | Length |
| Самый длинный | ATBW01009134 | contig8909 | 942,236 |
| Самый короткий | ATBW01121767 | contig121677 | 200 |
Задание 2. Составление таблицы митохондриальных генов указанного мха.
Данный мне вид мха - Racomitrium ericoides.

Требовалось найти полный геном митохондрий указанного мха в базе данных Nucleotide (NCBI), для чего нужно было составить поисковый запрос:

Первая запись лежит в NCBI, вторая - в GenBank. Можно предположить, что обе записи содержат результаты одного и того же секвенирования, т к у обеих одинаковые название, авторы и количество пар нуклеотидов. В комментариях к описанию записи в NCBI говорится, что референсная последовательность совпадает с приведенной в GenBank. ("The reference sequence is identical to KP233863.")
Запись в NCBI датируется более поздним временем (27-MAR-2015 - NCBI, 14-FEB-2015 - GenBank), поэтому я выбрала для дальнейшей работы именно ее.
Далее нужно было получить список всех генов и информацию об их количестве. Для этого я перешла по ссылкам Genome и Gene в разделе Related information.

Как видно из таблицы, в геноме митохондрий Racomitrium ericoides присутствует 66 генов, из которых 49 кодируют белки, 3 - рибосомальные РНК и 24 - тРНК.
Затем я сделала таблицу генов, отсортированных по положению на хромосоме. (для этого я сначала отсортировала файл на странице Gene: Sort by Chromosome, вот, что получилось в NCBI, затем сказала новый изменённый файл)
gene_result.txt
Задание 3. Опиcать десять ключей, используемых в таблицах особенностей.
ссылка на таблицу таблицу особенностей с сайта INSDC
| ключ | его описание | пример |
| CDS | Protein-coding sequence |
803..1344 /gene="traN" /product="transfer protein N" |
| RBS | ribosome binding site |
95..100 /gene="sod" |
| rep_origin | Origin of replication |
6 /direction=LEFT /note="ori" |
| repeat_region | region of genome containing repeating units |
80..401 /rpt_type=DISPERSED /rpt_family="Alu-J" |
| tRNA | mature transfer RNA |
655..730 /gene="tRNA-Leu(UUR)" /anticodon=(pos:678..680,aa:Leu,seq:taa) /product="transfer RNA-Leu(UUR)" |
| mat_peptide | mature peptide or protein coding sequence; coding sequence for the mature or final peptide or protein product following post-translational modification; the location does not include the stop codon (unlike the corresponding CDS) |
55..399 /gene="TCR1A" /product="T-cell receptor alpha chain" |
| mobile_element | region of genome containing mobile elements | /mobile_element_type="transposon:Tnp9" |
| LTR | long terminal repeat, a sequence directly repeated at both ends of a defined sequence, of the sort typically found in retroviruses | 1..244 /note="5'/LTR" |
| misc_signal | any region containing a signal controlling or altering gene function or expression that cannot be described by other signal keys (promoter, CAAT_signal, TATA_signal, -35_signal, -10_signal, GC_signal, RBS, polyA_signal, enhancer, attenuator, terminator, and rep_origin) | 83..86 /regulatory_class="CAAT_signal" /gene="Ambn" |
| 5'UTR | 1) region at the 5' end of a mature transcript (preceding the initiation codon) that is not translated into a protein; 2) region at the 5' end of an RNA virus genome (preceding the first initiation codon) that is not translated into a protein; | 1..63 /gene="crasp" |
Для выполнения этой задачи использовался BLASTN.
На вход была дана нуклеотидная последовательность, полученная в практикуме 6.
данная последовательность в формате fasta
Первые результаты выдачи BLASTN: (использовался алгоритм "samewhat similar sequences" по банку nr)
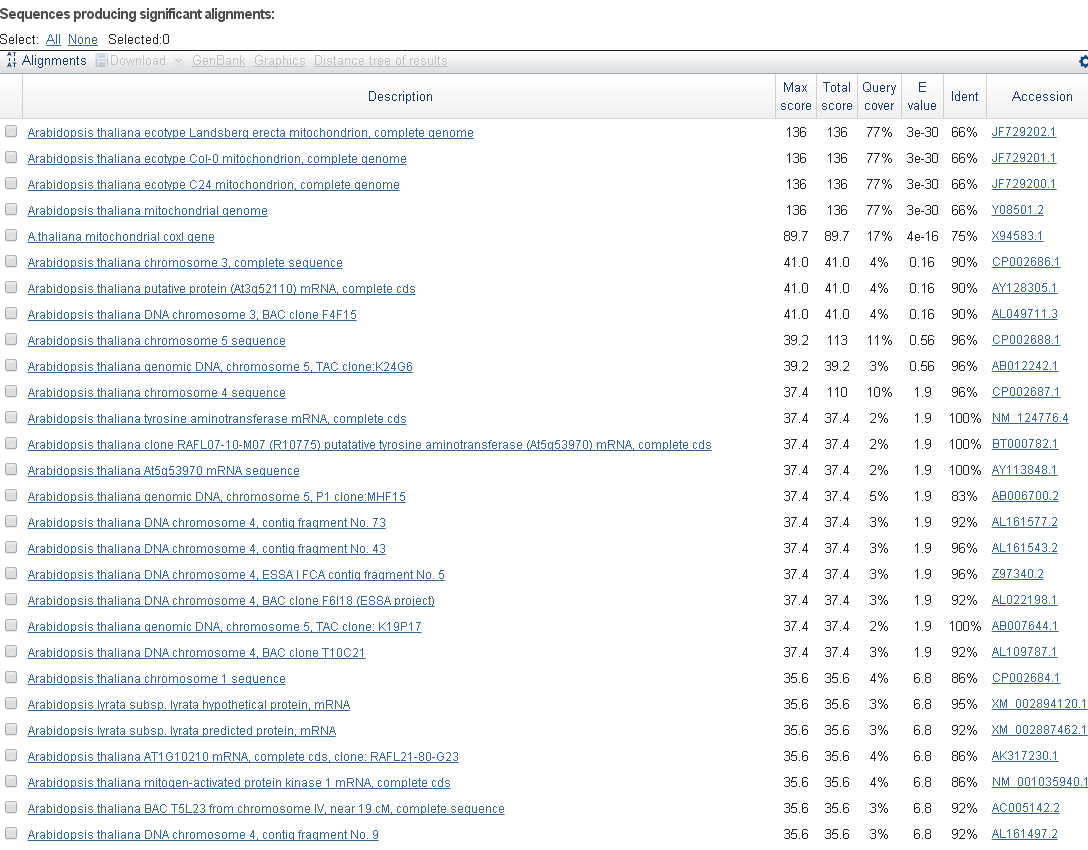
Для последующей работы я отобрала 4 лучшие находки из достоверных, у которых процент идентичности превышает 70%.
Как видно из рисунка они принадлежат к роду - Arabidopsis thaliana.
 |
 |
Все четыре находки являются последовательностями митохондриального гена.
Таблица с информацией о находках:
| Вид | Ссылка на GenBank | Identities | Выравнивание | Выравнивание в fasta-формате |
| Arabidopsis thaliana ecotype Landsberg erecta mitochondrion, complete genome | 1 | 359/546(66%) | Рис. а | 1.fasta |
| Arabidopsis thaliana ecotype Col-0 mitochondrion, complete genome | 2 | 359/546(66%) | Рис. b | 2.fasta |
| Arabidopsis thaliana ecotype C24 mitochondrion, complete genome | 3 | 359/546(66%) | Рис. c | 3.fasta |
| Arabidopsis thaliana mitochondrial genome | 4 | 359/546(66%) | Рис. d | 4.fasta |
Изображения выравниваний:




Можно заключить, что:
1) Последовательность, полученная в практикуме 6, - митохондриальный ген.
2) С высокой вероятностью она принадлежит организму из таксономических групп Eukaryota; Plantae; Angiosperms; Eudicots; Rosids; Brassicales; Brassicaceae; Arabidopsis; Arabidopsis thaliana.
СПАСИБО ЗА ПРОСМОТР
© Мария Медведева