Упражнения
1) SEGRET для сборки нескольких файлов в формате fasta в единый файл.команда:
maria@kodomo:~/public_html/education/term3/pr9$ seqret
Read and write (return) sequences
Input (gapped) sequence(s): sw: *pr9_seq
output sequence(s) [1pr9_seq.fasta]: out.fasta
2) SEGRETSPLIT для того, чтобы один файл в формате fasta с несколькими последовательностями разделить на отдельные fasta файлы.
Входной файл: ext.fasta
команда:
maria@kodomo:~/public_html/education/term3/pr9$ seqretsplitB
Read sequences and write them to individual files
Input (gapped) sequence(s): ext.fasta
output sequence(s) [1pr9_seq.fasta]:
команда:
maria@kodomo:~/public_html/education/term3/pr9$ seqret
Read and write (return) sequences
Input (gapped) sequence(s): ali.fasta
output sequence(s) [WS2933_1-353.fasta]: msf::ali.msf
команда:
maria@kodomo:~/public_html/education/term3/pr9$ featcopy
Read and write feature table
Input featute table: NC.gb
Features output [NC.gff]: NC.gff
команда:
maria@kodomo:~/public_html/education/term3/pr9$ cusp
Create a codon usage table from nucleotide sequence(s)
Input nucleotide sequence(s): embl: Y11489.1
Output file [y11489.cusp]:
Сравннение аннотаций генов белков в хромосоме бактерии Gordonibacter pamelaeae 7-10-1-b с трансляциями длинных открытых рамок считывания
Для работы я взяла анаэробную кокко-образную бактерию Gordonibacter pamelaeae 7-10-1-b, которая мне досталась в первом семестре. (Бактерия имеет только одну хромосому)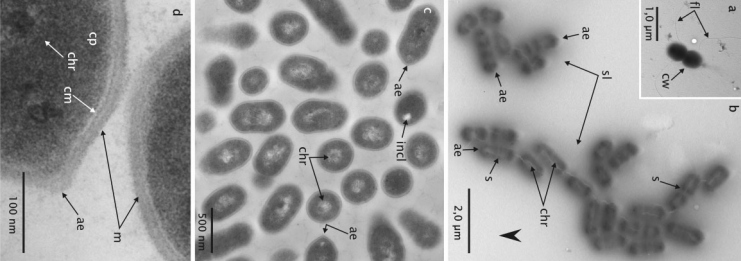
Её геном NC_021021.1 . Длина последовательности генома: 3 608 022 п. н.
Нуклеотидная последовательность приведена в формате fasta.
Задание 1. Список трансляций открытых рамок с помощью команды getorf пакета EMBOSS
Запись генома бактерии была получена из банка EMBL с помощью команды:getorf -minsize 180 -table 11 -find 1 -circular yes bacter.fasta out.fasta
bacter.fasta - это последовательность генома бактерии Gordonibacter pamelaeae, а out.fasta - результат.
-minsize 180 -минимальная длина рамки
-table 11 – бактериальный генетический код
-find 1 – поиск, начиная со старт- и заканчивая стоп-кодоном
-circular yes – кольцевая хромосома
Затем, с помощью команды "infoseq FQ859176.orf -only -name -length -description -outfile FQ_out" получены ID открытой рамки, координаты в геноме и длина трансляции.
-only - показывает только указанные параметры
-name - ID открытой рамки
-length - длина трансляции в остатках
-description - описание содержит координаты открытых рамок
Полученная информация была обработана в Excel.
Задание 2. Список аннотированных генов белков
Аминокислотные последовательности всех белков - NC_021021.faaСписок аннотированных белков был получен из файла NC_021021.ptt
Результат (в виде таблицы с полями: locus_tag, from, to, ori, length, PID, product) - Excel.
Задание 3. Сравнение 2ух таблиц Excel
По заданию нужно было сравнить две полученные таблицы, для чего была создана сводная таблица.Рассмотрев первые 150 случаев, полное совпадение обнаружено не было.
Частыми были случаи несовпадения ни длины, ни начала, ни конца.
Annotation GPA_00120 3358 3504 1 48
ORF NC_021021.1_12 3461 3646 -1 62
Положение конца смещено на три нуклеотида может объясняться тем, что в таблице с аннотированными белками стоп-кодон учитывается, а в таблице с рамками считывания – нет.
Annotation GPA_27080 2716887 2717231 1 114
ORF NC_021021.1_8133 2716887 2717228 -1 114
Annotation GPA_01520 147006 147353 1 115
ORF NC_021021.1_420 147006 147350 1 115
Также были случаи, когда не было открытой рамки считывания (нельзя было сопоставить начало, конец).
СПАСИБО ЗА ПРОСМОТР
© Мария Медведева