Была найдена страница бактерии Gordonibacte rpamelaeae 7-10-1-b в базе данных Assembly и ее последовательность. Со страницы последовательности был скачан fasta-файл с хромосомой , а также файл с особенностями . Последний файл был обработан скриптом features2CDSs.py . Выходной файл: newfile.xls.
Для анализа были выбраны 300 самых длинных кодирующих последовательностей. Они представлены в файле newfile1.xls), где на вкладке newfile.xlsпредставлены также данные выходного файла.
Последовательность Шайн-Дальгарно – сайт связывания рибосом на молекуле мРНК прокариот, расположенная обычно на расстоянии около 10 нуклеотидов до стартового кодона AUG. Консенсусная последовательность состоит из 6 нуклеотидов AGGAGG.
В качестве области поиска у отобранных генов был взят участок [-16;0]. (вкладка search1 в файле newfile1.xls). Для всех генов, в соответствии с рекомендацией в задании, область поиска была немножко расширена до [-20;0] (вкладка search2 в newfile1.xls).
Координаты последовательностей, предположительно включающих последовательности Шайн-Дальгарно.
Далее использовались онлайн версии программы MEME.
Я запускала MEME со следующими параметрами: длина мотива - от 6 до 10 н., поиск только по данной цепи (поскольку даны кодирующие последовательности), в последовательности ожидается от 0 до 3 появления мотива.

Параметры, с которыми была запущена MEME.
HTML-выдача программы MEME - здесь.
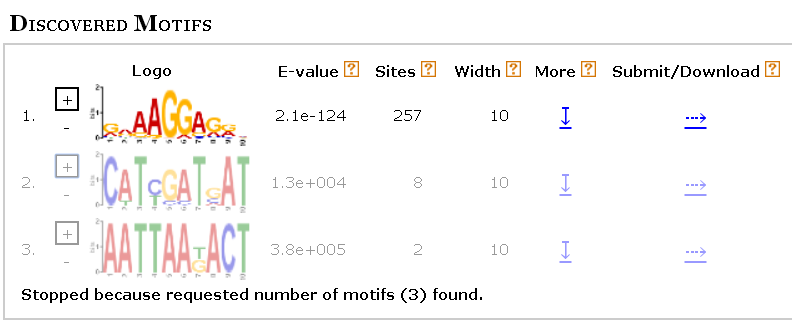
Три разных мотива, найденные MEME.

Лого мотивов, найденных программой MAST.
Первый их этих мотивов - искомая последовательность Шайна-Дальгарно. Видно, что e-value первого найденного мотива намного меньше, чем e-value остальных.
Затем я произвела поиск в MEME с теми же параметрами, но уже только одного наилучшего мотива. (html) В результате была получена позиционная матрица весов (PWM).
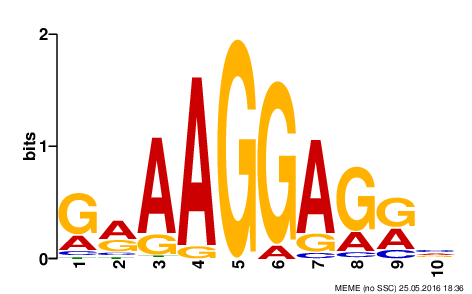
Позиционная матрица весов для первого мотива:
Затем уже проводился поиск по регионам перед всеми генами с помощью ресурса FIMO, предварительно расширив область поиска c -26 до -1 позиции до начала кодирующей последовательности.
Найденная PWM была подана на вход алгоритму FIMO, который искал мотив последовательности Шайна-Дальгарно уже по всем генам.
Порог E-value: 0.01.
В результате было найдено 1510 генов, удовлетворяющих заданным условиям.
Выдача программы приведена в таблице FIMOresults.xls.
Задание 3. Определить сайты связывания данного транскрипционного фактора в данном участке хромосомы человека.
Мне был выдан файл chipseq_chunk1.fastq с ридами Illumina, полученный в результате chip-seq эксперимента. С помощью команды fastqcchipseq_chunk1.fasta был сделан контроль качества прочтений. В выдаче программы содержались файлы chipseq_chunk1_fastqc.html и chipseq_chunk1_fastqc.zip. В файле формата html содержалась картинка, представленная ниже:
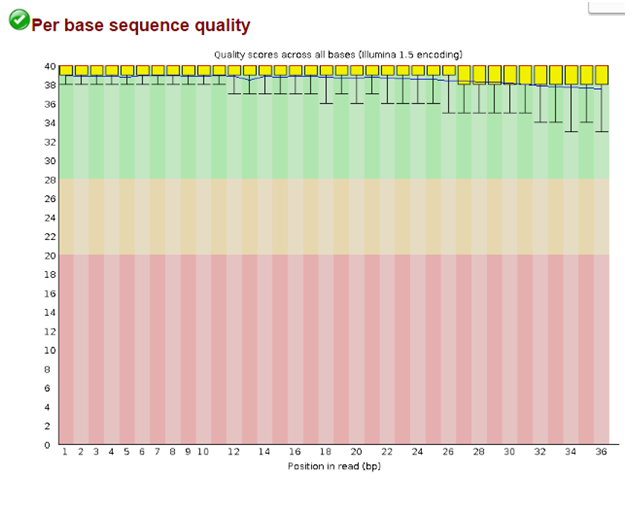
Рисунок 1. График качества чтений
Общее число чтений: 4463, длина ридов: 36. Качество ридов хорошее, так как все значения в зеленой области, поэтому чистить их программой Trimmomatic не нужно.
Затем было выполнено картирование прочтений на геном человека hg19.
Команда: bwamem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk1.fastq > chipseq_chunk1.sam.
Затем был произведен анализ полученного выравнивания из файла в формате sam при помощи следующих команд:
1) samtoolsview -bSochipseq_chunk1.bamchipseq_chunk1.sam (перевод выравнивания чтений с референсом в бинарный формат bam, -b–переводит в бинарный формат bam, -o– выходной файл, -S – входной формат файла определяется автоматически).
2) samtoolssortchipseq_chunk1.bam -Tchip_temp -ochipseq_chunk1.sorted.bam (сортировка выравнивания чтений с референсом по координате в референсе начала чтения, -Т – задание директории для временных файлов).
3) samtoolsindexchipseq_chunk1.sorted.bam (индексирование отсортированного bam-файла).
4) samtoolsidxstatschipseq_chunk1.sorted.bam>chipseq_chunk1.idxstats (выяснить, сколько чтений откартировалось на геном).
5) samtoolsview -cchipseq_chunk1.sorted.bam (общее количество откартировавшихся ридов).
Откартировалось 4463 рида, то есть все.
Затем была запущена программа для поиска пиков:
macs2callpeak -t chipseq_chunk1.sorted.bam
Однако пиков оказалось слишком мало, поэтому программа была запущена с другими параметрами:
macs2callpeak -tchipseq_chunk1.sorted.bam --nomodel -nchunk1 (из-за опции – nв названии полученных файлов в начале chunk1)
Были получены файлы chunk1_peaks.narrowPeak, chunk1_peaks.xls , chunk1_summits.bed и chunk1_peaks.xls, chunk1_summits.bed.
В файлchunk1_peaks.narrowPeak были добавлены строки:
track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="Peaks from chunk 17"
browserpositionchr12: 132060000-133000000
Всего было найдено 5 пиков. Визуализация найденного представлена на рисунке 2.
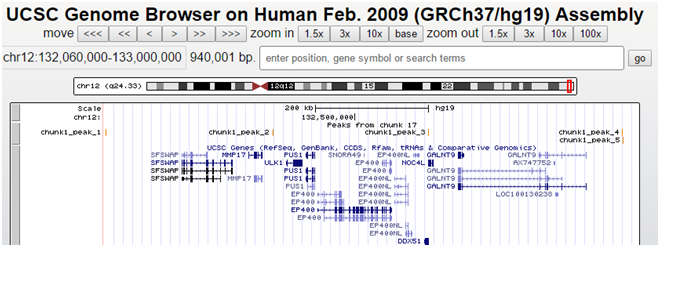
Рисунок 2. Визуализация информации о пиках при помощи UCSCGenomeBrowser (расширение файла narrowPeak)
Так же была произведена визуализация данных из файла с расширением .bed. Результат представлен на рисунке 3.
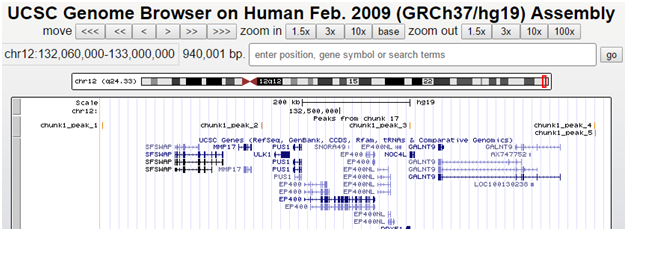
Рисунок 3. Визуализация информации о пиках при помощи UCSCGenomeBrowser (расширение файла bed)
Подробнее были исследованы пики 1, 3 и 5.
| Пик | 1 | 5 | 3 |
| Ширина пика | 575 | 208 | 299 |
| Достоверность пиков (-log10pvalue) | 33.74101 | 13.14490 | 33.72623 |
| Положение пика относительно начала | 163 | 93 | 156 |
Чем p-value меньше, тем достовернее пик, значит, чем больше –log10p-value, тем достовернее пик. Таким образом, пик 1 наиболее достоверен, а пик 5 наименее достоверен.
Пик 1 не перекрывается ни с какими генами (он слишком далеко расположен от генов). Пик 2 расположен на некотором расстоянии (но меньшем, чем в случае пика 1) от гена GALNT9 (кодирует член семейства UDP-N-acetyl-alpha-D-galactosamine: polypeptideN-acetylgalactosamintransferase). Рядом же с пиком 3 расположено множество различных генов: в частности, он расположен перед геном GALNT9 (EnsemblIDENSG00000182870), а также EP400 (человеческий белок, связывающий E1A), NOC4L (ядрышковый комплекс, связанный с 4 гомологом S.cerevisae), EP400 (EP400 N-terminallike, некодирующая РНК), HumanGeneDDX51 (HomoSapiensDEAD (Asp-Glu-Ala-Asp) boxpolypeptide 51-DDX51, мРНК).
Задание 4. Поиск сигналов TATA-бокс связывающего белка (TBP) в геноме человека.
TATA-бокс связывающий фактор TBP - архейный и эукариотический белок, узнающий восьминуклеотидный сигнал в ДНК с консенсусом TATA(А/Т)A(A/T)(A/G). Не все промоторы имеют сигнал TATA-box.
Первый ген, для которого был найден ТАТА-бокс:
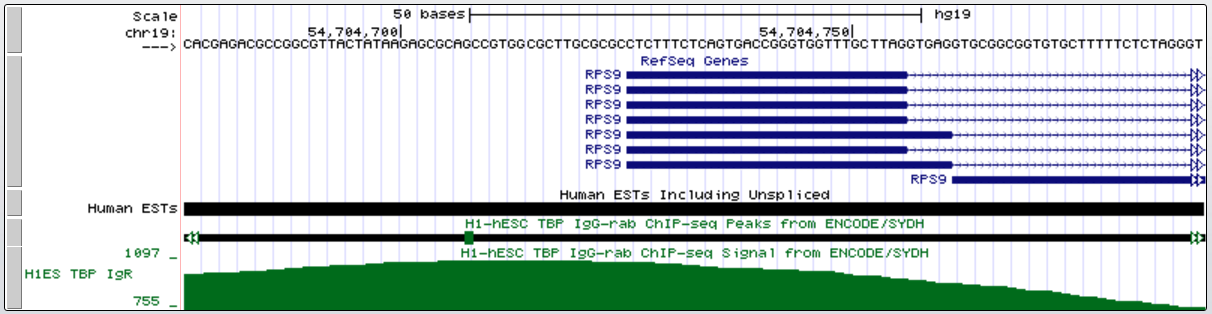
промоторная область в крупном масштабе
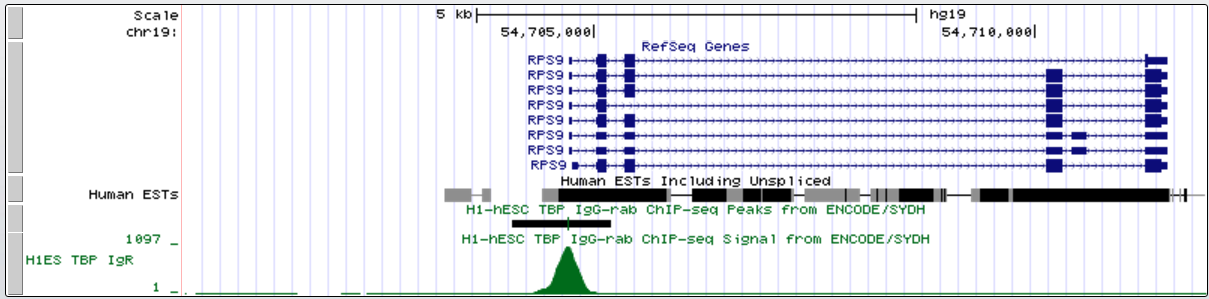
промоторная область в мелком масштабе
| Ген | Координаты старта транскрибции | Длина гена | Координаты ТАТА-бокса | Последовательность ТАТА |
| Homo sapiens ribosomal protein S9 (RPS9), transcript variant 6 | chr19:на прямой цепи:54 704 725 | 6 651 | 54 704 695 | ТАТААGAG |
Второй ген, для которого был найден ТАТА-бокс:
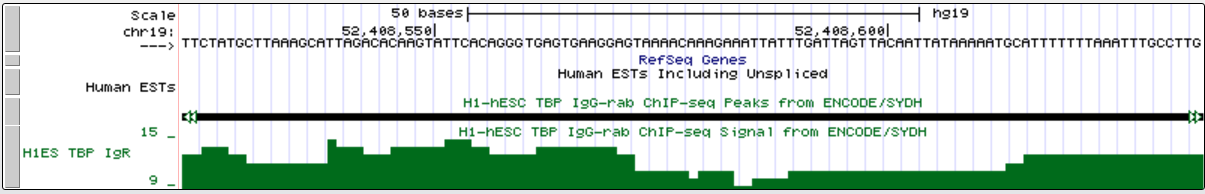
промоторная область в крупном масштабе
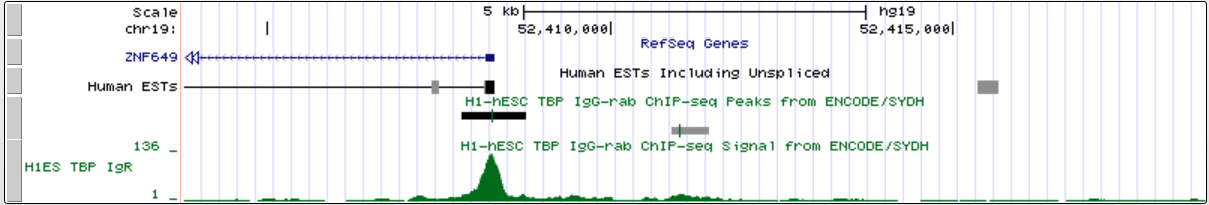
промоторная область в мелком масштабе
| Ген | Координаты старта транскрибции | Длина гена | Координаты ТАТА-бокса | Последовательность ТАТА |
| Homo sapiens ZNF649 antisense RNA 1 (ZNF649-AS1) | chr19:на обратной цепи:52 391 278 | 17 026 | 52 408 605 | ТАTAAAAAT |
Третий ген, для которого был найден ТАТА-бокс:
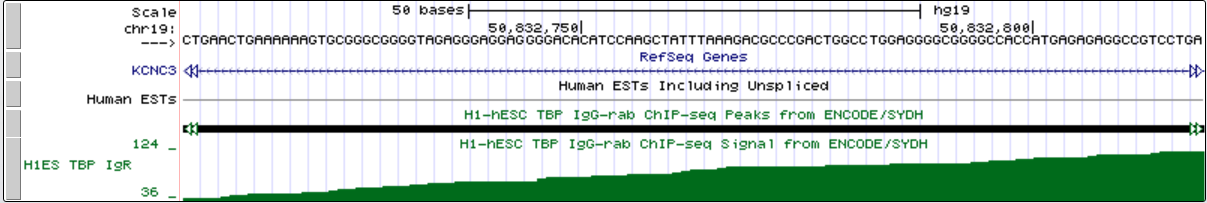
промоторная область в крупном масштабе
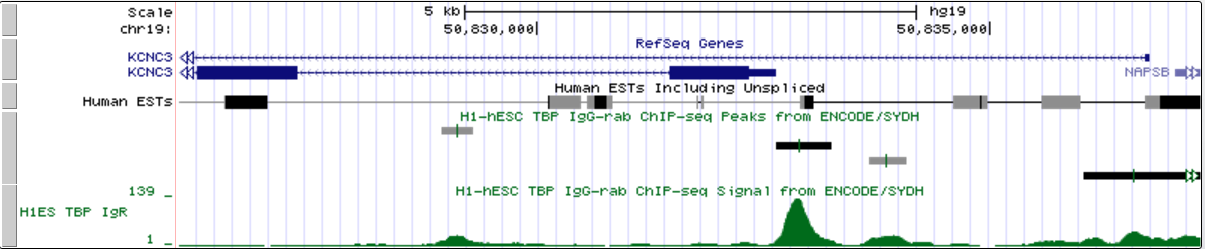
промоторная область в мелком масштабе
| Ген | Координаты старта транскрибции | Длина гена | Координаты ТАТА-бокса | Последовательность ТАТА |
| Ген | chr19:на обратной цепи: 50 832 633 | 13 869 | 50 832 762 | TATTTAAA |
Во всех трех описанных случаях сигнал TATA-box расположен перед началом гена на довольно большом расстоянии (100 нуклеотидов). Часто было так, что рядом с генами был сигнал TBP, а в последовательности был не мас ТАТА-бокс, а бласть очень похожую на него.
Белков без сигнала TATA-бокса в промоторной области было намного больше, чем с. Вот пример одного из них:
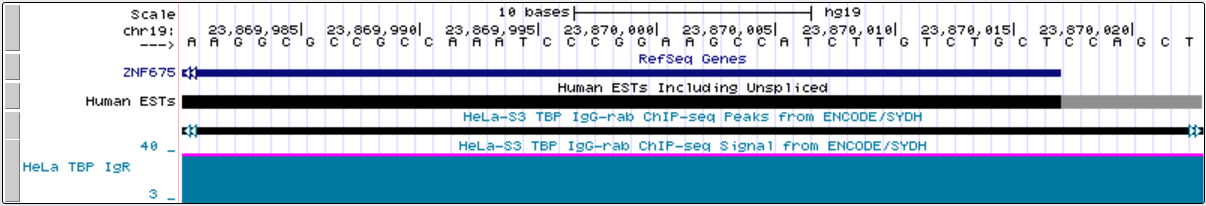
промоторная область в крупном масштабе
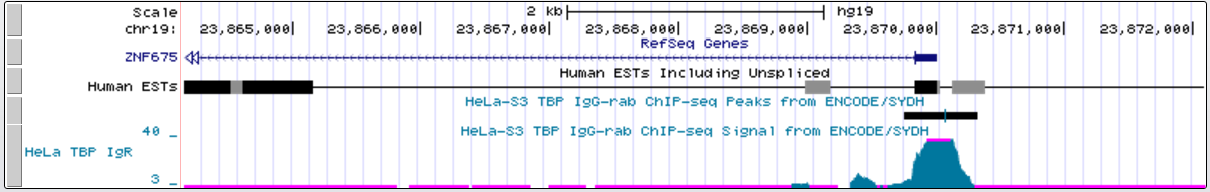
промоторная область в мелком масштабе
СПАСИБО ЗА ПРОСМОТР
© Мария Медведева