В рамках данного практикума был исследован предполагаемый фосфорибозил трнсферазный домен PF00156.
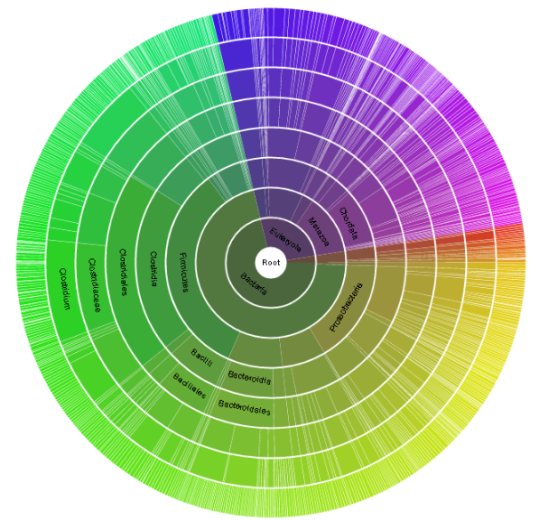

Представители с данным доменом.
Этот домен содержит 73 доменные архитектуры.
Проект JalView содержит выравнивание всех последовательностей, содержащих данный домен.
Для дальнейшего рассмотрения были выбраны архитектуры, изображённые на рисунке:
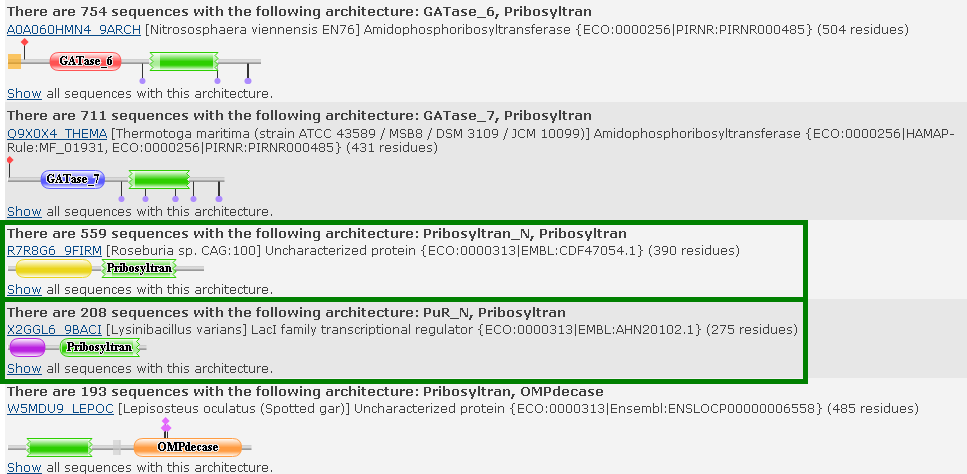
Архитектура "1" - содержащая домены Pribosyltran_N, Pribosyltran (559 последовательностей)
Архитектура "2" - содержащая домены PuR_N, Pribosyltran (208 последовательностей)
Далее была получены таблица, содержащая информацию о доменной структуре каждой последовательности, содержащей данный домен. Это было сделано с помощью скрипта swisspfam-to-xls.py. Команда: "python swisspfam-to-xls.py -z /srv/databases/pfam/swisspfam.gz -m domain_AC.txt -o PF00156.txt". Файл domain_AC.txt должен содержать идентификатор исследуемого домена.
Также были получены сведения о таксономии каждой последовательности. Для этого сначала (из файла PF000156.txt) был получен список идентификаторов последовательностей ), затем из базы данных uniprot были скачаны соответствующие записи (файл). И затем с помощью другого скрипта (команда: "python uniprot_to_taxonomy.py -i PF00156_uniprot.txt -o PF00156_taxonomy.txt") был получен файл с таксономией.
На основе всех этих данных с помощью скрипта и ресурсов Excel была получена сводная таблица, содержащая список последовательностей с указанием их доменной архитектуры, длины домена PF00156 и таксономии.
В качестве таксонов, в которых представлены домены этих белков я выбрала два царства (Bacteria, Eukaryota), общий для них таксон - Cellular organisms.
Затем было выбрано примерно по 25-30 представителей с архитектурами "1" и "2" (выбирались последовательности как первого вхождения домена, так и второго).
Соответствующие им последовательности были вырезаны из общего выравнивания с помощью скрипта, открыты в Jalview. Были удалены плохо выравненные последовательности и небольшие участки с C и N концов. Проект получившегося выравнивания можно скачать здесь. Выравнивание в фаста-формате - vur.fasta.
Задание 2. Построение дерева.
По полученному выравниванию в программе MEGA было построено филогенетическое дерево последовательностей методом максимального правдоподобия.
Скобочная формула дерева: здесь.
Изображение дерева:
На дереве буква "B" в названии последовательности означает, что она принадлежит типу Бактерий, "E" - Эукариот. Цифры "1" и "2" - обозначения доменов (вводились выше).
По дереву видно, что последовательности с разными архитектурами чётко разделились на 2 клады. (розовая и зелёная рамки).
Интересно наличие эукариотных организмов (оранжевая рамка) среди бакерий с архитектурой "2"( зелёная рамка). Ещё также интересно, как в последовательностях с первой архитектурой произошло разделение на бактерий и эукарирот (голубая рамка). Присутствие бактериального организма в кладе с голубой рамкой можно объяснить либо ошибкой на этапе определения последовательности, либо явлением горизонтального переноса.
Задание 3. Построить профиль подсемейства и охарактеризовать качество его работы .
Для построения профиля в качестве подсемейства последовательностей были выбраны последовательности из клады, выделенной голубой рамочкой (т. к. они отделены от остального дерева).
Выравнивание всех этих последовательностей можно посмотреть здесь.
По ним с помощью программы hmm2biuld был построен профиль последовательностей и затем он был откалиброван (программа hmm2calibrate). Результат работы этих двух программ можно увидеть здесь. По этому профилю был проведен поиск по всем белкам, включающим семейство доменов Phosphoribosyl transferase с помощью программы hmm2search без каких-либо дополнительных параметров.
Результат поиска можно увидеть здесь.
Данные о всех находках и их E-value были перенесены в таблицу Excel. Находки были отсортированы по увеличению E-value. Далее с помощью функции vlookup последовательности (среди всех находок), участвовавшие в построении профиля были отмечены как "ИСТИНА", а все остальные находки - как "ЛОЖЬ".
Далее рассчитываются параметры чувствительности и специфичности поиска. Для их расчёта найдём TP, TN, FP, FN. Для каждого значения веса предположим, что именно оно является порогом, и посчитаем 4 переменных: 1) количество последовательностей, которое выше этого порога и достоверно содержит искомый домен (переменная TP - TruePositive) , 2) количество последовательностей, которое ниже порога и достоверно не содержит домен (TN - TrueNegative), 3) количество последовательностей, которое выше порога, но на самом деле не содержит домена (FP - FalsePositive), и 4) количество последовательностей, которое ниже порога, но достоверно содержит домен (FN - FalseNegative).
Чувствительность поиска — это доля достоверно предсказанных белков, содержащих домен, от общего количества последовательностей.
A специфичность — доля достоверно предсказанных белков, не содержащих домен, от общего количества последовательностей.
Далее строится график зависимости чувствительности от параметра 1 минус специфичность.
В итоге получаем ROC-кривую.
Таблица Excel, в которой производились расчёты - здесь.
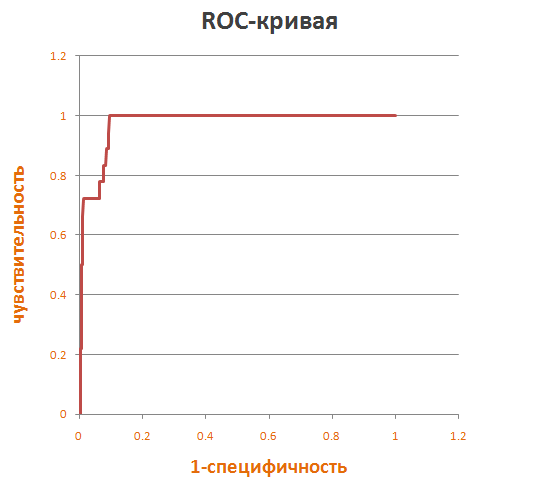
С помощью ROC-кривой я выбрала порог E-value, по которому можно судить о принадлежности последовательности подсемейству.
Пороговое значение E-value оказалось равным 7,6E-43.
Значения при выбранном пороге:
| На самом деле | приналежит семейству | не приналежит семейству | сумма |
|---|---|---|---|
| Выше порога по профилю | 14 | 45 | 59 |
| Ниже порога по профилю | 6 | 2906 | 2912 |
| Сумма | 20 | 2951 | 2971 |
СПАСИБО ЗА ПРОСМОТР
© Мария Медведева