Практикум 6Определение вторичной структуры |
|||||||||||||||||||
|
|
В данном практикуме нужно было сравнить предсказания вторичной структуры белка PyrR,
который регулирует экспрессию "pyr" генов в организме многих бактерий. Предсказания строились с помощью
алгоритмов DSSP и Stride.
2. Сравнение границ четырёх элементов вторичной структуры.
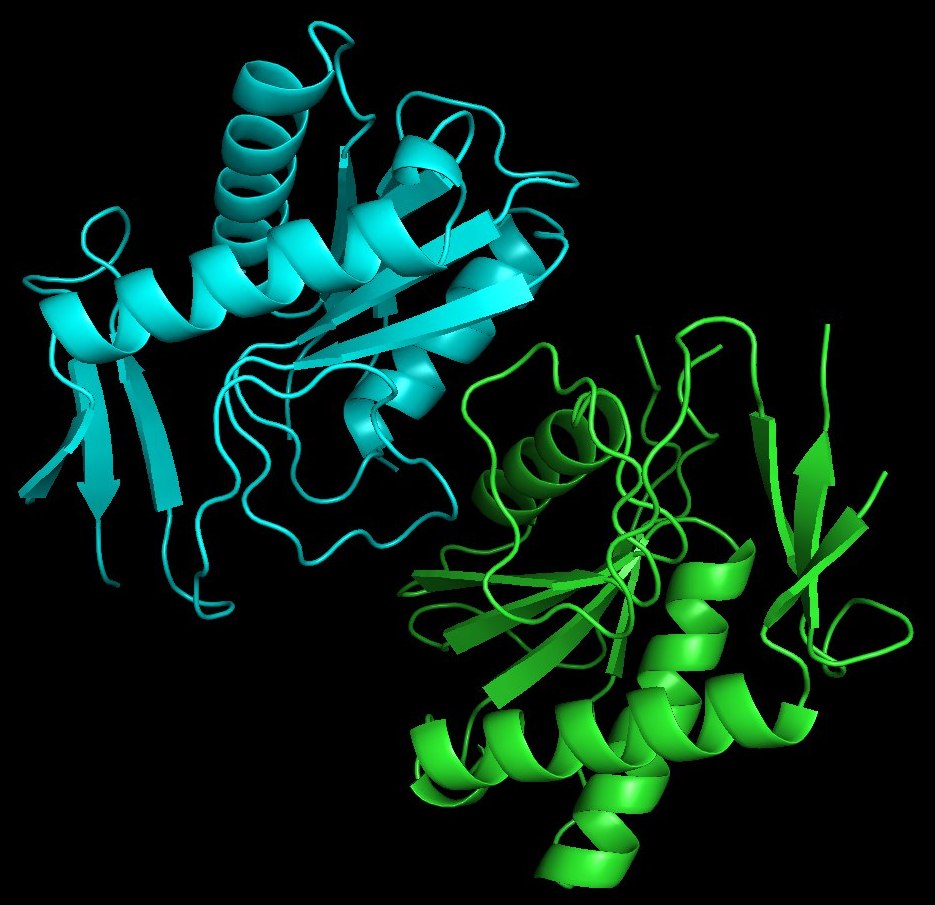
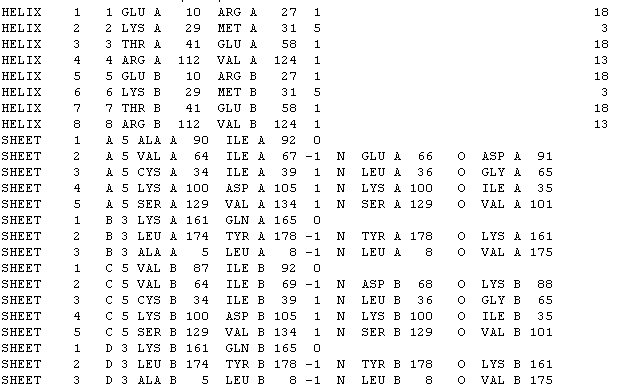
Из таблицы можно заметить, что границы элементов вторичной структуры, предсказанные DSSP, практически совпадают с границами из PDB. Координаты всех (в том числе и не рассмотренных в таблице) α-спиралей совпадают полностью. В случае β-листов координаты не совпали: для выбранных структур в таблице совпали только координаты их начала, но есть посмотреть и на координаты других β-листов, то видно, что координаты очень "скачут". 3. Описание редких элементов. При рассмотрении структуры с помощью DSSP, было найдено несколько редких элементов: один β-мостик (ILE125), например (Рис. 4). 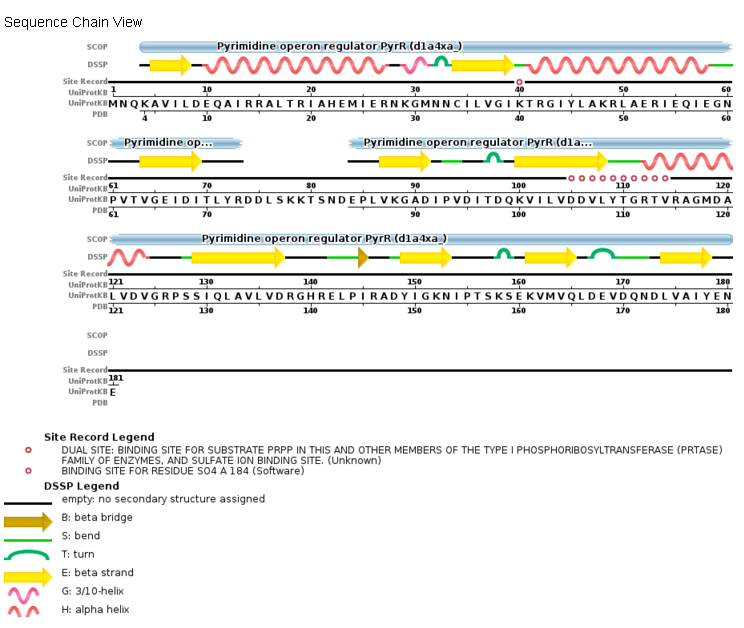 Рисунок 4. Выдача DSSP. В структуре содержится 4 поворота: 32-33, 97-98, 158-159 и 167-169. Все повороты маленькие - размером всего в 2-3 аминокислоты, не сильно разворачивают структуру. Сравнение аннотаций, полученных алгоритмами DSSP и STRIDE (Рис. 5):  Рисунок 5. Выдача DSSP и STRIDE. Видно, что в выдачах обоих алгоритмов совпадают основные структурные единицы, такие как α-спирали и β-листы. И β-мостик находится в том же положении. А вот количество поворотов и их координаты сильно "гуляют". |
||||||||||||||||||
|
© Мария Медведева 2017 |
|||||||||||||||||||