|
|
1. Поиск гидрофобных кластеров в структуре белка с pdb-кодом 5OWI.
Для поиска гидрофобных кластеров использовался сервер
CluD. В ходе работы изменялись параметры: порог расстояния и порог размера кластера, чтобы получить
наилучший результат для интерпретации. Согласно данным из UniProt в белке содержится
три домена (N-концевой, связывающий рибосому, срединный с пептидил-пролил-цис/транс изомеразной активностью и
С-концевой домен, связывающий субстрат), интересно, будет ли наблюдаться корреляция между количеством и расположением
этих доменов в белке и количеством и расположением гидрофобных кластеров.
При пороге дистанции (в пределах которой атомы объединяются в один кластер) 5.4Å и
параметром размера кластера 3 (не показывать кластеры менее трех атомов) было найдено 66 кластеров,
из которых 24 состоят из 3 атомов, 10 ядер - из четырёх атомов, 5 ядер - из пяти, 5 ядер - из шести,
3 ядра - из восьми, оставшиеся - больше девяти. Крупных ядер, состоящих из больше, чем 20 атомов, - 12.
Самое крупное 48-ое ядро состоит из 233 атомов (Рис. 1).
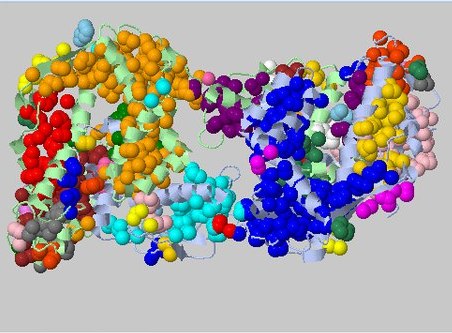 
Рисунок 1. Выдача CluD при пороге дистанции в 5.4Å и пороге размера кластера 3.
Показаны все найденные ядра.
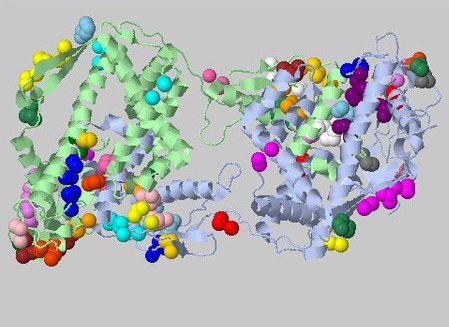
Рисунок 2. Выдача CluD при пороге дистанции в 5.4Å и пороге размера кластера 3.
Показаны ядра меньше, чем в 20 атомов.
На рис. 3 с кластерами, состоящими из меньше, чем восьми атомов,
заметно, что все они принадлежат петлевым участкам.
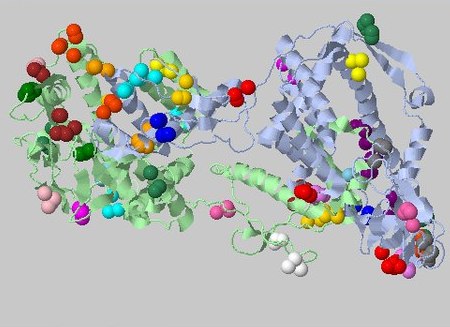
Рисунок 3. Выдача CluD при пороге дистанции в 5.4Å и пороге размера кластера 3.
Показаны ядра меньше, чем в 8 атомов.
При таких параметрах (при пороге дистанции в 5.4Å и пороге размера кластера 3)
появляется очень много "маленьких" ядер - попробуем дальше поварьировать параметры.
Попробуем снизить порог дистанции до 4.
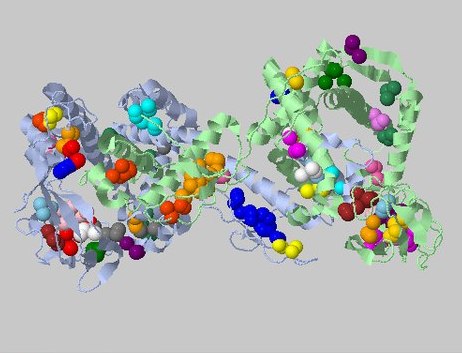
Рисунок 4. Выдача CluD при пороге дистанции в 4Å и пороге размера кластера 3.
Показаны все ядра.
С новым порогом CluD нашёл 59 гидрофобных ядер, из которых 3 ядра из 6 атомов, 5 ядер из 5 атомов, 14 ядер
содержат по 4 атома и 29 ядер из 3 атомов. Самое крупное ядро состоит из 18 атомов (Рис. 4).
Теперь попробуем, наоборот, повысить порог димтанции до 7.
При таком пороге действительность тоже искажается - сервер нашёл всего 4 гидрофобных ядра, причём из них два ядра по 3 атома, одно из 6 атомов, а
самое крупное включает в себя 1864 атома (Рис. 5).
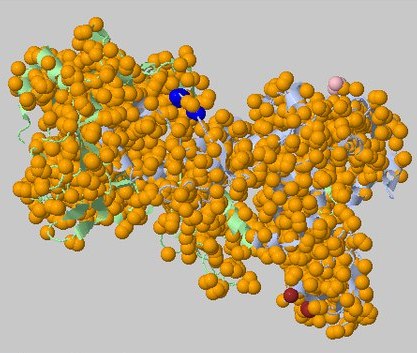 
Рисунок 5. Выдача CluD при пороге дистанции в 7Å и пороге размера кластера 3.
Показаны все ядра.
При пороге дистанции в 6Å находится 28 ядер, из которых размер самого большого составляет 1610 атомов (Рис. 6).
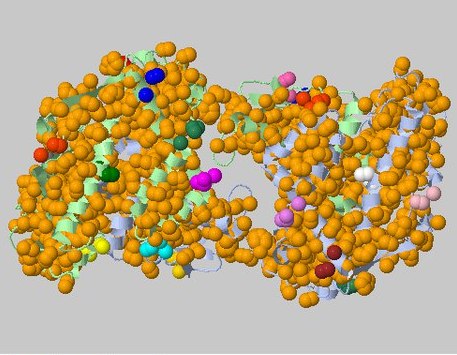
Рисунок 6. Выдача CluD при пороге дистанции в 6Å и пороге размера кластера 3.
Показаны все ядра.
На данном этапе самыми правдоподобными оказались "дефолтные" параметры. Сравним тогда выдачи, полученные
с близкими к ним параметрами.
При пороге дистанции в 5Å в выдаче содержится 73 ядра, самое большое из которых состоит из
1610 атомов и опять большое количество маленьких кластеров по 3 атома (Рис. 7).
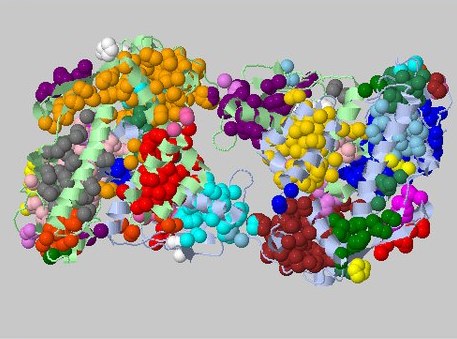
Рисунок 7. Выдача CluD при пороге дистанции в 5Å и пороге размера кластера 3.
Показаны все ядра.
При пороге в 5.8Å: всего находится 37 ядер, из них 4 крупных по 716, 696, 57 и 23 атома (Рис. 8).
А при пороге в 5.6: всего 50 ядер. 21 кластер содержит как минимум 6 атомов.
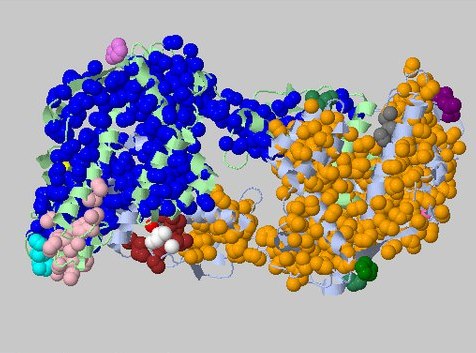
Рисунок 8. Выдача CluD при пороге дистанции в 5.8Å и пороге размера кластера 3.
Показаны все ядра.
При пороге дистанции в 5.6Å видно, что в один кластер попали участки из разных цепей (синий кластер на Рис. 9).
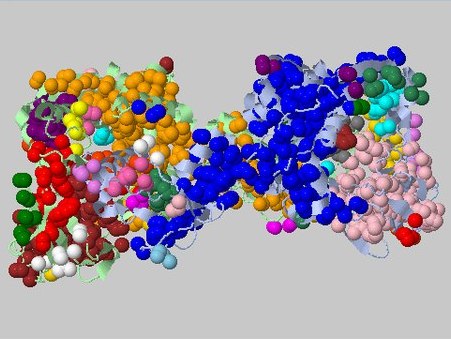
Рисунок 9. Выдача CluD при пороге дистанции в 5.6Å и пороге размера кластера 3.
Показаны все ядра.
В целом, видна следующая тенденция: при уменьшении порога дистанции крупные кластеры распадаются
на более мелкие, а при его увеличении маленькие кластеры объединяются в большие.
Мне кажется, в нашем лучае наиболее оптимальными оказались "дефолтные" параметры.
2. Поиск гидрофобных кластеров на интерфейсе двух цепочек в структуре 2NQB.
Были найдены гидрофобные кластеры в структуре 2NQB.
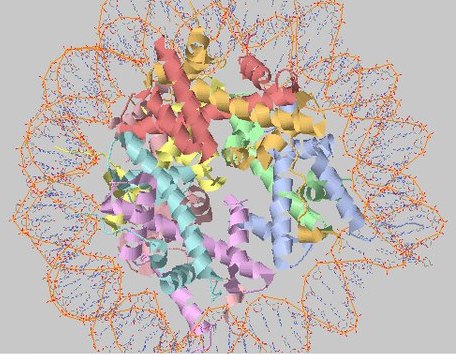
Рисунок 10. Структура белка с pdb-кодом 2NQB.
При запуске с исходными параметрами 5.4Å и >3 атома было найдено 39 кластеров, принадлежащих белку.
Один кластер ( 1 core) принадлежит ДНК.
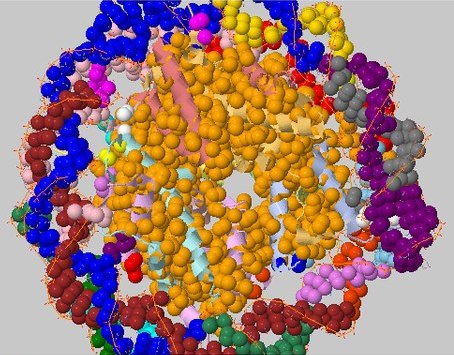
Рисунок 11. Выдача CluD при пороге дистанции в 5.4Å и пороге размера кластера >=3.
Видно, что в белке есть гидрофобные кластеры для взаимодействия с ДНК.
При близких порогах (5.6Å и 5.2Å дистанции) программа находит 20 и 41 гидрофобное ядро соответственно.
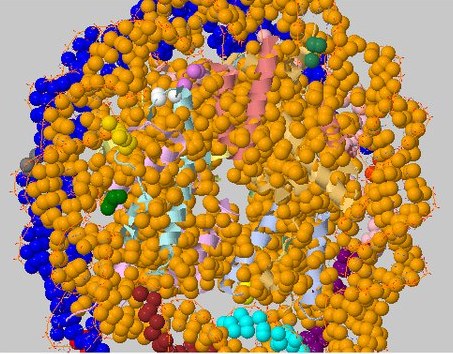
Рисунок 12. Выдача CluD при пороге дистанции в 5.6Å и пороге размера кластера >=3.
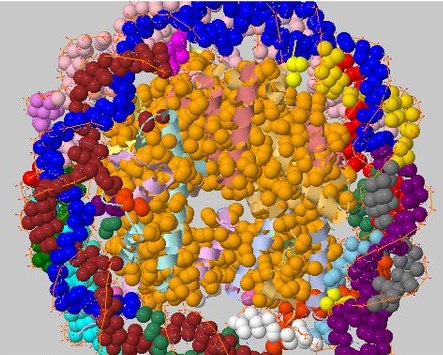
Рисунок 13. Выдача CluD при пороге дистанции в 5.2Å и пороге размера кластера >=3.
При варьировании параметров до 5.6Å - кластер ДНК объединяется с
кластером белка в один, что не является верным. При уменьшении порога (ниже 5Å) большие кластеры разделяются на
более маленькие, при этом не удалось добиться хорошего разделения кластера, принадлежащего ДНК.
Таким образом, оптимальным является запуск с исходными параметрами и с параметром дистанции в 5.2Å. Сравним их
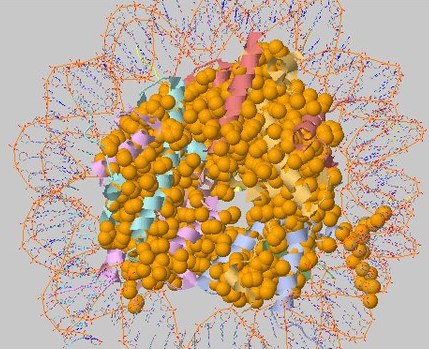
Рисунок 14. Выдача CluD при пороге дистанции в 5.4Å и пороге размера кластера >=3. Показаны
гидрофобные кластеры белковой части молекулы.
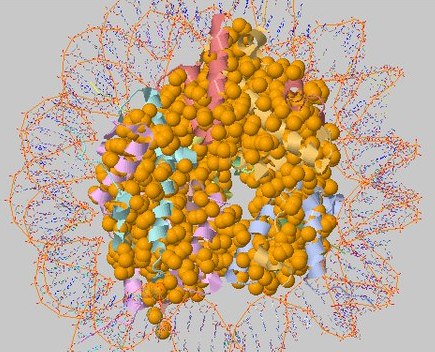
Рисунок 15. Выдача CluD при пороге дистанции в 5.2Å и пороге размера кластера >=3. Показаны
гидрофобные кластеры белковой части молекулы.
При пороге в 5.4Å часть одного белкового кластера "налезает" на ДНК-кластеры, поэтому, мне кажется,
в случае этого белка оптимальнее использовать не априорные параметры, а дистанцию в 5.4Å.
Рисунок 16. Выдача CluD при пороге дистанции в 5.2Å и пороге размера кластера >=3. Показаны
гидрофобные кластеры ДНК-части молекулы.
|


















