BLAST
Задание 1.
BLASTp — это алгоритм для поиска гомологичных последовательностей белков по белковой последовательности. Для наиболее эффективного поиска в настройках этого алгоритма существует множество параметров. Ниже я напишу для чего они нужны.
Database — база данных из которой выбираются гомологичные белки.
Organism — организм по которому будет производиться поиск.
Exclude — благодаря этой опции можно исключить из поиска некоторые группы белков (Models (XM/XP), Non-redudant RefSeq proteins (WP), Uncultured/enviromental sample sequences).
Algorithm — параметр для выбора алгоритма поиска (быстрый, обычный писк по последовательности, алгоритм для построения позиционной весовой матрицы, поиск по шаблону и поиск в базе данных "сохраненных доменов" с построением позиционной весовой матрицы по полученным данным).
Max target sequences — максимальное количество отображаемых выровненных последовательностей.
Short queries — возможность подключить автоматический подбор параметров для наиболее эффективного поиска, проводимого для коротких последовательностей.
Expect threshold — этот параметр определяет допустимое количество случайных совпадений. Значение этого параметра рассчитывается по формуле и учитывает вес выравнивания, длину исходной последовательности и размер базы данных. Чем меньше значение этого параметра, тем меньше вероятность случайных совпадений. Но бывают и исключения: иногда белок выравнивание с которым имеет высокое E-value может быть более гомологичным с исходным, чем белок у которого оно сильно ниже.
Word size — параметр, определяющий длину участков, на которые разбиваются последовательности и которые потом сравниваются, на сравнении этих участков базируются общие выравнивания последовательностей. Можно регулировать чувствительность и скорость поиска уменьшая и увеличивая этот параметр.
Max matches in a query range — параметр, позволяющий ограничить количество совпадений, чтобы увидеть совпадения по более слабой части запроса, исключив совпадения по более сильной.
Matrix — параметр для выбора матрицы весов замен.
Gap Costs — стоимость появления и продления гэпов.
Compositional adjusments — функция для минимизации вероятности учитывания участков малой сложности.
Filter — этот параметр маскирует участки низкой сложности (например, участки, не имеющие биологического интереса).
Mask — тут есть 2 пункта. Первый маскирует участки малой сложности только для построения таблицы поиска. Второй маскирует строчные буквы в FASTA файле.
Для некоторых других алгоритмов BLAST существуют дополнительные параметры, но здесь я про них не напишу.
При помощи BLAST я провела поиск гомологов белка эндоглюконазы бактерии Acidothermus cellulolyticus. Сначала я получила общую таблицу и для удобства перевела ее в формат excel (скачать таблицу). Потом из всех предложенных программой белков я выбрала несколько для построения множественного выравнивания. При выборе белков я отсортировала их по покрытию выравнивания и старалась найти белки с как можно более разнообразными названиями, а также выбрала несколько белков с относительно большой E-value. Когда я построила множественное выравнивание всех этих белков, то все белки с более менее большим E-value оказались негомологичными. Это стало понятно, когда в программе Jalview использовала функцию покраски выравнивания above identity threshold на 90%. В изначально полученном мной выравнивании практически не было цветных столбцов, но, после удаления некоторых последовательностей, появились достаточно большие гомологичные куски.
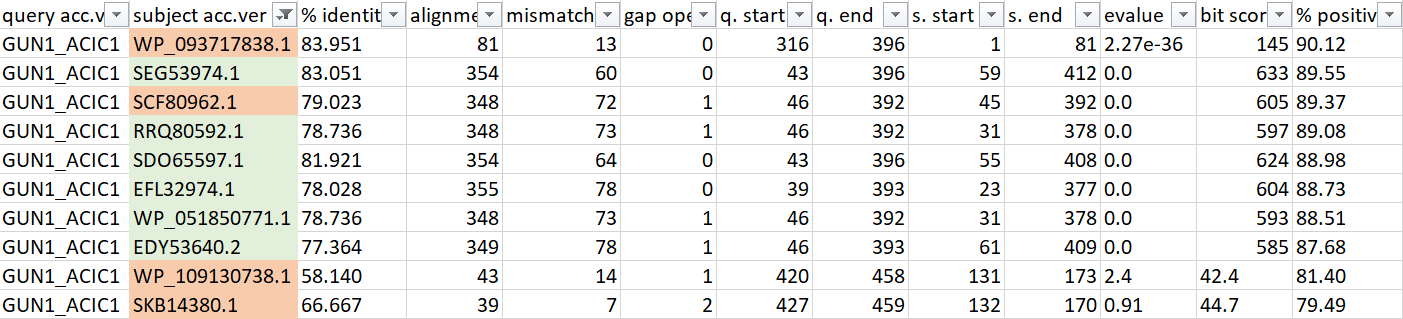
Рисунок 1.
На рисунке 1 изображена часть таблички с изначально выбранными мной белками, а красным цветом отмечены белки, которые в общем выравнивании оказались негомологичными.
На рисунках 2 и 3 представлены некоторые гомологичные участки множественного выравнивания. Само выраснивание можно скачать здесь.
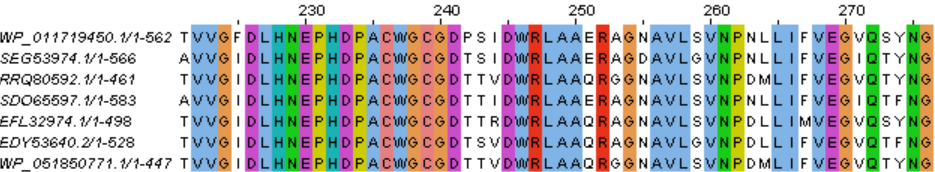
Рисунок 2.
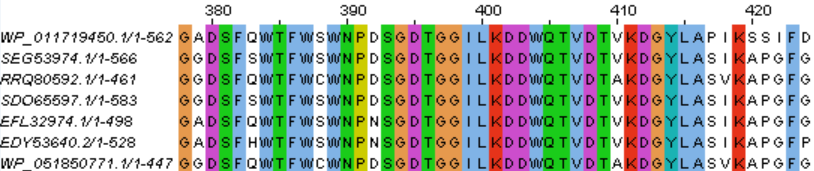
Рисунок 3.
В итоге я нашла 6 белков гомологичных исходному. Их гомологичность проявляется в том, что в этом множественном выравнивании достаточно большая плотность консервативных позиций и есть несколько участков, длиной 6 и более колонок, без гэпов, где все позиции консервативны. Например участки с 226 по 241, с 245 по 250 позиции на рисунке 2 и с 393 по 409, с 411 по 416 позиции на рисунке 3.
Задание 2.
Это задание посвящено карте локального сходства. Из предложенных групп белков я выбрала 2 для сравнения: 7,8-дигидронеоптерин альдолазу из организма Thermaerobacter marianensis и неохарактиризованный белок из организма Leptosphaeria maculans.
Воспользовавшись функцией для выравнивания двух и более последовательностей, я загрузила последовательности выбранных мной белков в FASTA формате в BLASTp и построила карту локального сходства, которую можно увидеть на рисунке 4.
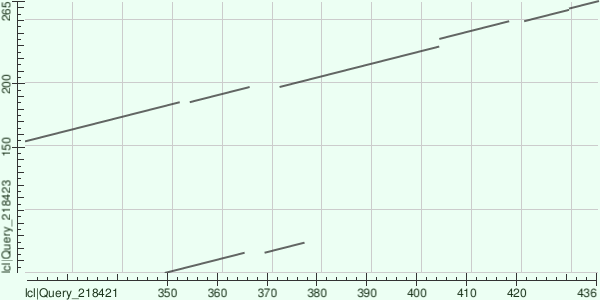
Рисунок 4.
На этой карте изображены два локальных выравнивания, сделанных бластом (рисунок 5). Нижнее выравнивание очень короткое, поэтому я не буду его рассматривать, это же выравнивание представлено нижней линией на карте локального сходства. В общих словах карта локального сходства отражает области сходства последовательностей. В данном случае по оси OX располагается участок последовательности 7,8-дигидронеоптерин альдолазы (первая последовательность), а по OY - неохарактеризованного белка (вторая последовательность).
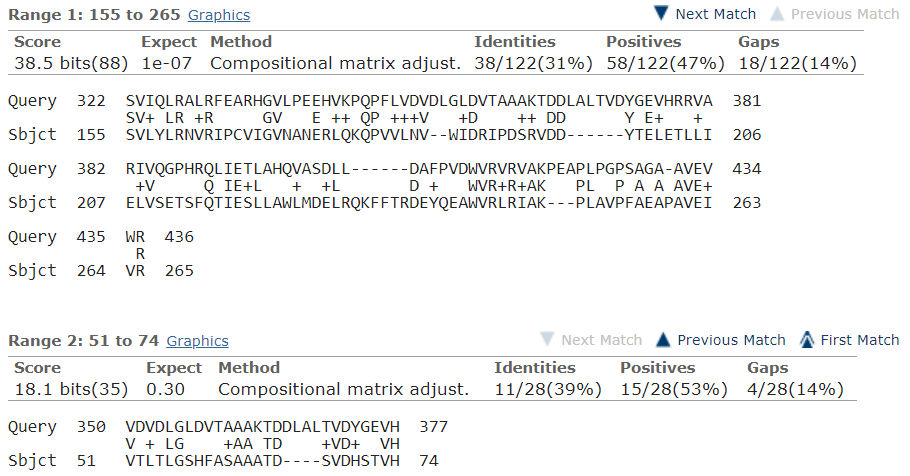
Рисунок 5.
Покрытие рассматриваемого выравнивания 47%. Эти белки определенно нельзя считать гомологичными. Исходя из карты локального сходства можно сказать, что последовательности выравниваются не полностью, в том числе и из-за разной длины и что во второй последовательности произошло три делеции, а в первой - 2. Идентичность этих последовательностей 31%, что немало, но не дотягивет до гомологичности, тем более это могли быть отчасти случайные совпадения, потому что по-настоящему гомологичных участков я не вижу.
Задание 3.
Последнее задание этого практикума позволяет нам немного пощупать границы возможностей этого алгоритма и просто поближе с ним познакомиться.
Сначала я решила узнать, что же BLAST будет делать не с белковой последовательность, а с обычным текстом. В качестве фразы для эксперимента я выбрала пару строчек из одной из моих любимых песен: "NOS CORPS a la derive dans un OCEAN DE LARMES". К сожалению, ни одного белка гомологичного этой фразе не нашлось.
Затем я попробовала провести поиск гомологов своего белка в базе данных SwissProt. Результатов стало значительно меньше по сравнению с поиском описанным в первом задании, хотя я специально выставила количество отбражаемых последовательностей 20000, как в прошлый раз. Еще в этот раз стало гораздо меньше выравниваний с весом более 200, которые преобладали опять же при поиске в первом задании.
Я попробовала ограничить число находок BLAST в одной последовательности из банка и не трогала остальные параметры (только опять выставила 20000 отображаемых последовательностей). В результате поиска BLAST выдал мне не очень много последовательностей. Причем, несмотря на отсутствие ограничений, не было ни одной последовательности с E-value больше 7e-15.
Изменив длину участков Word Size с 6 на 3, я не заметила особых изменений в результатах поиска.
После активации маски участков малой сложности количество выравниваний весом больше 200 на участке последовательности после приблизительно 400 аминокислоты очень сильно сократилось. А участок от 400 до 450 аминокислот перестал выравниваться с чем либо.
Как итог, я могу сказать, что в зависимости от требующейся точности поиска, его объемности, учитываемости тех или иных участков последовательностей, можно подстроить этот алгоритм под свои цели, чтобы максимально эффективно достичь своих целей.