Визуализация, паралоги
Составление списка гомологичных белков, включающих паралоги
Целью этой части задания было найти достоверные гомологи белка CLPX_ECOLI среди белков выбранных мной ранее бактерий (см. практикум 1).
Чтобы найти гомологов я воспользовалась необходимыми мне файлами из папки /P/y18/term4/Proteomes. При помощи команды cat *.fasta > proteomes.fasta я обьединила мои файли в один. Затем, использовав команду makeblastdb -dbtype prot -in proteomes.fasta -out proteomes я проиндексировала содержание файла, чтобы использовать его в качестве database для бласта. После этого я сделала поиск бластом по последовательности данного белка. Команда: blastp -query CLPX_ECOLI.fasta -db proteomes -evalue 0.001 -out blastp.txt -outfmt 7. Скачать выдачу blastp. Только ID выданных бластом белков.
При помощи полученных ID и сайта uniprot.org я скачала последовательности всех необходимых мне белков. Скачать файл с последовательностями белков.
Оставив в файле только названия белков и организмов, я выровняла последовательности командой muscle. Выравненные белковые последовательности.
Реконструкция и визуализация
При помощи программы MEGA и используя метод максимального правдоподобия я реконструировала дерево найденных гомологов. Скачать скобочную формулу дерева.
Опираясь на получненное дерево я нашла три пары ортологов (белки из разных организмов или если разделение их общего предка на линии, ведущей к ним, произошло в результате видообразования) и три пары паралогов (гомологичные белки из одного организма).
Пары ортологов:
- Q8YAC6_LISMO & Q5HRP3_STAEQ
- HSLU_STAA8 & HSLU_STAEQ
- Q5FKR6_LACAC & CLPX_ENTFA
Пары паралогов:
- A5I7Q0_CLOBH & A5I766_CLOBH
- Q891B9_CLOTE & Q898D1_CLOTE
- Q8Y8B1_LISMO & Q8YAC6_LISMO
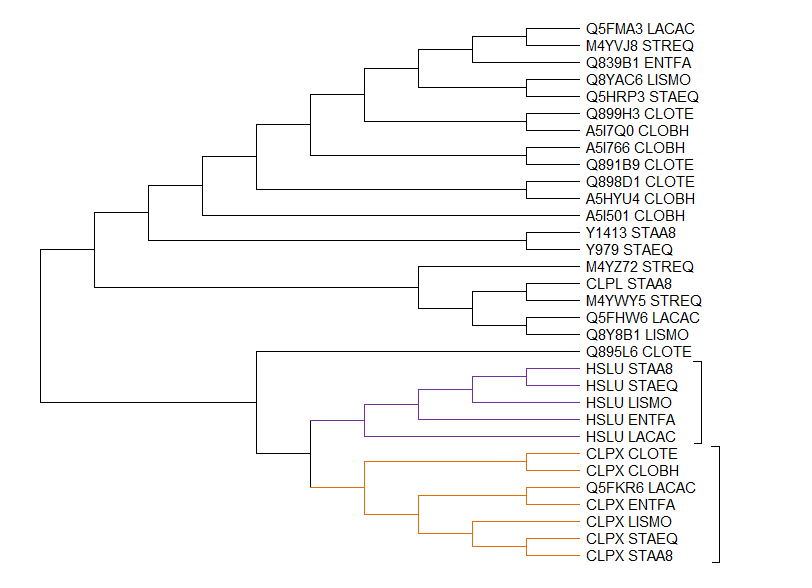
Рисунок 1. Филогенетическое дерево с выделенными группами ортологов.
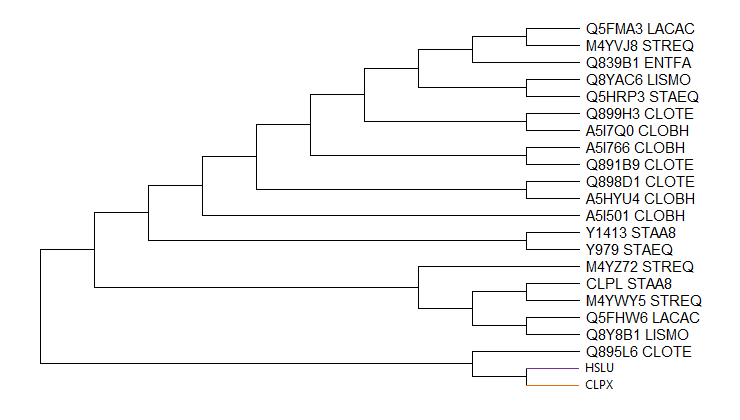
Рисунок 2. Филогенетическое дерево с объединенными группами ортологов. Группа HSLU: HSLU_STAA8, HSLU_STAEQ, HSLU_LISMO, HSLU_ENTFA, HSLU_LACAC. Группа CLPX: CLPX_CLOTE, CLPX_CLOBH, Q5FKR6_LACAC, CLPX_ENTFA, CLPX_LISMO, CLPX_STAEQ, CLPX_STAA8.