AlphaFold2
Амилоиды - это агрегаты белков, обладающих характерной фибриллярной морфологией, образованной бета-листами (cross-β). В организме человека амилоиды связаны с развитием различных патологий: амилоидозов или нейродегеративных заболеваний. Например, болезнь Альцгеймера, болезнь Паркинсона, болезнь Хантингтона и т.д. Патогенные амилоиды образуются, когда нормальный белок (или фрагмент белка) теряет свою структуру и функции и образует фиброзные отложения в амилоидных бляшках. Однако, амилоиды могут иметь и нормальные биологические функции.
В данном практикуме был использован ColabFold для того, чтобы предположить структуру для аминокислотной последовательности амилоида. Поиск по последовательности в UniProt показал, что данный амилоид является фрагментом связанного с микротрубочками белка тау. Белок тау стабилизирует микротрубочки. Нарушение сети микротрубочек, которое может быть вызвано потерей функции тау-белка, наблюдается в группе родственных заболеваний, называемых таупатиями, включая болезнь Альцгеймера. С помощью Protein BLAST я нашла несколько структур в базе данных PDB, части которых соответствовали данной последовательности. Три из топ-5 структур относятся к филаментам из ткани человеческого мозга с болезнью Альцгеймера. У всех топ-5 записей из PDB одинаковые структуры. Все 5 структур были получены методом электронной микроскопии.
Я выбрала в качестве референса структуру 6HRE (рис. 1) с разрешением 3.20 Å. Clashscore структуры 6, что является нормальным показателем для структур с данным разрешением. Маргиналов по Рамачандрану или по ротамерам боковых остатков в данной структуре нет. Структура состоит из 6 цепей, к сожалению, мономерной структуры для исходной последовательности найти не удалось.

Рисунок 1.
Первое предсказание структуры с помощью ColabFold было проведено для мономера. Предположительно, мономер не обладает вторичной структурой, так как характерные для амилоидов бета-листы образуются несколькими цепями, что хорошо видно на рисунке 1, где структура покрашена по цепям. Однако, мономер может обладать какой-то своей вторичной структурой.
Лучшее предсказание структуры мономера с помощью AlphaFold2 (ссылка на архив), выровненное на одну цепь референсной структуры, представлено на рисунке 2. Для предсказания использовалось 3000 других структур.
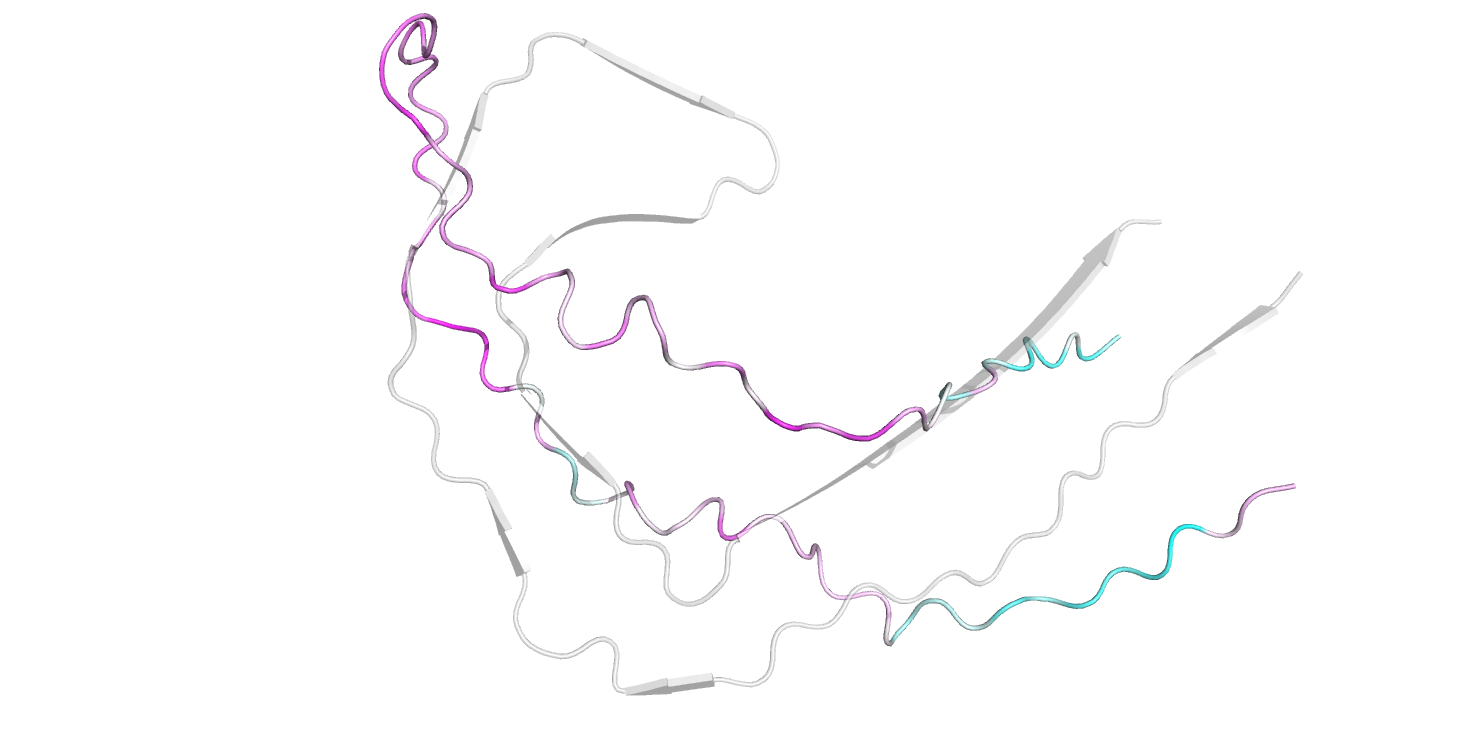
Рисунок 2.
Предсказанная структура действительно не обладает никакой вторичной структурой, но у нее есть некоторое сходство с референсной структурой. Поскольку, без связей с другими цепями белок хуже стабилизирован в случае амилоидов, существующих в виде агрегатов, то предсказание имеет место быть. Тем не менее, не стоит считать, что структура верная. Предсказанная структура окрашена по plddt, т.е. в фиолетовых участках программа наиболее "уверена". Структура единичного амилоида очень нестабильна, возможно вообще нет какой-то одной определенной структуры, в которой пребывает единичная цепь амилоида. Дальнейшие выводы о качестве предсказанной структуре можно сделать, посмотрев на предсказания для 5 цепей с данной последовательностью и для 10 цепей.
На рисунке 3 представлено лучшее предсказание структуры 5 цепей с помощью AlphaFold2 (ссылка на архив), выровненное на одну референсную структуру.
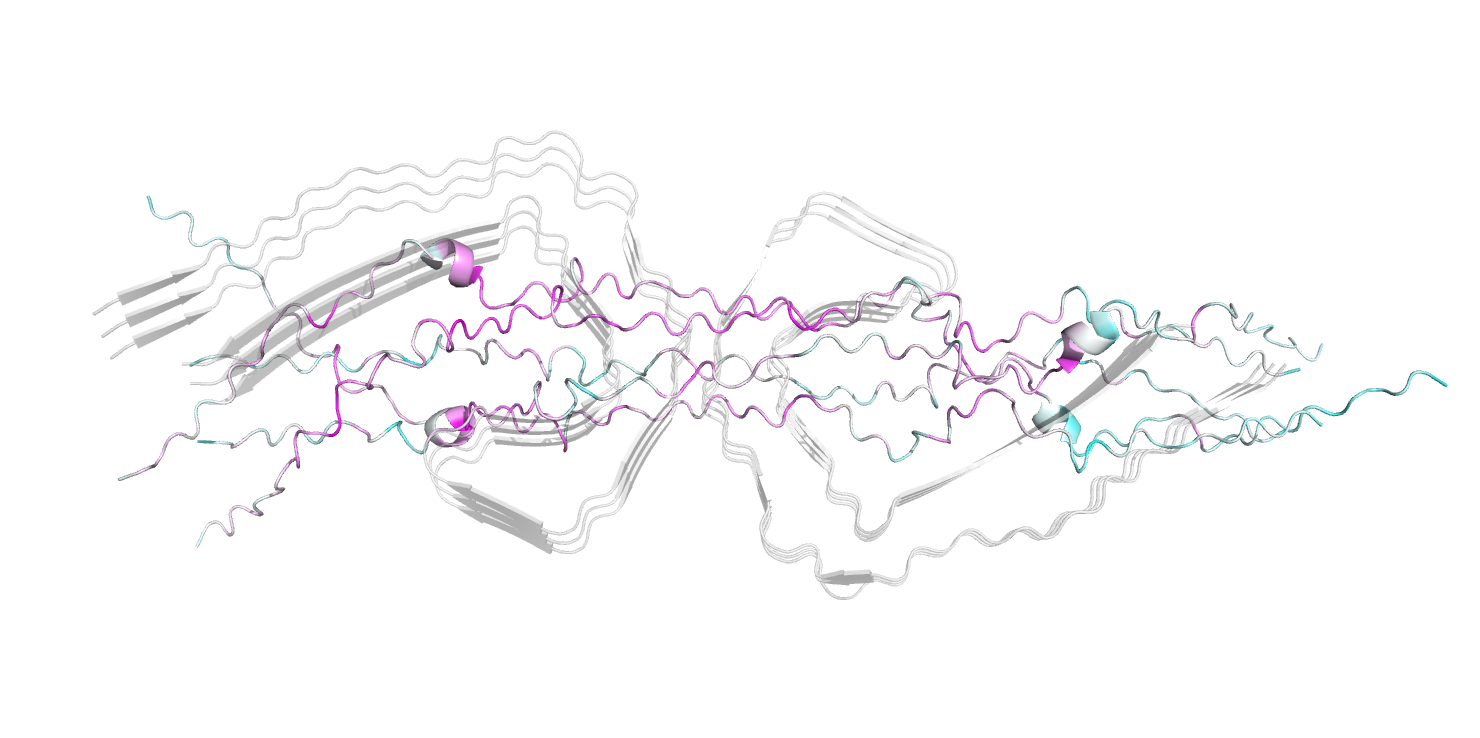
Рисунок 3.
Здесь структура вообще не похожа на референсную. Кроме того, AlphaFold2 предсказал спирали, которые не характерны для амилоидов. Извилисты остов напоминает остов бета-листа, однако ни одного бета-листа нет. Предсказанная структура кроме всего не выглядит очень устойчивой и стабильной. Это тоже не характерно для амилоидных агрегатов. Три спирали хотя бы частично окрашены фиолетовым, что говорит о высокой уверенности программы в их верности. Тем не менее, это совсем не согласуется с реальностью.
Осталось рассмотреть предсказание для 10 цепей (ссылка на архив). Оно представлено на рисунке 4.
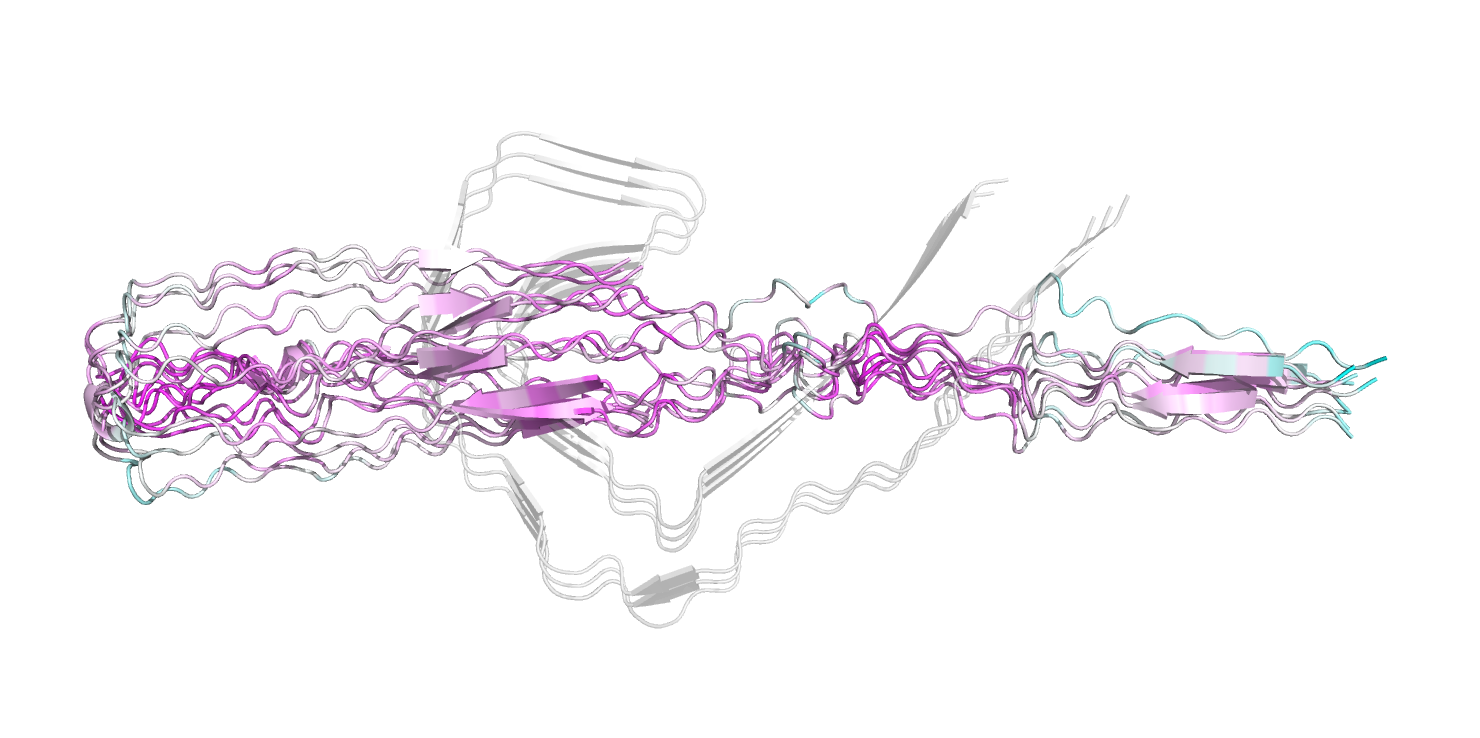
Рисунок 4.
Предсказанная структура для 10 цепей тоже не похожа не референс. Однако, тут хотя бы есть бета-листы, что приближает структуру к реальной, но не сильно. Вторичные структуры похожи, но не наблюдается упорядоченности цепей и изгиба цепей. Кроме того, в левой части молекулы наблюдается парочка спиралей.
В целом, можно сделать вывод, что AlphaFold2 здесь предсказал структуры абсолютно не похожие на правду. Возможно, в целом сложно предсказывать структуры амилоидов. Возможно, в будущем, предсказания станут более правдоподобными. Однако, сейчас предсказание амилоидных структур оставляет желать лучшего.
Ссылки:
- https://en.wikipedia.org/wiki/Amyloid
- https://www.frontiersin.org/articles/10.3389/fnagi.2019.00204/full