ЯМР vs РСА
В течение данного практикума я работала с мембранным белком дисферлином C2A. Этот белок связан с восстановлением мышц скелета. Его нехватка вызывает различные типы мышечной дистрофии.
Задание 1. Введение.
В рамках этого задания я сравнивала две структуры одного и того же мембранного белка, полученные разными методами. Одна структура была получена с помощью ядерно магнитного резонанса (PDB ID: 7K6B), а другая - с помощью рентгеноструктурного анализа (PDB ID: 4IQH). Структура, полученная с помощью ЯМР представлена 20 моделями. Резрешение для РСА структуры 1.76 Å (полнота 87.2%).
Обе структуры были отображены с помощью программы pymol и наложены друг на друга.
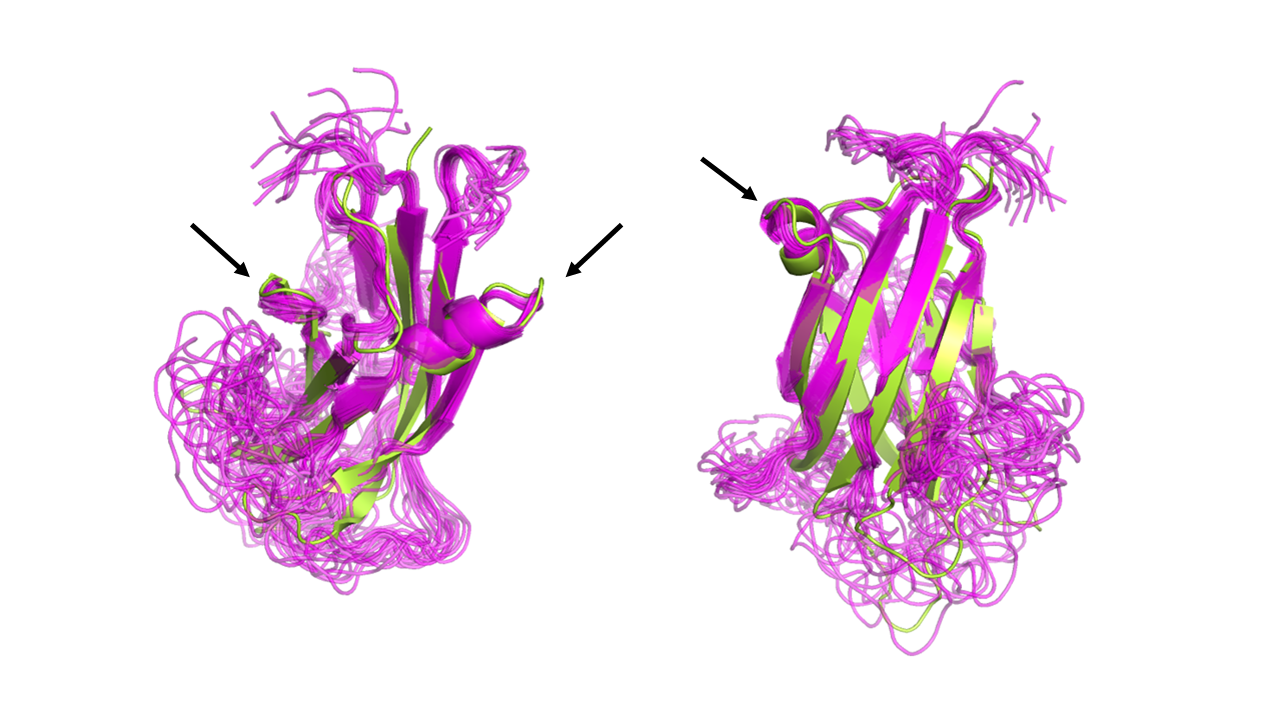
Рисунок 1.
На рисунке 1 изображены структура, полученная РСА (желтый), и набор моделей, полученных ЯМР (фиолетовый). Из рисунка видно насколько похожи обе структуры. Основные элементы белка, такие как бета-листы и альфа-спираль наложились друг на друга практически идеально. Это было ожидаемо, так так это наиболее стабильные структуры и при использовании данных двух методов должны хорошо идентифицироваться. Что касается свободных петель, то тут уже наблюдаются более значительные различия, но на рисунке видно, что некоторые петли обеих структур практически идеально ложатся друг на друга (показаны стрелочками).
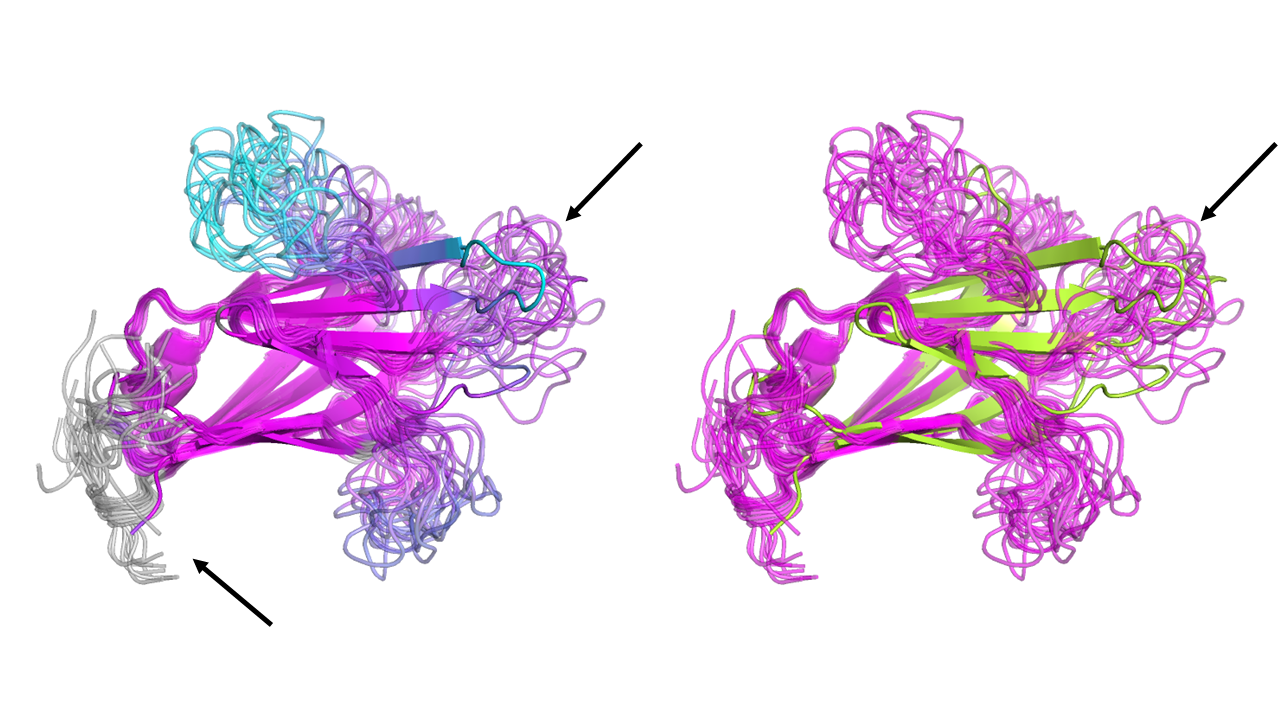
Рисунок 2.
На рисунке 2 я акцентировала внимание на различиях структур. Слева структуры окрашены в соответсвии с RMSD (root mean square deviation) расстояний между одинаковыми атомами моделей. Фиолетовый цвет характеризует маленькое расстояние, а голубой - большое. Серые же части - фрагменты, для которых RMSD не было посчитано, т.е. данные части присутствуют только в одной из структур. Модели были покрашены с помощью скрипта. Справа изображены те же структуры, в том же ракурсе, но покрашены аналогично первому рисунку, чтобы было понятнее, что к какой структуре относится.
Слева преобладает фиолетовый цвет (в частности альфа-петля полностью фиолетовая, преимущественная часть бета-листов тоже), что характеризует схожесть структур. Однако, есть и голубые фрагменты. Черными стрелочками слева и справа я показала петлю, которая очень сильно отличается в этих структурах. В РСА структуре бета-лист больше, чем в ЯМР структуре. Поэтому данная петля оказалась в разных положениях на данных структурах. Ещё в ЯМР структуре присутствуют серые фрагменты (тоже показано черной стрелочкой), что свидетельствует о том, что даже аминокислотная последовательность для этих структур немного отличается (далее об этом будет говориться более подробно).
Данные различия объясняются разными условиями для проведения РСА и ЯМР. Для РСА белок кристаллизуют, и могут возникнуть некоторые связи, которых нет в нативном состоянии. Кроме того, данный белок в норме встроен в мембрану, и при кристаллизации теряются его взаимодействия с липидным окружением, что тоже может изменить положения некоторых его участков. В эксперименте ЯМР белок находится в растворе, что близко к нативному состоянию, однако при анализе ЯМР данных можно не найти некоторые структруры или их части из-за специфики полученных данных.
Задание 2. RMSF.
В этом задании я сравнивала B-фактор для РСА структуры, который характеризует различия в положениях атомов в разных молекулах белка в кристалле, и RMSF, посчитанный по всем моделям ЯМР. RMSF (root mean square fluctuation) похоже на RMSD, которое я вычисляла в предыдущем задании, однако данная велечина усредняется по времени, в отличие от RMSD. RMSF для моделей ЯМР тоже характеризует различия положений атомов в разных моделях, поэтому эту величину можно считать аналогом В-фактора в РСА (если модели ЯМР действительно характеризуют пожвижность). В данном задании вычислялись В-факторы и RMSF средние по остаткам.
Для того, чтобы сравнивать RMSF и В-факторы для одних и тех же остатков структур, я сделала выравнивание моделей в pymol и открыла его с помощью программы UGENE.

Рисунок 3.
На рисунке 3 представлено выравнивание аминокислотных остатков для двух структур. Верхняя строчка для структуры РСА, а нижняя - для ЯМР. Из выравнивания хорошо видно, что аминокислотные последовательности структур совпадают не полностью. Для сравнения я взяла только аминоксислтные остатки в красной рамочке.
С помощью ProDy я рассчитала средние RMSF и В-факторы для необходимых остатков и отобразила их зависимость на графике, который представлен ниже.
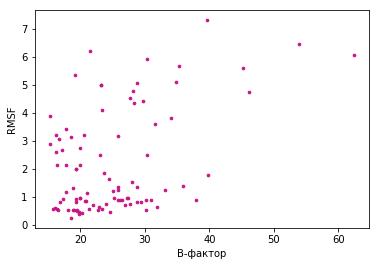
Рисунок 4.
На графике зависимости RMSF от В-фактора (рис.4) видно, что большинство точек находятся в нижнем левом углу. Это логично, так как это свидетельствует о том, что разница между положениями большинства остатков в молекулах кристалла и моделях ЯМР не очень велика. Однако, я особо не наблюдаю корреляции данных величин, что подтверждает мысль, что различия в моделях ЯМР в большинстве случаев никак не характеризуют подвижность, а просто вызваны шумом в данных и неидеальностью эксперимента.
Задание 3.
В данном задании нужно было найти в структуре РСА три водородные связи: водородную связь между атомами остова в ядре белка (в альфа-спирали или бета-листе), водородную связь боковых цепей в ядре белка и водородную связь в петлях, выходящих на поверхность глобулы. Затем наличие или отсутствие этих водородных связей рассматривалось в рамках каждой модели ЯМР.
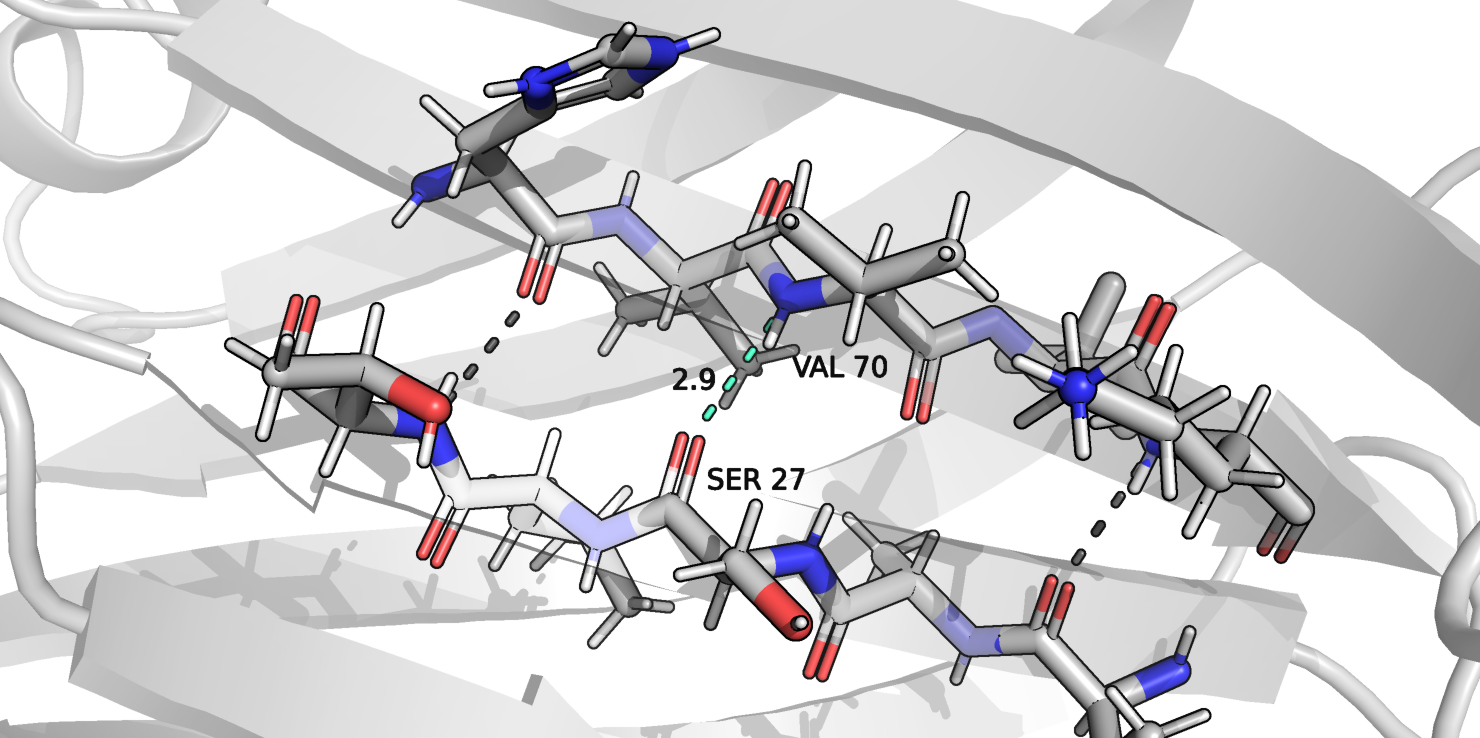
Рисунок 5.
На рисунке 5 показана первая водородная связь (голубой цвет) в бета-листе белка. Эта водородная связь образована атомами частей остова, которые принадлежат остаткам VAL70 и SER27.
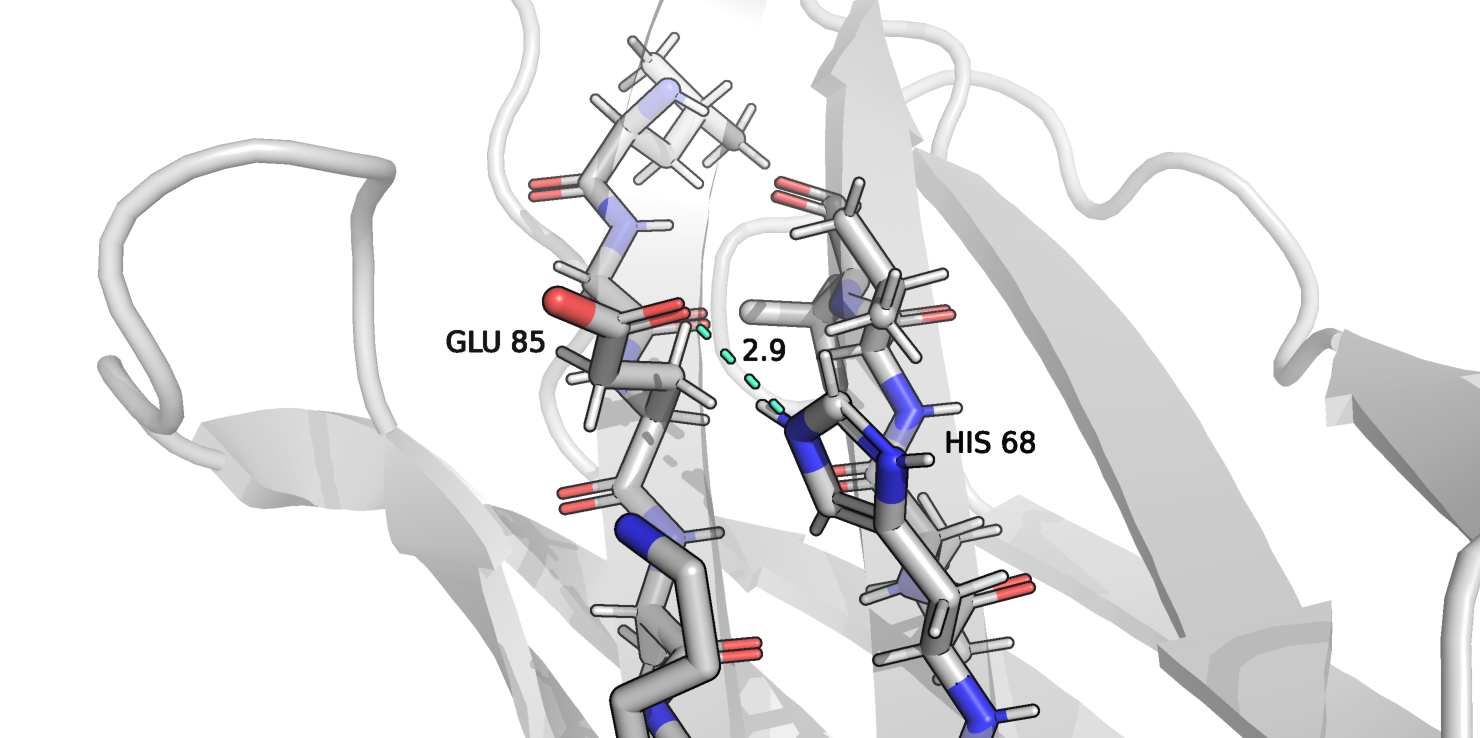
Рисунок 6.
На рисунке 6 я отобразила вторую водородную связь. Она образована атомом кислорода в остатке GLU85 и атомом азота в остатке HIS68. Эти остатки находятся снаружи молекулы. Я хотела найти водородные связи в пространстве между бета-листами, но все остатки в данном пространстве оказались неполярными, что в целом логично.
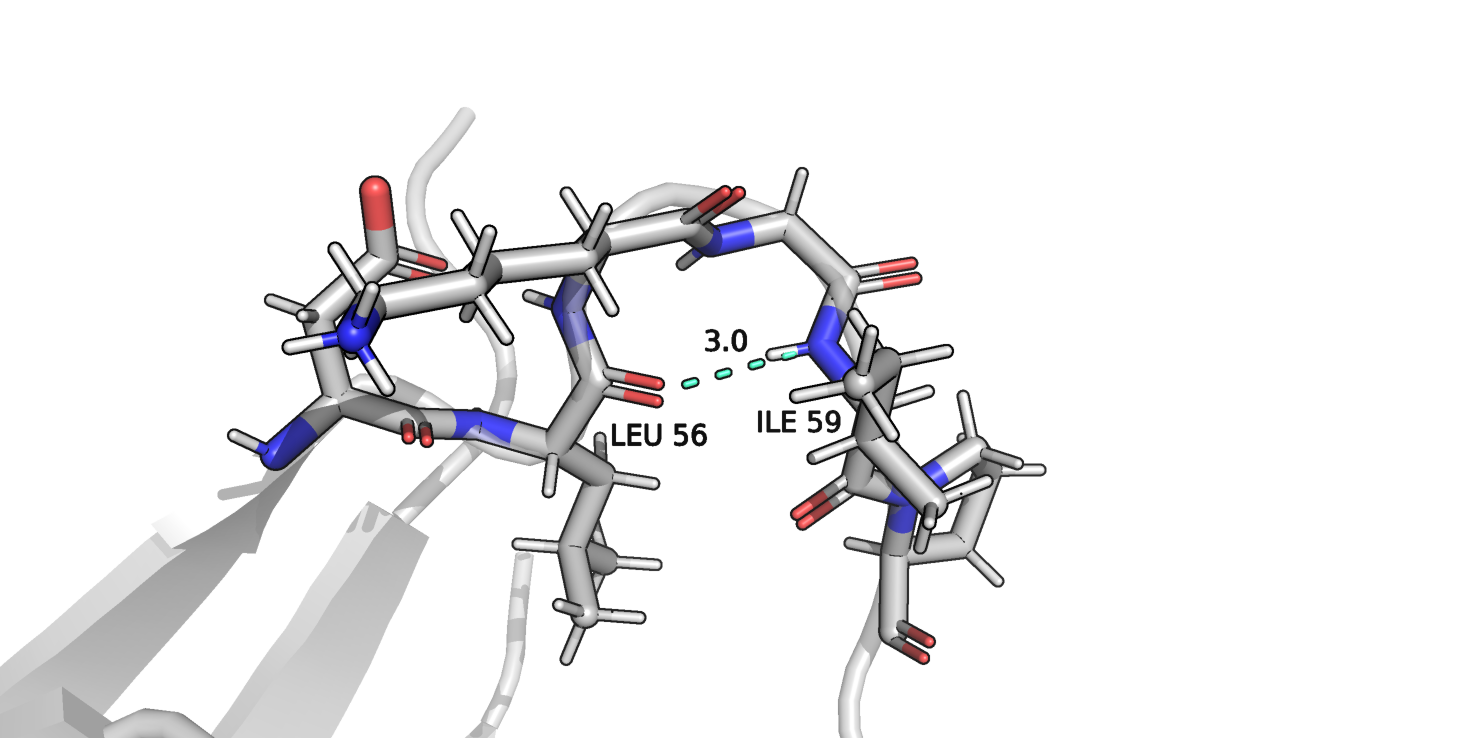
Рисунок 7.
На рисунке 7 показана третья водородная связь. Эта связь находится в петле, торчащей из глобулы. Она аналогично первой образована атомами остова, только теперь атомы принадлежат остаткам LEU56 и ILE59.
В таблице ниже представлена информация по наличию/отсутствию и характеристикам данных водородных связей в моделях ЯМР. Как уже было сказано ранее, аминокислотные остатки структур РСА и ЯМР немного отличались, поэтому для ЯМР структуры номера всех остатков меньше на 2 относительно структуры РСА. Я считала, что водородная связь присутствует в модели, если расстояние между атомами было не больше 3.6 Å.
| № | Остатки, образующие связь | Расстояние в РСА (Å) | Модели ЯМР с водородной связью | Минимальное расстояние в ЯМР (Å) | Максимальное расстояние в ЯМР (Å) | Медианное расстояние в ЯМР (Å) |
|---|---|---|---|---|---|---|
| 1 | VAL70(68), SER27(25) | 2.9 | 13 (65%) | 2.7 | 4.4 | 3.35 |
| 2 | HIS68(66), GLU85(83) | 2.9 | 2 (10%) | 2.7 | 12.8 | 7.6 |
| 3 | ILE59(57), LEU56(54) | 3.0 | 15 (75%) | 3.2 | 3.8 | 3.6 |
По результатам в таблице видно, что водородные связи между атомами остова наблюдаются больше, чем в половине моделей ЯМР. Что касается водородных связей между атомами боковых цепей, торчащих наружу, то в большинстве ЯМР моделей расстояние между атомами слишком большое. Когда я переключалась между моделями в pymol, было видно насколько сильно различаются положения данных остатков в разных моделях.
Для того, чтобы лучше понять, почему я получила такие результаты, я решила посмотреть на места водородных связей во всей молекуле целиком.
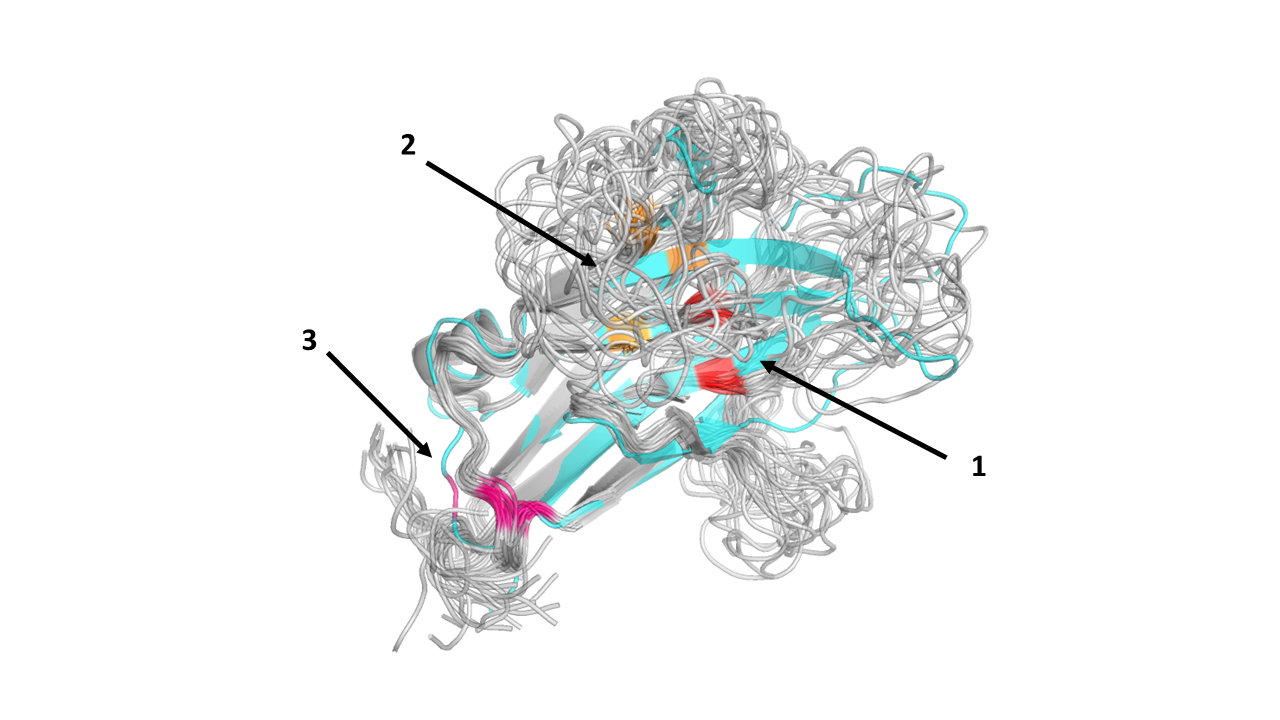
Рисунок 8.
На рисунке выше изображены выровненные структуры РСА (голубой) и ЯМР (серый), на них покрашены в разные цвета остатки, между которыми я рассматривала водородные связи. Номера на рисунке соответствуют номерам в таблице 1. Из рисунка 8 становится понятнее, почему вторая связь так редко встречается в моделях ЯМР. Для структуры, полученной ЯМР один из остатков, образующих эту связь, уже не входит в состав бета-листа и находится на достаточно большом расстоянии от другого.
Тем не менее, связи 1 и 3 содержатся в большом количестве моделей. Петля со связью 3 в структуре ЯМР немного повернута относительно структуры РСА, но, видимо, водородная связь в этой петле отвечает за ее формирование, поэтому встречается в 75% моделей.