Структура и функция
Данный практикум посвящен влиянию мутаций на структуру и функцию белка.
Я рассматривала субъединицу 1 рецептора гамма-аминомасляной кислоты типа B из организма домашняя лошадь (Uniprot ID: A0A5F5PPR1).
Субъединица 1 рецептора гамма-аминомасляной кислоты типа B - это субъединица трансмембранного рецептора GPCR, лигандом которой является, очевидно, гамма-аминомасляная кислота (рис. 1).
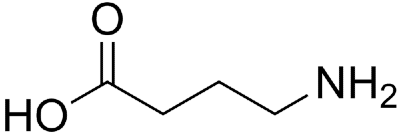
Рисунок 1.
На Uniprot не было структур для данного белка, поэтому было необходимо найти похожий белок в другом организме, для которого есть структура. Для этого я использовала белковый BLAST с поисом по базе данных PDB.
В результате была найдена 21 последовательность. При этом для 7 последовательностей идентичность была больше 95%. Все 7 последовательностей пренадлежат организму Homo sapiens. Кроме того для двух последовательностей было написано, что это структура рецептора в активном состоянии. Тем не менее я решила поподробнее рассмотреть все 7 с помощью pymol. Во всех структурах были различные лиганды, многие из них находились за пределами белка. Практически во всех структурах встречались остатки хитина (лиганд 2-acetamido-2-deoxy-beta-D-glucopyranose). Кроме того в некоторых структурах присутствовал позитивный аллостерический модулятор GABAB рецептора - N~4~,N~6~-dicyclopentyl-2-(methylsulfanyl)-5-nitropyrimidine-4,6-diamine. Однако, в одной из двух структур активного состояния рецептора, структуре 6UO8 я обратила внимание на лиганд, очень похожий на гамма-аминомасляную кислоту. Это была (R)-(3-аминопропил)метилфосфиновая кислота (рис. 2). В литературе этот лиганд ещё называют SKF-97,541 и описывают как соединение, используемое в научных исследованиях и действующее в первую очередь как селективный агонист рецептора GABAB.
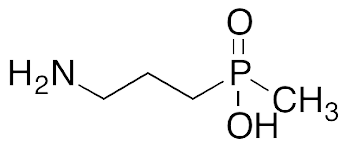
Рисунок 2.
Во второй структуре активного состояния 7C7Q тоже был интересный лиганд, поскольку он имел тривиальное название: баклофен (рис. 3). Оказалось, что баклофен - это лекаство, стимулятор GABAB. При рассмотрении структуры в pymol оба данных лиганда оказались приблизительно в одном месте, очевидно, это центр связывания лиганда данного рецептора.
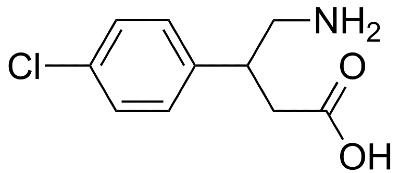
Рисунок 3.
Что интересно, в некоторых других структурах на этом месте нашлись другие лиганды. В структуре неактивного состояния 6W2X на этом месте оказался лиганд [(2~{S})-3-[[(1~{S})-1-(3,4-dichlorophenyl)ethyl]amino]-2-oxidanyl-propyl]-(phenylmethyl)phosphinic acid (рис. 4). Найти информацию про применение данного лиганда мне найти не удалось.
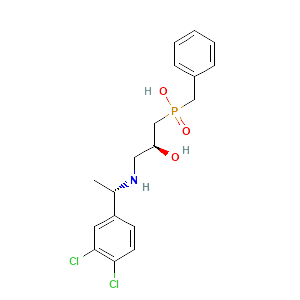
Рисунок 4.
В ещё одной структуре 7C7S оказался тот же самый лиганд, в том же положении. Только вот в описании структуры он называется (R)-(cyclohexylmethyl)[(2S)-3-{[(1S)-1-(3,4-dichlorophenyl)ethyl]amino}-2-hydroxypropyl]phosphinic acid (рис. 4). В результате поиска по этому названию, стало понятно, что это селективный антагонист GABAB рецептора.
В оставшихся 3 структурах в активном центре ничего найдено не было.
В надежде найти структуру с гамма-аминомасляной кислотой в качестве лиганда, я произвела поиск похожих структур с помощью PDBeFold. В качестве исходной структуры я выбрала 6UO8, т.к. её лиганд больше всего похож на необходимый. В результате было найдено 27 структур, но в топе были те же, что выдал белковый BLAST. Таким образом ничего особо новоги и полезного в данном случае я не нашла.
В конечном итоге, для дальней шей работы я остановилась на структуре 6UO8.
Чтобы окончательно удостовериться, что структура подходит, я посмотрела на выравнивание:
Query 1 MSGGWPGGQACQPAVEMALEDVNSRRDILPDYELKLIHHDSKCDPGQATKYLYELLYNDP 60
MSGGWPGGQACQPAVEMALEDVNSRRDILPDYELKLIHHDSKCDPGQATKYLYELLYNDP
Sbjct 15 MSGGWPGGQACQPAVEMALEDVNSRRDILPDYELKLIHHDSKCDPGQATKYLYELLYNDP 74
Query 61 IKIILMPGCSSVSTLVAEAARMWNLIVLSYGSSSPALSNRQRFPTFFRTHPSATLHNPTR 120
IKIILMPGCSSVSTLVAEAARMWNLIVLSYGSSSPALSNRQRFPTFFRTHPSATLHNPTR
Sbjct 75 IKIILMPGCSSVSTLVAEAARMWNLIVLSYGSSSPALSNRQRFPTFFRTHPSATLHNPTR 134
Query 121 VKLFEKWGWKKIATIQQTTEVFTSTLDDLEERVKEAGIEITFRQSFFSDPAVPVKNLKRQ 180
VKLFEKWGWKKIATIQQTTEVFTSTLDDLEERVKEAGIEITFRQSFFSDPAVPVKNLKRQ
Sbjct 135 VKLFEKWGWKKIATIQQTTEVFTSTLDDLEERVKEAGIEITFRQSFFSDPAVPVKNLKRQ 194
Query 181 DARIIVGLFYETEARKVFCEVYKERLFGKKYVWFLIGWYADNWFKIYDPSINCTVDEMTE 240
DARIIVGLFYETEARKVFCEVYKERLFGKKYVWFLIGWYADNWFKIYDPSINCTVDEMTE
Sbjct 195 DARIIVGLFYETEARKVFCEVYKERLFGKKYVWFLIGWYADNWFKIYDPSINCTVDEMTE 254
Query 241 AVEGHITTEIVMLNPANTRSISNMTSQEFVEKLTKRLKRHPEETGGFQEAPLAYDAIWAL 300
AVEGHITTEIVMLNPANTRSISNMTSQEFVEKLTKRLKRHPEETGGFQEAPLAYDAIWAL
Sbjct 255 AVEGHITTEIVMLNPANTRSISNMTSQEFVEKLTKRLKRHPEETGGFQEAPLAYDAIWAL 314
Query 301 ALALNKTSGGGGRSGVRLEDFNYNNQTITDQIYRAMNSSSFEGVSGHVVFDASGSRMAWT 360
ALALNKTSGGGGRSGVRLEDFNYNNQTITDQIYRAMNSSSFEGVSGHVVFDASGSRMAWT
Sbjct 315 ALALNKTSGGGGRSGVRLEDFNYNNQTITDQIYRAMNSSSFEGVSGHVVFDASGSRMAWT 374
Query 361 LIEQLQDLLCNRDKRSGLQPWSKGCGSYKKIGYYDSTKDDLSWSKTDKWIGGSPPADQTL 420
LIEQLQ GSYKKIGYYDSTKDDLSWSKTDKWIGGSPPADQTL
Sbjct 375 LIEQLQG------------------GSYKKIGYYDSTKDDLSWSKTDKWIGGSPPADQTL 416
Query 421 VIKTFRFLSQKLFISVSVLSSLGIVLAVVCLSFNIYNSHVRYIQNSQPNLNNLTAVGCSL 480
VIKTFRFLSQKLFISVSVLSSLGIVLAVVCLSFNIYNSHVRYIQNSQPNLNNLTAVGCSL
Sbjct 417 VIKTFRFLSQKLFISVSVLSSLGIVLAVVCLSFNIYNSHVRYIQNSQPNLNNLTAVGCSL 476
Query 481 ALAAVFPLGLDGYHIGRNQFPFVCQARLWLLGLGFSLGYGSMFTKIWWVHTVFTKKEEKK 540
ALAAVFPLGLDGYHIGRNQFPFVCQARLWLLGLGFSLGYGSMFTKIWWVHTVFTKKEEKK
Sbjct 477 ALAAVFPLGLDGYHIGRNQFPFVCQARLWLLGLGFSLGYGSMFTKIWWVHTVFTKKEEKK 536
Query 541 EWRKTLEPWKLYATVGLLVGMDVLTLAIWQIVDPLHRTIETFAKEEPKEDIDVSILPQLE 600
EWRKTLEPWKLYATVGLLVGMDVLTLAIWQIVDPLHRTIETFAKEEPKEDIDVSILPQLE
Sbjct 537 EWRKTLEPWKLYATVGLLVGMDVLTLAIWQIVDPLHRTIETFAKEEPKEDIDVSILPQLE 596
Query 601 HCSSRKMNTWLGIFYGYKGLLLLLGIFLAYETKSVSTEKINDHRAVGMAIYNVAVLCLIT 660
HCSSRKMNTWLGIFYGYKGLLLLLGIFLAYETKSVSTEKINDHRAVGMAIYNVAVLCLIT
Sbjct 597 HCSSRKMNTWLGIFYGYKGLLLLLGIFLAYETKSVSTEKINDHRAVGMAIYNVAVLCLIT 656
Query 661 APVTMILSSQQDAAFAFASLAIVFSSYITLVVLFVPKMRRLITRGEWQSEAQDTMKTGSS 720
APVTMILSSQQDAAFAFASLAIVFSSYITLVVLFVPKMRRLITRGEWQSEAQDTMKTGSS
Sbjct 657 APVTMILSSQQDAAFAFASLAIVFSSYITLVVLFVPKMRRLITRGEWQSEAQDTMKTGSS 716
Query 721 TNNNEEEKSRLLEKENRELEKIIAEKEERVSELRHQLQSRQQL 763
TNNNEEEKSRLLEKENRELEKIIAEKEERVSELRHQLQSR ++
Sbjct 717 TNNNEEEKSRLLEKENRELEKIIAEKEERVSELRHQLQSRLEV 759
Выравнивание хорошее (идентичность 97%), однако, важно отметить, что в выбранной структуре сначала присутствует сдвиг -14. Кроме того есть участок гэпов, который тоже сдвигает нумерацию. Любопытно, что данный участок гэпов присутствует во всех выравниваниях с индентичностью более 95%. Исходный белок принадлежит лошади, а найденные последовательности из организса человека, это объясняет различия и одинаковый участок гэпов во всех выравниваниях.
В итоге далее я буду использовать А цепь структуры 6UO8. Разрешение данной структуры 3.63 Å. Показатели хорошие: clashscore 4, маргиналов по рамачандрану нет, а маргиналов по ротамерам боковых цепей 0.1%.
Важно отметить, что в данной структуре присутствует позитивный аллостерический модулятор, упомянутый выше. Однако при выравнивании на другие структуры, не содержащие модулятор, я не заметила особой разницы, так что его присутствие не должно помешать дальнейшему исследованию.
Мутации.
В выбранную структуру я вносила мутации, заменяя один аминокислотный остаток на другой, и проверяла, насколько определенная мутация влияет на белок: улучшает, не влияет или портит белок. То есть насколько индуцированная мутация влияет на выполнение белком его функции.
Первой мутацией была замена глутаминовой кислоты 289 (в выбранной структуре 466) на глутамин. Исходная структура изображена на рисунке 5.
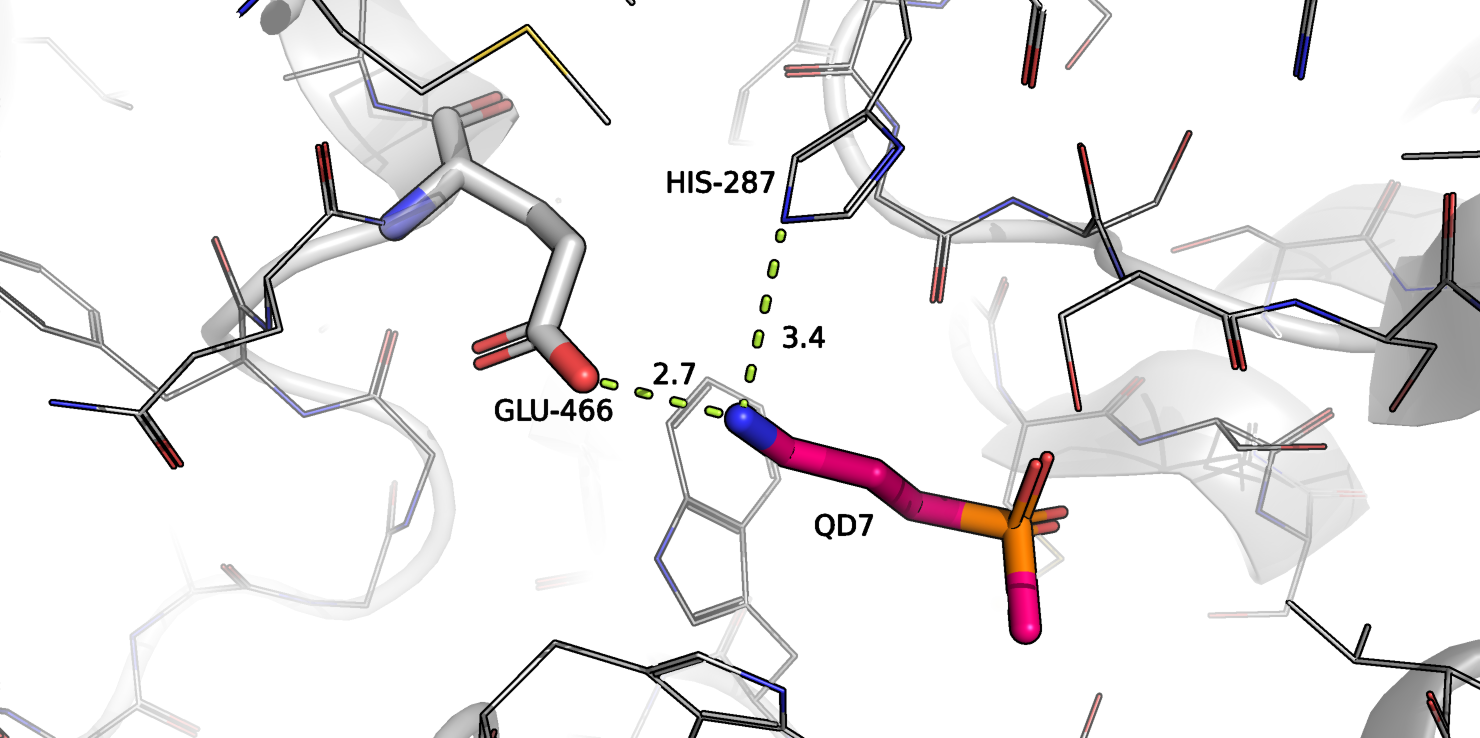
Рисунок 5.
Из рисунка 5 видно, что глутаминовая кислота находится в центре связывания лиганда белком и образует водородную связь с азотом лиганда. Однако, данный атом лиганда еше образует связь с имидазольным кольцом гистидина 287. Размер у глутамина и глатамата примерно одинаковый, тем не менее, замена кислорода на NH2-группу нарушит существующую систему водородных связей. Замениться может один из двух кислородов. Если заменить левый, нарушится связь с остовом, поэтому его заменить нельзя. Правый кислород образует связь с лигандом, при этом в норме у ГАМК азот протонирован 3 протонами (содержит NH3+-группу), поэтому замена кислорода на NH2-группу сделает невозможным образование водродной связи с лигандом. Таким образом, такая замена негативно скажется на связывании белком лиганда, что сильно снизит эффективность белка в выполнении его функции (эффективно сможет связываться только минорное состояние лиганда с группой NH2, а лиганда в таком состоянии очень мало). Кроме того, замена аспартата на аспарагин сделает невозможным образование солевого мостика, образуемого аспартатом. В итоге, эта на первый взгляд незаметная мутация оказалась сильно отрицательной, потому что сильно ухудшает связывание лиганда, что приводит с значительному ухудшению выполнения функции белком.
Вторая мутация это замена аспарагиновой кислоты 400 (559) на аспарагин.
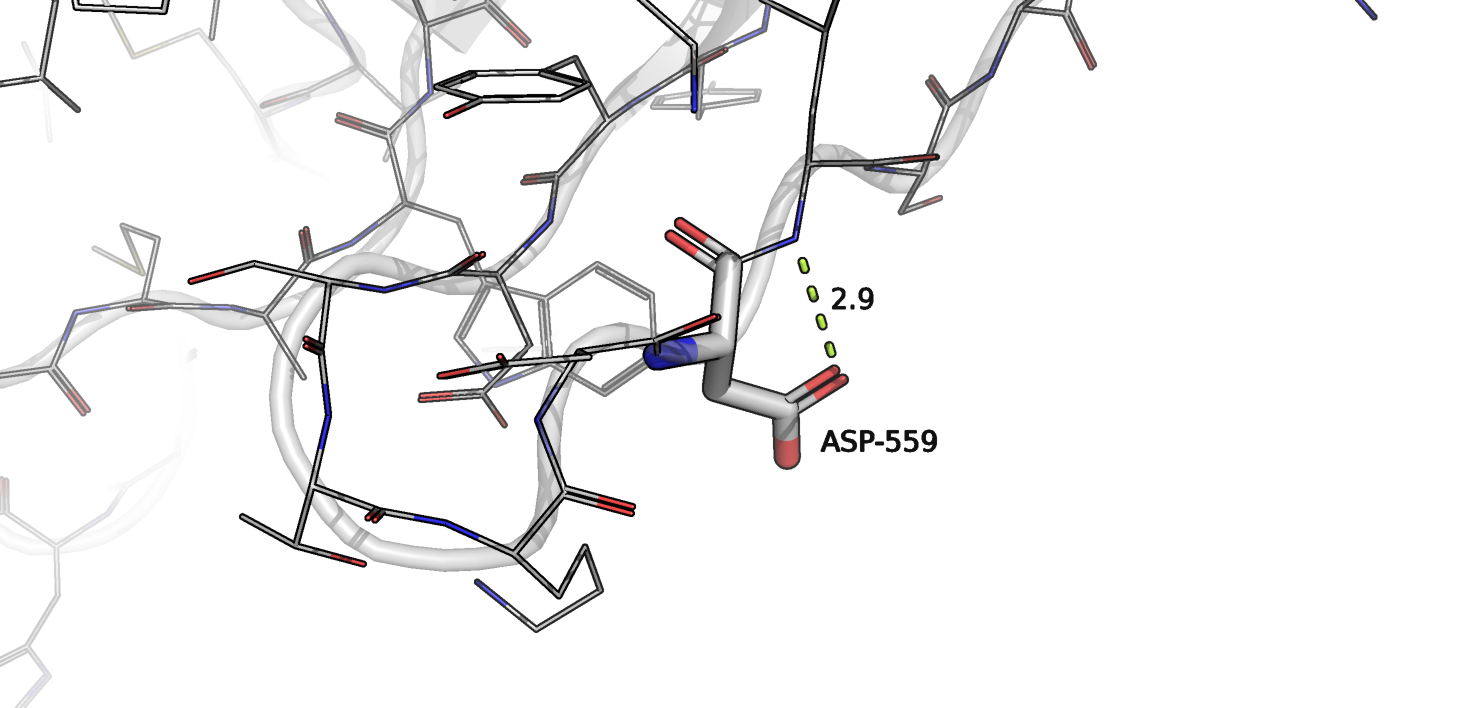
Рисунок 6.
Как видно из рисунка 6, где изображена аспарагиновая кислота, этот остаток находится на краю молекулы белка. Данная аминокислота образует водородную связь с остовом. Поскольку аспарагин тоже сможет образовать данную водородную связь и является полярным, то замену можно считать нейтральной.
Последней была замена цистеина 43 (220) на серин. Расположение данного остатка в белке можно на рисунке 7.
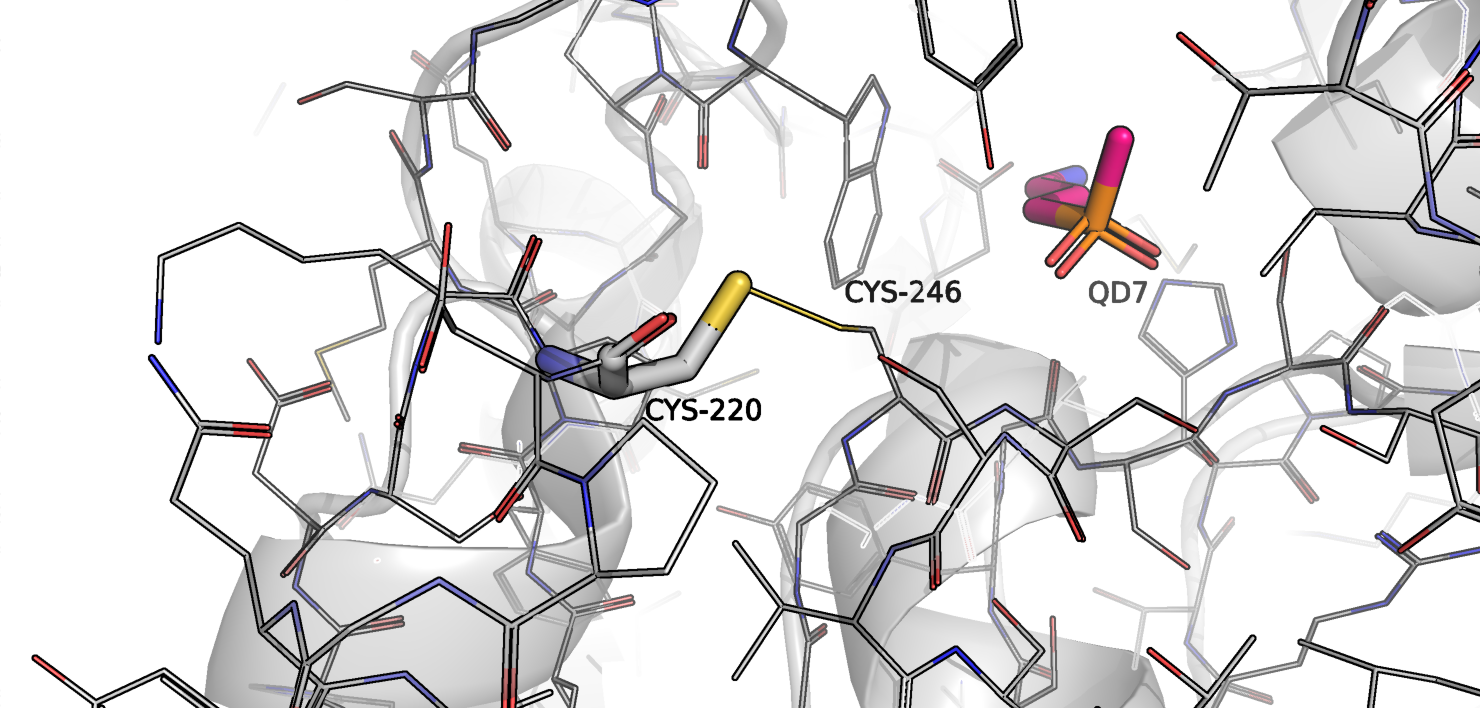
Рисунок 7.
Из рисунка 7 видно, что цистеин образует S-S связь с цистеином 246. Более того, данный цистеин находится около центра связывания лиганда. Из этого уже можно сдлеать вывод, что замена этого остатка на любой другой скорее всего будет вредной. Однако, я решила воспользоваться возможностями pymol и посмотреть на эту мутацию в структуре. Pymol предложил 3 ротамера. При этом минимальный strain был 29.95. Это уже тоже свидетельствует о том, что замена негативная. Мутированная и отрелаксированная структура избражена на рисунке 8.

Рисунок 8.
В итоге, серин, заменивший цистеин образовал водородную связь с тем же цистеином 246. В целом геометрия особо не попортилась. Тем не менее, водородная связь, в сравнении с S-S связью, менее хороша для стабилизации структуры, но хорошо, что есть хотя бы она. Таким образом, данную мутацию можно назвать отрицательной, но не критичной.