Домен PF00166
Задание 1
| Таблица 12-1 | Информация | Комментарии | ||||||
|---|---|---|---|---|---|---|---|---|
| AC pfam | PF00166 | |||||||
| ID pfam | Chaperonin 10 Kd subunit | Короткое название Cpn10, используется далее | ||||||
| #SEED | 37 | |||||||
| #All | 61 тыс. | |||||||
| #SW | 719 | |||||||
| #architectures | 154 | Доменная архитектура более 59 тыс. белков с изучаемым доменом состоит единственно из одного такого домена. Оставшиеся белки содержат другие домены или повторения Cpn10 (есть архитектура из трех). | ||||||
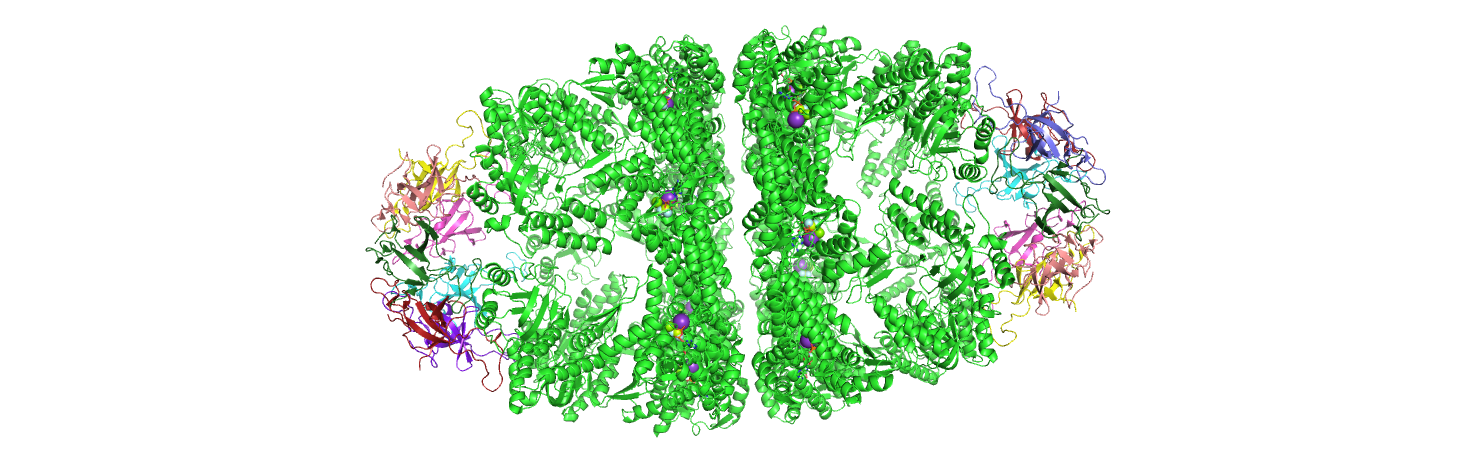
| #3D | 51 | Во вторичной структуре преобладают антипараллельные бета-листы. Семь субъединиц ко-шаперонина Hsp10/GroES (с доменом Cpn10) образуют кольцо. Оно "закрывает" гептамерное же кольцо шаперонина Hsp60/GroEL, которое связывает субстратный белок. Такой комлекс функционирует в виде димера, имеющего форму мяча для американского футбола. | ||||||
| Taxonomy | ||||||||
| #eukaryota | 8237 | |||||||
| #archaea | 359 | |||||||
| #bacteria | 49823 | |||||||
| #viruses | 1683 | |||||||
Задание 2
| Таблица 12-2 | Информация | Комментарии |
|---|---|---|
| Доменная архитектура 1 | PF00166 | 59146 белков |
| Белок с архитектурой 1 | P07889 | из Synechococcus sp., 103 а.к. |
| Доменная архитектура 2 | PF00166 - PF00166 | 1345 белков |
| Белок с архитектурой 2 | Q02073 | из хлоропласта Spinacia oleracea, 255 а.к. |
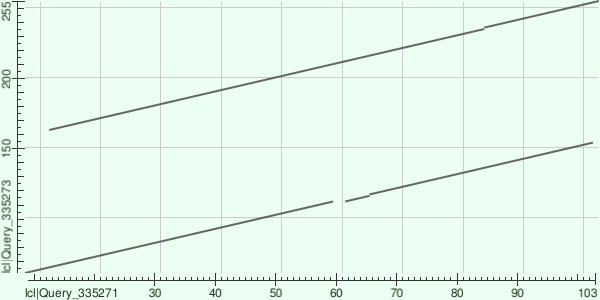
Согласно информации c соответствующих страниц InterPro, домен Cpn10 имеет координаты 4–103 в P07889 и 56–156, 157–255 в Q02073. BLAST выровнял домен первого белка с обоими доменами второго (E-value 3e-28 и 5e-26 соответственно). При этом эти два локальных выравнивания не включают первые несколько аминокислотных остатков каждого из доменов, что свидетельствует об их вариабельности. По карте видно, что в первом выравнивании (с N-концевым доменом второго белка) два небольших инделя (по одному в каждом белке) , а во втором (с C-концевым доменом второго белка) — один (в двухдоменном белке). Участков сходства вне доменов обнаружено не было (применялось пороговое значение E-value 0.05).
Задание 3
Я решила сравнить N-концевой и C-концевой домены в структуре PF00166 - PF00166. Я скачала 1345 последовательностей белков с такой структурой, загрузила в Jalview, выполнила remove redundancy (threshold 90), выровняла алгоритмом Mafft с параметрами по умолчанию и еще раз произвела remove redundancy (threshold 97). Затем я удалила из выравнивания все колонки за пределами доменов и разместила последовательности второго домена под последовательностями первого. Выровняла, создала группы N- и C-концевого доменов. Из выравнивания видно, что общий паттерн косервативных позиций для двух доменов совпадает. Однако, для N-концевого характерна функциональная консервативность позиций 6 (K/R), 20 (G, во втором домене чаще S), а для C-концевого — позиций 95 (D/E), 106 (Y). Также можно заметить, что N-концевой домен несколько длиннее. По расположению указанных консервативных остатков на N-конце N-концевого домена и C-конце C-концевого могу предположить, что они важны для олигомеризации белка.
Насколько я могу видеть, данная доменная архитектура встречается только у некоторых бактерий и фотосинтезирующих или имеющих гомологи пластид (например, Plasmodium sp.) эукариот. Было бы интересно выяснить, один или несколько раз появлялась такая доменная организация в эволюции. Думаю, для этого нужно выровнять домены из этой архитектуры с доменами белков с самой представленной архитектурой, состоящей из единственного домена. Тогда можно будет попробовать найти достоверные блоки выравнивания, распространяющиеся только на исследуемую архитектуру, и судить о происхождении ее от одной предковой последовательности. Но я столкнулась с техническими сложностями, связанными с тем, что однодоменная архитектура характерна для очень большого числа белков и на сайте InterPro невозможно сгенерировать fasta-файл с их последовательностями. Нужно каким-то образом отобрать репрезентативную выборку из этих белков, но я пока не очень понимаю как.