Обзор генома бактерии Xenorhabdus doucetiae
Введение
Xenorhabdus doucetiae – вид бактерий рода Xenorhabdus, относящегося к семейству Enterobacteriaceae, выделенный из нематоды Steinernema diaprepesi [1]. Она получила своё видовое название в честь учёного, впервые описавшего род нематод Steinernema (M. M. A. Doucet) [2].
Бактерии рода Xenorhabdus являются мутуалистическими симбионтами энтомопатогенных нематод Steinernema. Связь между видами бактерий и нематод высокоспецифична вследствие большого разнообразия наборов вторичных метаболитов разных видов Xenorhabdus. Так, в зависимости от вида-симбионта Xenorhabdus, его наличие может влиять как мутуалистически, так и патогенно на одну и ту же нематоду-хозяина [3].
Среди вторичных метаболитов Xenorhabdus doucetiae были найдены ксенорабдины и ксенокумарины – вещества, обладающие антибактериальным, противогрибковым, инсектицидным и противоопухолевым действием, что позволяет рассматривать их как перспективный объект для медицинской биотехнологии [4].
Материалы и методы
Для написания обзора использовались данные о бактерии из базы NCBI. В работе использовался сервис Google Sheets, такие его возможности, как построение таблиц, графиков, использование фильтров, функции СЧЁТЕСЛИ, СЧЁТЕСЛИМН, МАКС, МИН, БИНОМРАСП. Использовались также скрипты, написанные на языке программирования Питон.
Результаты
1. Основные данные о геноме
Геном бактерии содержит в себе одну хромосому и одну плазмиду. Хромосома содержит 4 195 202 пары оснований, плазмида – 8 449 пар оснований.
Процент GC-оснований в хромосоме составляет 45.7087%, в плазмиде – 45.0941%, то есть менее половины от всех оснований в геноме. На основании этих результатов можно считать, что геном бактерии не обладает повышенной устойчивостью к денатурации
2. Нуклеотидный состав генома
Было установлено, что помимо A, T, G, C, в последовательностях не встречается других букв.
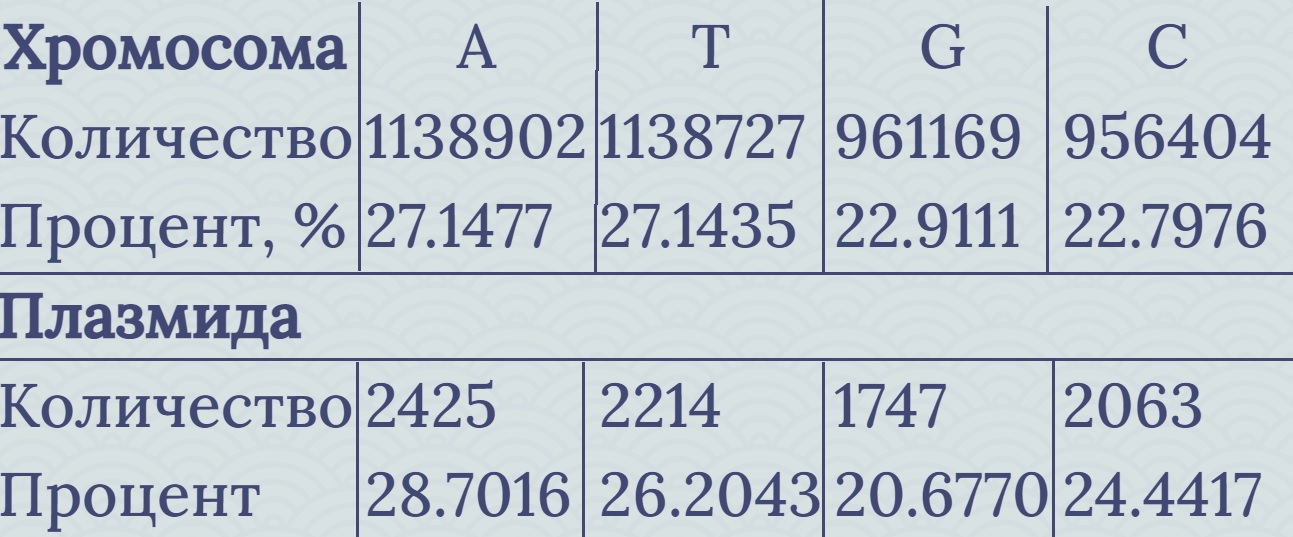
В результате исследования генома можно заметить, что второе правило Чаргаффа о равенстве числа A и T, G и C лучше выполняется для хромосомы. В плазмиде количества A и C преобладают на целые проценты.
3. Гистограмма длин белков
Была построена гистограмма распределения длин белков, которую можно увидеть на рис. 1
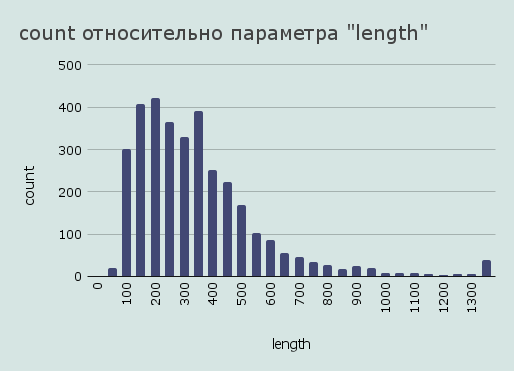
Большая часть белков имеет длину от 150 до 350 аминокислотных остатков. Самый короткий белок имеет длину 15, это лидерный пептид гистидинового оперона. Среди наиболее коротких белков находятся другие лидерные пептиды, гипотетические белки и другие.
Наибольшую длину имеет фермент синтетаза нерибосомных пептидов, его длина составляет 7 632 аминокислотных остатка. Интересно отметить, что 5 самых длинных белков аннотированы так же.
4. Распределение генов по цепям ДНК
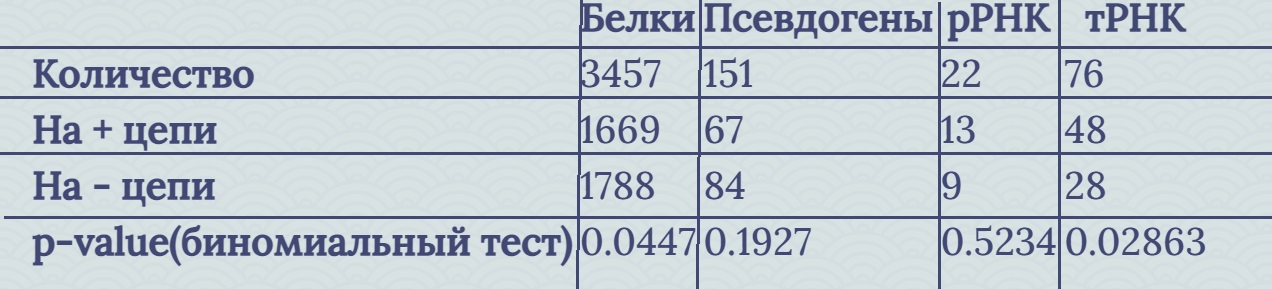
Распределение считается случайным, когда результаты теста более 0.05. Для псевдогенов и рРНК распределение можно считать случайным. Для белков и тРНК распределение нельзя считать случайным.
Обсуждение
По предположению авторов статьи [5], GC-состав задаёт модель использования кодонов и аминокислот в бактериальных геномах. Авторы перечисляют GC-состав множества видов бактерий из разных таксонов.
Наиболее близки к исследуемой бактерии Atopobium parvulum DSM 20469 из филума актинобактерий, Chloroherpeton thalassium ATCC 35110 из филума бактероиды, Lactobacillus brevis ATCC 367 Из филума фирмикуты, Hirschia baltica ATCC 49814 из филума альфапротеобактерии, Hirschia baltica ATCC 49814 из филума бетапротеобактерии, Shewanella denitrificans OS217 из филума гаммапротеобактерии.
В результате работы в геноме и протеоме бактерии были выявлены нестандартные распределения (нуклеотидов в плазмиде, генов на + и – цепях), природу которых было бы интересно изучить.
Сопроводительные материалы
Таблицы, код и файл с геномом, использованные для написания обзора, будут доступны по ссылке [6].
Список литературы
- Alexandrescu,A. (Entomopathogenic nematodes systematics, phylogeny and bacterial symbionts / Nguyen, Hunt, Под ред. Khuong B.Brill, 2007.
- Tailliez, P., Pages, S., Ginibre, N., & Boemare, N..New insight into diversity in the genus Xenorhabdus, including the description of ten novel species.// INTERNATIONAL JOURNAL OF SYSTEMATIC AND EVOLUTIONARY MICROBIOLOGY. 2006. 56(12), 2805–2818. doi:10.1099/ijs.0.64287-0
- Sicard, M., Ferdy, J.-B., Pagès, S., Le Brun, N., Godelle, B., Boemare, N., & Moulia, C.. When mutualists are pathogens: an experimental study of the symbioses between Steinernema (entomopathogenic nematodes) and Xenorhabdus (bacteria). //Journal of Evolutionary Biology. 2004. 17(5), 985–993. doi:10.1111/j.1420-9101.2004.00748.x
- Jönike Dreyer, Antoinette P Malan, Leon M T Dicks. Bacteria of the Genus Xenorhabdus, a Novel Source of Bioactive Compounds.// Front Microbiol. 2018. 19;9:3177. doi: 10.3389/fmicb.2018.03177
- Lightfield, J., Fram, N. R., & Ely, B. (2011). Across Bacterial Phyla, Distantly-Related Genomes with Similar Genomic GC Content Have Similar Patterns of Amino Acid Usage. PLoS ONE, 6(3), e17677.
- Сопроводительные материалы