Практикум 10: построение карт локального сходства
Для дальнейшей работы я решила выбрать две бактерии, относящиеся к одному роду и являющиеся паразитами человека. Первая из них — Chlamydophila pneumoniae, основной возбудитель пневмонии, вторая — Chlamydia trachomatis, возбудитель хламидиоза.
Чтобы найти их геномы, я использовала страницу геномы NCBI и перешла на страницу "browse by organism". Там установила фильтры на хромосомную и полную сборку, а также фильтр на референсные геномы.
Далее я получила номера сиквенсов их геномов и направила их в BLAST.
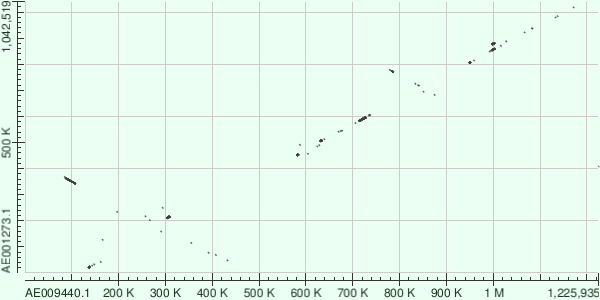
Рис.1 DotPlot при использовании megablast
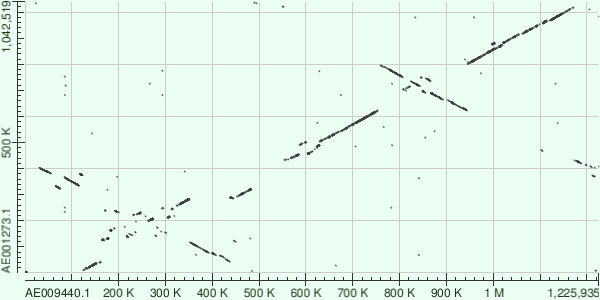
Рис.2 DotPlot при использовании blastn
Можно заметить, что график, полученный при помощи megablast, даёт мало информации об отношениях между последовательностями. Геномы имеют недостаточную схожесть для работы megablast.
Однако, если сравнить его с графиком для blastn, можно увидеть некоторую схожесть. И, действительно, при понижении размера слова при использовании megablast, графики становятся более похожими.
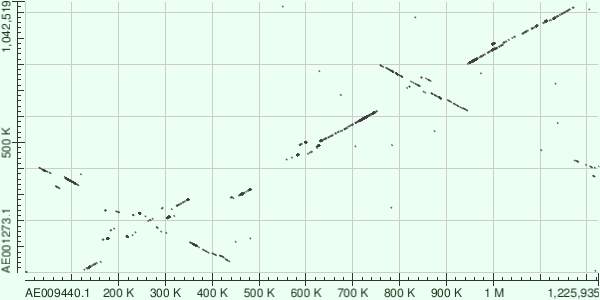
Рис.3 DotPlot при использовании megablast с длиной слова 16
График при blastn более информативен. Здесь видно несколько участков, для которых можно предположить гомологию (например, 960-1160 у AE009440.1 и 800-1000 у AE001273.1)
Также можно наблюдать две группы генов, в которых наблюдаются инверсии (на графике выглядят как прямые, идущие вниз)
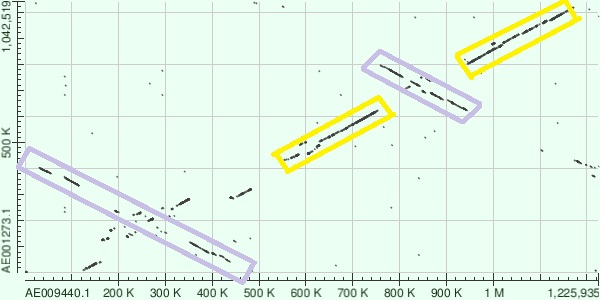
Рис.4 Комментарий к графикам. Сиреневыми рамками выделены инвертированные участки, для которых возможна гомология. Жёлтым выделены некоторые неинвертированные участки с возможной гомологией