Практикум 15
1. Подготовка чтений программой trimmomatic
Архив с чтениями был получен с помощью следующей команды:
Далее был создан файл с адаптерами:
Программа для удаления адаптеров:
Было удалено 130303 (или 1.76%) последовательностей
Удаление чтений с качеством ниже 20 и длиной меньше 32:
Было удалено 295585 (или 4.07%) чтений. Изначальный размер файла составил 167 М, после очистки - 156 М
2. Запуск velveth
Были подготовлены k-меры с k = 31:
3. Запуск velvetg
Далее проводилась сборка генома:
Далее были изучены основные параметры сборки. N50 составил 25646.
Таблица stats.txt была отсортирована при помощи программы sort -r -n -k 2 stats.txt | less. Таким образом, нашлись самые длинные контиги:
С помощью программы sort -r -n -k 6 stats.txt | less таблица была отсортирована по убыванию покрытия. В верху таблицы оказалось много фрагментов длины 1 с большим покрытием (например, ID 133 с покрытием 474299). Более содержательными находками оказались ID 60 - 181, ID 39 - 177, ID 30 -172
4. Анализ
Три самых больших контига были картированы на хромосому CP009253
ID 6
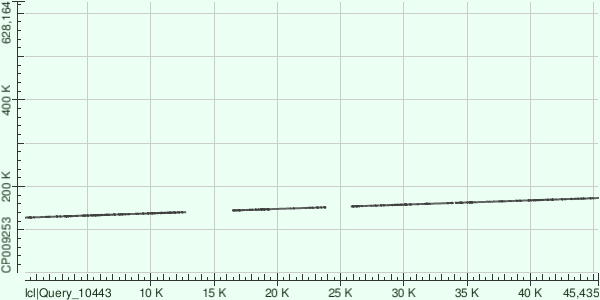
Выравнивание разбилось на 4 участка с негомологичными промежутками между ними. Четвёртый участок выровнялся дважды:
1. 127825-140555 (75% идентичных нуклеотидов, 4% гэпов)
2. 144368-151796 (78% идентичных нуклеотидов, 3% гэпов)
3. 153752-161738 (78% идентичных нуклеотидов, 3% гэпов)
4. 161898-166752 (80% идентичных нуклеотидов, 2% гэпов)
5. 166750-173180 (76% идентичных нуклеотидов, 2% гэпов)
ID 9
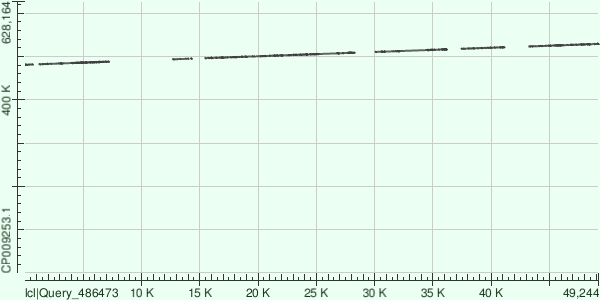
Выравнивание разбилось на 9 участков:
1. 480874-481545 (82% идентичных нуклеотидов, 2% гэпов)
2. 481997-488106 (74% идентичных нуклеотидов, 4% гэпов)
3. 493487-494864 (80% идентичных нуклеотидов, 0% гэпов)
4. 495033-495148 (90% идентичных нуклеотидов, 4% гэпов)
5. 496111-500325 (75% идентичных нуклеотидов, 3% гэпов)
6. 510438-516539 (79% идентичных нуклеотидов, 2% гэпов)
7. 517766-521500 (77% идентичных нуклеотидов, 2% гэпов)
8. 523105-528679 (77% идентичных нуклеотидов, 3% гэпов)
9. 528794-529211 (84% идентичных нуклеотидов, 6% гэпов)
ID 5
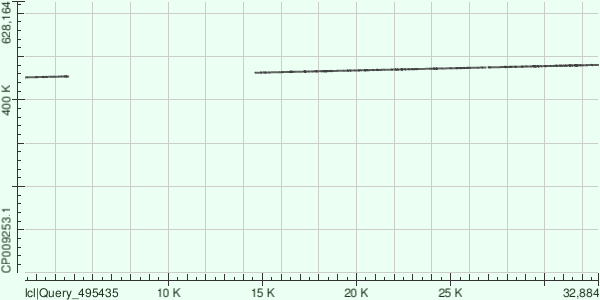
Выравнивание разбилось на 3 участка. третий участок выровнялся дважды:
1. 451729-454069 (77% идентичных нуклеотидов, 2% гэпов)
2. 462496-467421 (77% идентичных нуклеотидов, 3% гэпов)
3. 467412-474667 (77% идентичных нуклеотидов, 2% гэпов)
4. 474844-480660 (74% идентичных нуклеотидов, 4% гэпов)