Практикум 8
1. BLAST для контига
Я выбрала контиг JAKRYI020000379. Его размер составил 26140 bp, координаты CDS - 789..2,041. В этом контиге есть только один CDS.
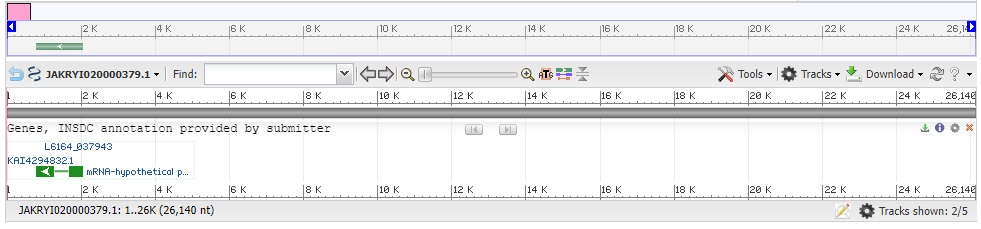
Рис. 1 Схема контига, зелёным обозначен CDS.
Ссылка на fasta-файл с последовательностью контига.
Далее я выполнила поиск BLAST с помощью разных алгоритмов. Во всех случаях я исключала таксон Magnoliophyta (цветковые растения). Результаты были следующими:
Megablast подходит для поиска очень похожих сиквенсов (например, когда у нас на руках непонятно чей ген материал и мы хотим узнать, что это), blastn подходит для поиска гомологов в менее родственных организмах. Blastx подходит для поиска гомологов белка, если у нас на руках только его нуклеотидная последовательность. Tblastx - для поиска гомологов белка по последовательностям, в которых белки не предсказаны
2. Поиск генов основных рибосомальных РНК по далёкому гомологу
Геном был проиндексирован при помощи программы makeblastdb. Сама программа:
< makeblastdb -in GCA_022379115.2_ASM2237911v2_genomic.fna -dbtype nucl -out db >
Позже я забустила blastn. Я выбрала его, потому что использовала нуклеотидные последовательности сильно разошедшихся организмов.
16S rRNA - малая субъединица рибосомы прокариот, 23S - большая.
< blastn -task blastn -query 16srRNA_ecoli.txt -db db.fasta -out result16.fasta >
< blastn -task blastn -query 23srRNA_ecoli.txt -db db.fasta -out result23.fasta >
Для 16s нашлось 124 результата, для 23s - 123. Для 16S нашлось 3 результата с скором около 1000 и хорошим Evalue. Они были найдены как на хомосомах, так и в скэффолде. Для 23S был найден один результат со скором выше 1000.
Мною были предприняты попытки выяснить, какие последовательности могут находиться в выровнявшихся местах. Однако, все результаты ложились "в пустоту". Возможно, последовательность была неверно аннотирована, из-за чего и не нашлось интересных результатов
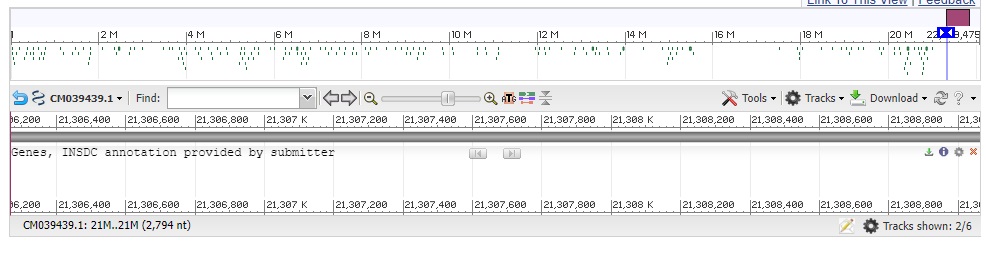
Рис. 2 Один из грустных результатов выравнивания