Практикум 4
1. 1. Составление списка гомологичных белков, включающих паралоги
Поиск гомологов белка CLPX_ECOLI осуществлялся при помощи следующих команд:
cat CLAMS.fasta CORDI.fasta COREF.fasta LEIXX.fasta NOCSJ.fasta RHOJR.fasta RUBXD.fasta STRAW.fasta > proteomes.fasta
makeblastdb -dbtype prot -in proteomes.fasta -out bac_db
blastp -query clpx_ecoli.fasta -num_threads 4 -db bac_db -evalue 0.001 -out blast_res.txt
В результаты был получен следующий список гомологов.
2. Реконструкция и визуализация
При помощи программы FastMe ("Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement", 100 бутстреп реплик) было реконструировано филогенетическое дерево найденных белков:
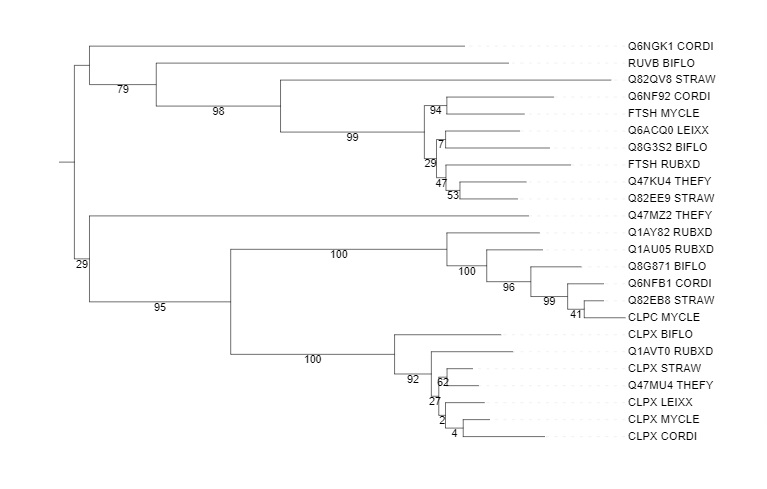
Рис. 1 Реконструированное филогенетическое дерево гомологов CLPX_ECOLI
Таким образом, можно выделить пары паралогов: Q1AY82_RUBXD и Q1AU05_RUBXD, Q82QV8_STRAW и Q82EE9_STRAW, RUVB_BIFLO и Q8G3S2_BIFLO, и пары ортологов:Q6NFB1_CORDI и CLPC_MYCLE, CLPX_MYCLE и CLPX_STRAW, Q82EE9 STRAW и Q47KU4 THEFY
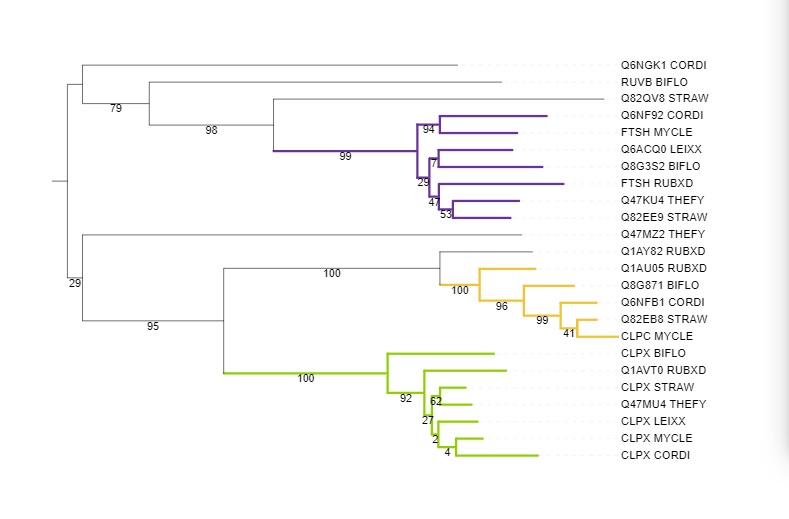
Рис. 2 Филогенетическое дерево гомологов CLPX_ECOLI с выделенными ортологическими группами
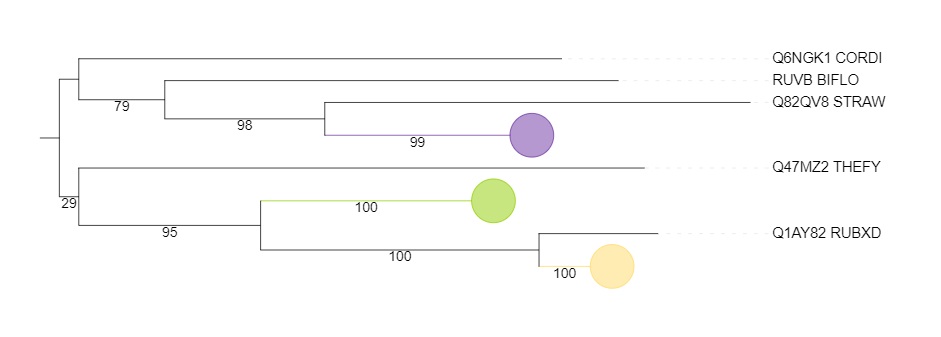
Рис. 3 Ортологические группы коллапсированы
Салатовым цветом обозначена группа АТФ-связывающих субъединиц протеаз Clp. Ветвь достаточно сильно отличается от исходного дерева бактерий. Можно заметить, что ветви сравнительно короткие, что может свидетелсьвтовать о быстрой эволюции и возможно неверном построении ветви.
Фиолетовым обозначена группа АТФ-зависимых цинковых металлопротеаз. Их филогения имеет очень высокое сходство с изначальным деревом бактерий, за исключением положения RUBXD.
Жёлтым цветом обозначена группа белков, связанных с Clp-протеазами. В группу вошли не все бактерии. Отношения белков из MYCLE, CORDI и STRAW соответствуют отношениям между бактериями.