Term4
Четвёртый семестр
Блок №1
1.Было создано изображение дерева, описанного заданной мне формулой (((А:70,B:70):30,(C:90,D:90):30):5,(E:70,F:70):5);.
Подписаны листья и длины ветвей.
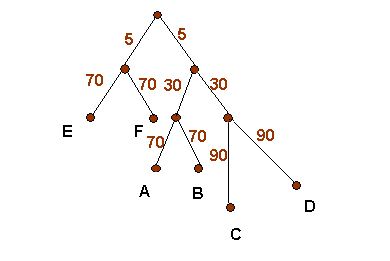 |
2.Ветви дерева были описаны с помощью таблицы как разбиения множества листьев.
|
A
|
B
|
C
|
D |
E |
F |
|
* |
* |
. |
. |
. |
. |
| . |
. |
* |
* |
. |
. |
| . |
. |
. |
. |
* |
* |
Получение искусственных мутантных последовательностей.
Считая, что в корне находится последовательность гена белка TIG_ECOLI, созданы возможные мутантные последовательности для листьев дерева.
Длина гена (Х17642) составляет 437 нуклеотидов.
Формулу для пересчёта расстояний в число мутаций- (длина гена*(число мутаций на 100 нуклеотидов)/100)
msbar tig_gene.fasta abcd.fasta -point 4 -count 22 -auto
msbar abcd.fasta ab.fasta -point 4 -count 131 -auto
msbar abcd.fasta cd.fasta -point 4 -count 131 -auto
msbar ab.fasta a.fasta -point 4 -count 306-auto
msbar ab.fasta b.fasta -point 4 -count 306 -auto
msbar cd.fasta c.fasta -point 4 -count 393 -auto
msbar cd.fasta d.fasta -point 4 -count 393-auto
msbar 2tptn.fasta ef.fasta -point 4 -count 22 -auto
msbar ef.fasta e.fasta -point 4 -count 306 -auto
msbar ef.fasta f.fasta -point 4 -count 306 -auto
4. На основе последовательностей, соответствующих листьям, реконструировано дерево алгоритмами UPGMA, Neighbor-joining и максимального правдоподобия.
С помощью следующей команды было построено филогенетичексое дерево алгоритмом максимального подобия:
Fdnaml all_leaves.fasta -ttratio 1 -auto
В итоге получилось следующее дерево:
+-------------b
|
| +----------------f
| +--4
| | +-----------------e
1--------3
| | +---------------------d
| +---2
| +--------------------c
|
+---------------a
Реконструкции алгоритмом UPGMA:
+-----------------------------a
+--------------1
! +-----------------------------b
+---4
! ! +----------------------------------e
! +---------2
--5 +----------------------------------f
!
! +-------------------------------------------c
+----3
+-------------------------------------------d
Видно, что реконструируя алгоритмом UPGMA, получаем укорененное дерево.
Реконструкция алгоритмом Neighbor-joining:
+------------b
!
! +---------------------c
! +----2
! ! +---------------------d
1---------3
! ! +-----------------e
! +--4
! +----------------f
!
+----------------a
UPGMA предполагает молекулярные часы и реконструирует укоренённое ультраметрическое дерево,
в то время как Neighbor-joining не предполагает МЧ и не укореняет деревья. Использовать алгоритм
UPGMA имеет смысл только в слу-чае объектов, которые эволюционируют с одинаковой скоростью.
UPGMA. При построении дерева вначале выбирается ближайшая пара таксонов.Далее из оставшихся
таксонов выбирается тот, среднее расстояние от которого до пары объединенных таксонов (кластера)
наименьшее.Процедура продолжается далее, причем расстояние между кластером X и кластером Y равно
среднему от парных расстояний между последовательностями этих кластеров. В итоге получаем дерево
с корнем.
Метод ближайшего соседа не требует постоянства скоростей замен по таксонам, он позволяет пра-
вильно восстанавливать топологию дерева, даже если такое постоянство нарушено. Сущность его заклю-
чается в определенном правиле пересчета эволюционных расстояний. Метод ближайшего соседа восста-
навливает топологию дерева без учета положения корня.
| Ветви
|
Различные деревья |
|
A |
B |
C |
D |
E |
F |
Дерево, построенное алгоритмом максимального подобия |
Дерево, построенное алгоритмом Neighbor-joining |
Дерево, построенное алгоритмом UPGMA |
| * |
* |
. |
. |
. |
. |
+ |
+ |
+ |
| . |
. |
* |
* |
. |
. |
+ |
+ |
+ |
| . |
. |
. |
. |
* |
* |
+ |
+ |
+ |
Как видно из таблицы, все полученные филогинетические деревья имеют одни и те же ветви.

