1. База данных OPM
Белок из α-спиралей
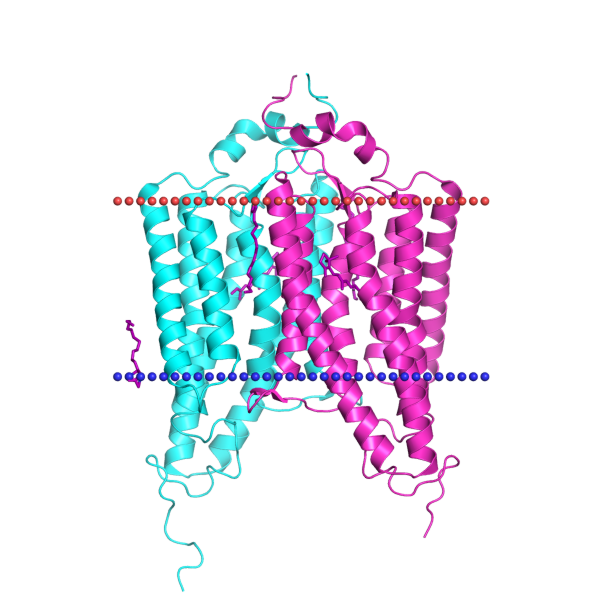
Для выполнения данного задания был выбран светозависимый анионный канал GtACR1 из криптофитовой водоросли Guillardia theta, структура которога представлена на рис. 1. Данный канал благодаря высокой проводимости аниона хлора потенциально можно использовать в качестве эффективного нейрон-ингибирующего "инструмента" в оптогенетике [1]. Остальную информацию о данном белке можно получить из таблицы 1.
| Название белка | Светозависимый анионный канал GtACR1 |
| PDB ID | 6csm |
| Вторичная структура в трансмембранной части | α-спирали |
| Толщина гидрофобной части белка в мембране (OPM) | 30.6 ± 0.6 Å |
| Координаты трансмембранных спиралей | Для цепей A, B: 31-52, 58-77, 92-110, 122-142, 147-168, 194-214, 223-245. |
| Среднее количество остатков в одной трансмембранной спирали | 21 (21.14) |
| В какой мембране находится белок | Эукариотическая плазматическая мембрана |
| Организм | Guillardia theta |
Белок из β-листов
Также для выполнения данного задания было необходимо рассмотреть белок, в трансмембранной части которого расположены β-листы; был выбран гемолитический лектин CEL-III (токсин, вырабатываемый голотуриями, представляет интерес для фармакологии), вторичная структура которого в трансмембранной части представлена в виде бета-бочонка (Рис. 2). CEL-III представляет собой гемолитический лектин, выделенный из морского огурца. Этот лектин состоит из двух углеводсвязывающих доменов (домены 1 и 2) и одного домена олигомеризации (домен 3). После связывания с поверхностью клетки доменов 1 и 2 домен 3 самоассоциируется с образованием трансмембранных пор, что приводит к лизису или гибели клеток [2]. Остальную информацию о данном белке можно получить из таблицы 2 (А ещё этот белок напоминает ядерный гриб после взрыва :D).
| Название белка | Гемолитический лектин CEL-III |
| PDB ID | 3w9t |
| Вторичная структура в трансмембранной части | β-листы |
| Толщина гидрофобной части белка в мембране (OPM) | 33.8 ± 0.8 Å |
| Координаты трансмембранных β-тяжей | Для цепей A, B, C, D, E, F, G: 318-330 И 340-353. |
| Среднее количество остатков в одном трансмембранной β-тяже | 14 (13.50) |
| В какой мембране находится белок | Секретированный (Secreted) |
| Организм | Cucumaria echinata (Sea cucumber) |

2. Анализ предсказания трансмембранных спиралей
Сравнение результатов выдачи программ TMHMM и Phobius с реальной структурной информацией из БД OPM
Для выполнения данного задания использовался белок - светозависимый анионный канал GtACR1 (6csm), содержащий только α-спирали в трансмембранной части. Поскольку данный белок является гомодимером, для анализа использовалась только цепь A.
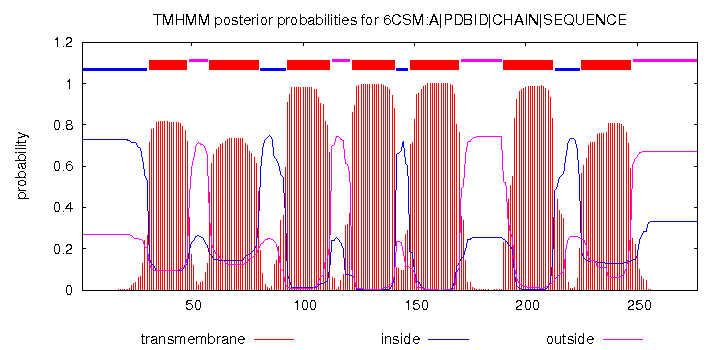
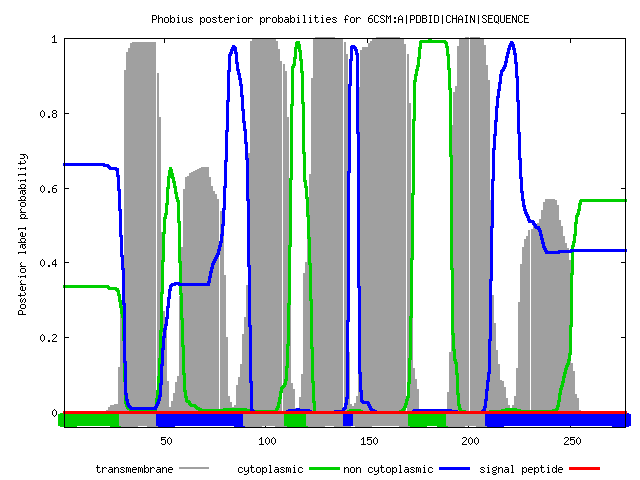
Согласно БД OPM белок содержит 7 участков α-спиралей, находящихся в мембране. Все эти участки довольно точно (отличие от реальных данных в большинстве случаев составляет не более 5 остатков) предсказал алгоритм TMHMM в отличие от Phobius, который верно предсказал лишь 5 участков из 7. Алгоритм Phobius не выявил участки с 58 по 77 а.о. и с 223 по 245 а.о.. Они лежат вне цитоплазмы согласно алгоритму.
Интересно, что при предсказании первого трансмембранного участка (31-52 в реальной структуре) оба алгоритма одинаково ошибаются на несколько остатков. Они приписывают 49-52 а.о. положение снаружи клетки, хотя они лежат внутри мембраны. По-видимому α-спираль более наклонена в реальной структуре, поэтому занимает больше трансмембранного пространства.
Таким образом, для данного белка предсказание алгоритма TMHMM оказалось точнее, чем Phobius.
Текстовая выдача TMHMM:
# 6CSM:A|PDBID|CHAIN|SEQUENCE Length: 277 # 6CSM:A|PDBID|CHAIN|SEQUENCE Number of predicted TMHs: 7 # 6CSM:A|PDBID|CHAIN|SEQUENCE Exp number of AAs in TMHs: 136.17393 # 6CSM:A|PDBID|CHAIN|SEQUENCE Exp number, first 60 AAs: 17.90022 # 6CSM:A|PDBID|CHAIN|SEQUENCE Total prob of N-in: 0.72999 # 6CSM:A|PDBID|CHAIN|SEQUENCE POSSIBLE N-term signal sequence 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 1 30 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 31 48 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 49 57 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 58 80 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 81 92 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 93 112 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 113 121 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 122 141 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 142 147 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 148 170 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 171 189 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 190 212 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 213 224 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 225 247 6CSM:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 248 277
Текстовая выдача Phobius:
Prediction of 6CSM:A|PDBID|CHAIN|SEQUENCE ID 6CSM:A|PDBID|CHAIN|SEQUENCE FT TOPO_DOM 1 30 CYTOPLASMIC. FT TRANSMEM 31 48 FT TOPO_DOM 49 91 NON CYTOPLASMIC. FT TRANSMEM 92 111 FT TOPO_DOM 112 122 CYTOPLASMIC. FT TRANSMEM 123 140 FT TOPO_DOM 141 145 NON CYTOPLASMIC. FT TRANSMEM 146 172 FT TOPO_DOM 173 191 CYTOPLASMIC. FT TRANSMEM 192 210 FT TOPO_DOM 211 277 NON CYTOPLASMIC.
Различие алгоритмов TMHMM и Phobius
Оба алгоритма, в том числе TMHMM [3] основаны на скрытой марковской модели, но Phobius может учитывать больше факторов, влияющих на положение белка внутри или вне мембраны.
При предсказании трансмембранных участков можно получить ошибочные результаты - программы часто интерпретируют N-концевые трансмембранные части белка как сигнальные пептиды, и наоборот, сигнальные пептиды обрабатываются как трансмембранные α-спирали, что связано с тем, что ядро сигнального пептида содержит длинный участок гидрофобных аминокислот (длиной около 5–16 остатков).
Веб-сервер Phobius предоставляет возможность принимать во внимание данную проблему, что делает предсказания данного алгоритма более точными [4]. Хотя для данного белка предсказание алгоритма TMHMM оказалось точнее, чем Phobius, что может свидетельствовать о том, что всё же лучше использовать несколько разных алгоритмов для поиска трансмембранных α-спиралей.
3. База данных TCDB - Transporter Classification Database
TC код обычно выглядит следующим образом: V.W.X.Y.Z, где V (число) - соответствует классу транспортера; W (буква) - подкласс транспортёров; X (число) - семейству транспортеров (иногда суперсемейство); Y (число) - подсемейство, в котором обнаружен транспортер, а Z соответствует конкретному транспортеру с конкретным субстратом или диапазоном транспортируемых субстратов.
При поиске по PDB ID был найден только гемолитический лектин CEL-III (3w9t) с TC кодом: 1.C.96.1.1, где 1 - каналы и поры, 1.C - порообразующий токсин, 1.C.96 - семейство гемолитических лектинов, CEL-III.
4. Список литературы:
- Li, H. et al. Crystal structure of a natural light-gated anion channelrhodopsin. Elife 8, (2019).
- Unno, H., Goda, S. & Hatakeyama, T. Hemolytic Lectin CEL-III Heptamerizes via a Large Structural Transition from α-Helices to a β-Barrel during the Transmembrane Pore Formation Process. J. Biol. Chem. 289, 12805–12812 (2014).
- Krogh, A., Larsson, B., von Heijne, G. & Sonnhammer, E. L. Predicting transmembrane protein topology with a hidden Markov model: application to complete genomes. J. Mol. Biol. 305, 567–80 (2001).
- Käll, L., Krogh, A. & Sonnhammer, E. L. L. Advantages of combined transmembrane topology and signal peptide prediction--the Phobius web server. Nucleic Acids Res. 35, W429-32 (2007).