1. Анализ качества чтений
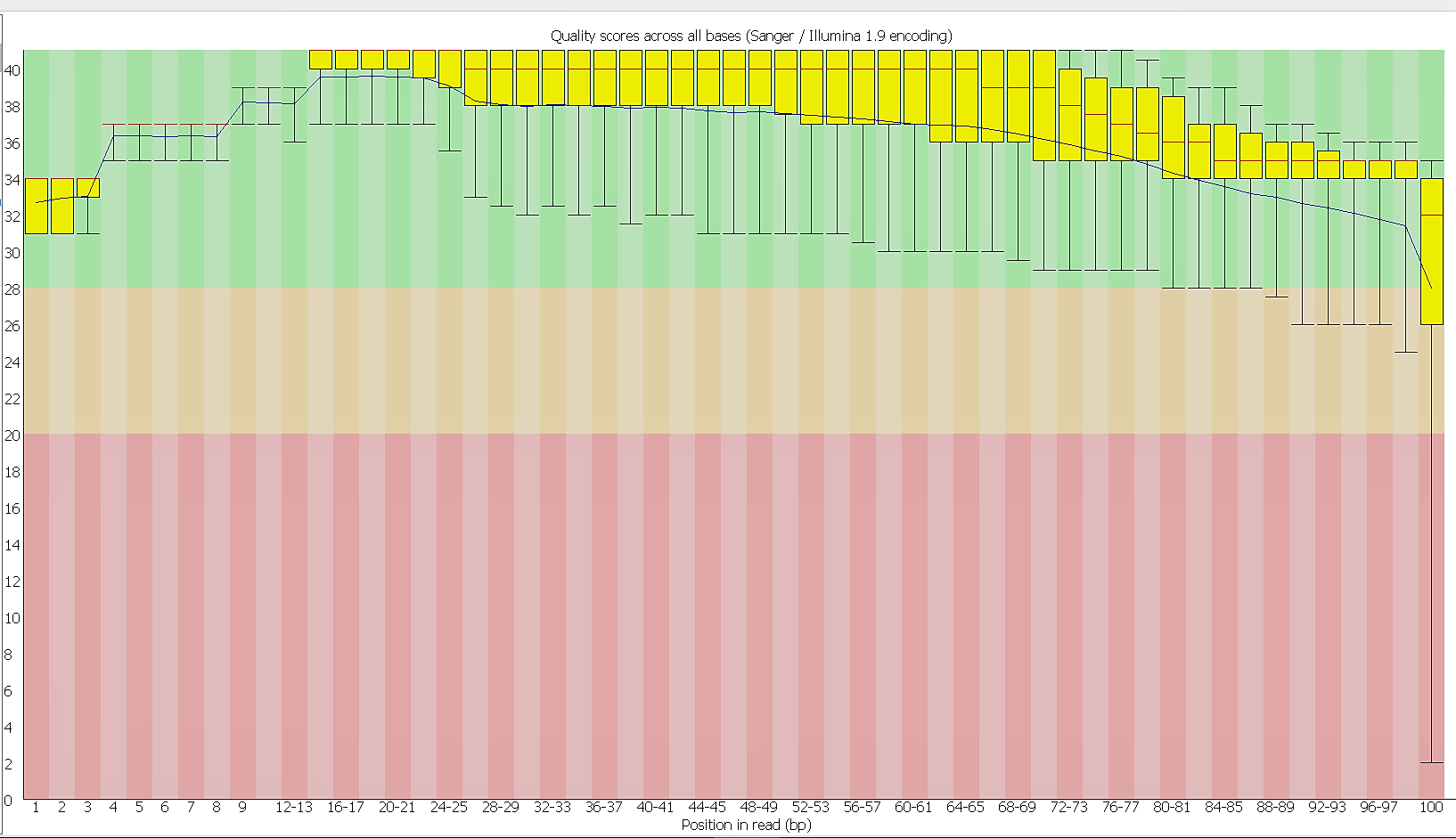
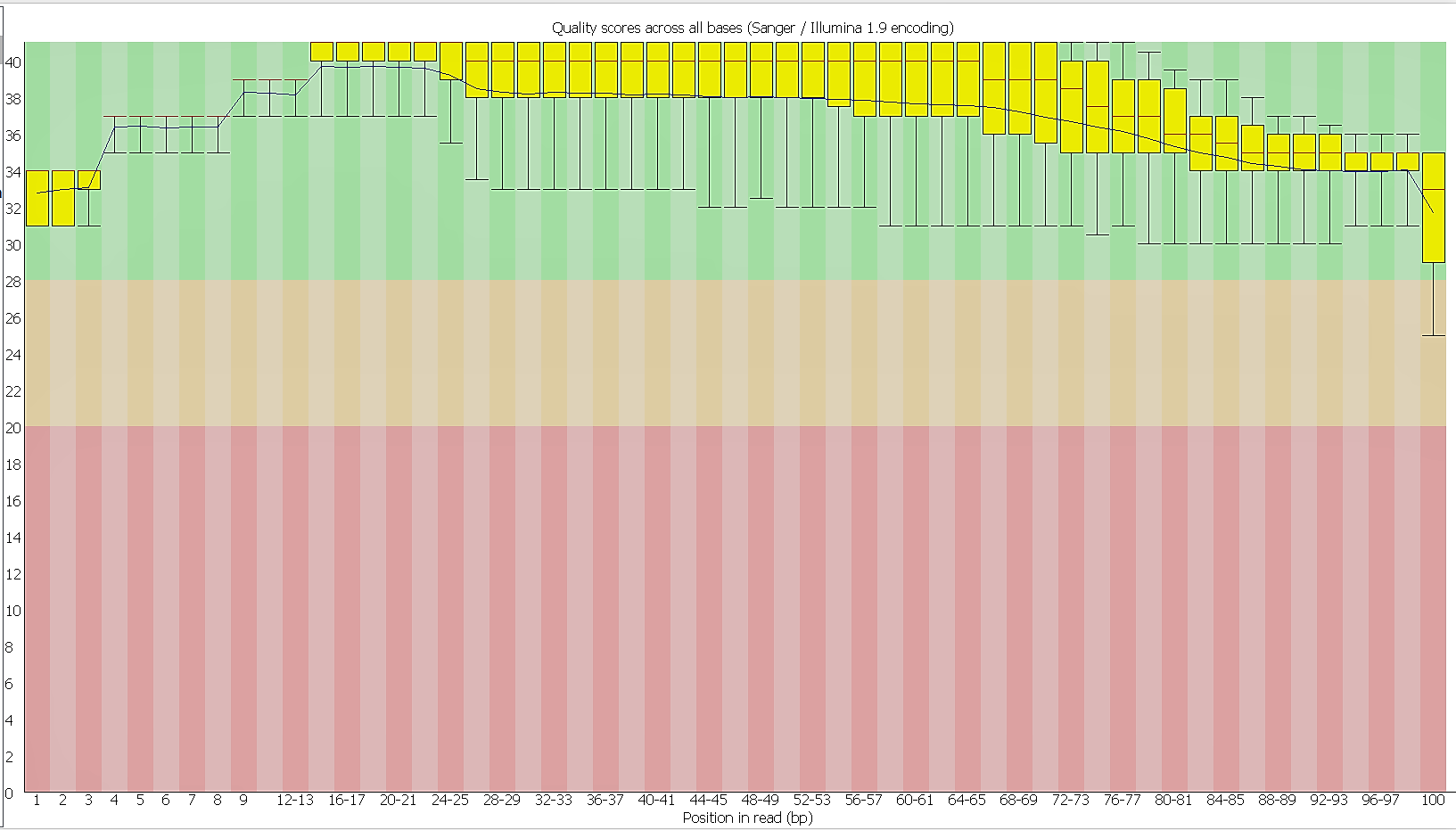
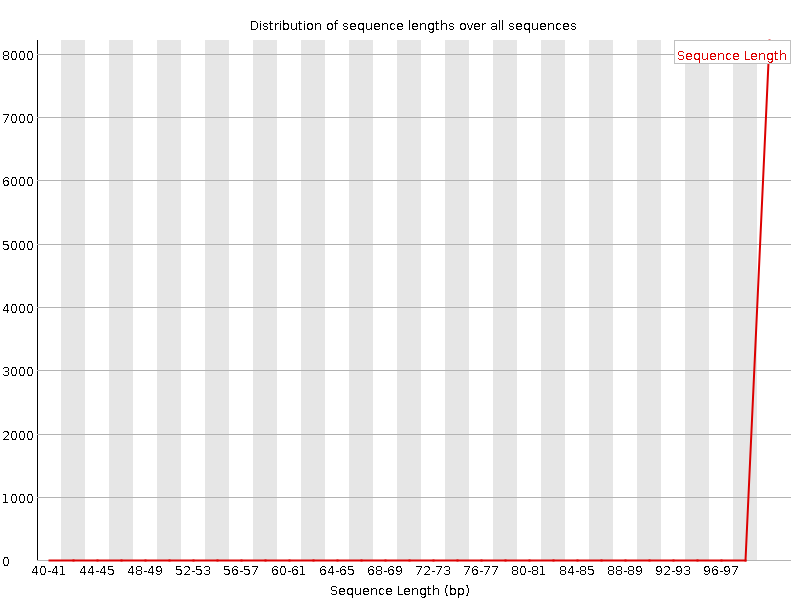
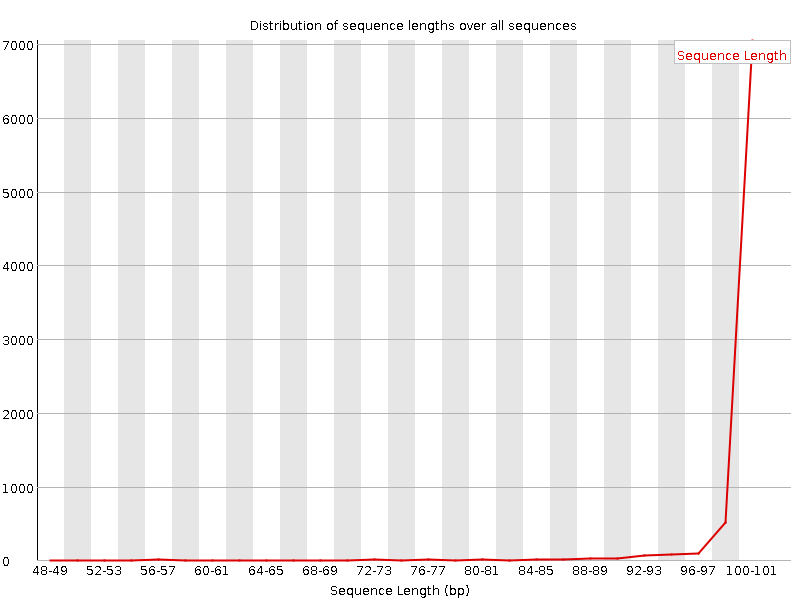
| Команда | Функция |
|---|---|
| hisat2-build chr5.fasta chr5 | Индексирует референсную последовательность |
| hisat2 --no-spliced-alignment --no-softclip -x chr5 -U chr5good.fastq -S chr5_al.sam | Строит выравнивание референсной
последовательности и прочтений и сохраняет результат в файл chr5_al.sam (опция '--no-spliced-alignment' запрещает разбивать чтения и выравнивать их отдельными участками, опция '--no-softclip' запрещает подрезать чтения с концов) |
| samtools view chr5_al.sam -b -o chr5_al.bam | Переводит выравнивание в бинарный формат |
| samtools sort chr5_al.bam -o chr5_al_s.bam | Сортирует выравнивание чтений с референсом по координате в референсе начала чтения |
| samtools index chr5_al_s.bam | Индексирует отсортированный .bam файл |
| Команда | Функция |
|---|---|
| samtools mpileup -uf chr5.fasta alignsort.bam -o snp.bcf | Создает файл с полиморфизмами в формате .bcf |
| bcftools call -cv chr5_al.bcf -o chr5_al.vcf | Создает файл со списком отличий между референсом и чтениями в формате .vcf |
| perl ../../annovar/convert2annovar.pl.old -format vcf4 chr5_al.vcf | grep - v '-' | more | Создает файл, необходимый для работы annovar |
| perl ../../annovar/annotate_variation.pl.old -build hg19 -out snp.ref chr5_ an.txt ../../annovar/humandb/ | Аннотирует полученные snp по базе данных refgene |
| perl ../../annovar/annotate_variation.pl.old -filter -out snp.dbsnp -build hg19 -dbtype snp138 chr5_an.txt ../../annovar/humandb/ | Аннотирует полученные snp по базе данных dbsnp |
| perl ../../annovar/annotate_variation.pl.old -filter -out snp.1000g -buildv er hg19 -dbtype 1000g2014oct_all chr5_an.txt ../../annovar/humandb.old/ | Аннотирует полученные snp по базе данных 1000 genomes |
| perl ../../annovar/annotate_variation.pl.old -regionanno -out snp.gwas -bui ld hg19 -dbtype gwasCatalog chr5_an.txt ../../annovar/humandb.old/ | Аннотирует полученные snp по базе данных Gwas |
| perl ../../annovar/annotate_variation.pl.old -filter -dbtype clinvar_201506 29 -build hg19 -out snp.clinvar chr5_an.txt ../../annovar/humandb.old/ | Аннотирует полученные snp по базе данных Clinvar |
| Координата | Референсная последовательность | Чтения | Тип полиморфизма | Глубина покрытия | Качество чтений |
|---|---|---|---|---|---|
| 35861068 | T | C | Замена нуклеотида | 44 | 221.999 |
| 35857308 | T | TC | Вставка нуклеотида | 40 | 177.458 |
| 74639544 | CTTGTATTGT | CTTGT | Делеция нуклеотидов | 23 | 73.4665 |
| Координата | SNP | Quality и DP | refgene | dbsnp | 1000 genomes | Gwas | Clinvar |
|---|---|---|---|---|---|---|---|
| 35857177 | G C | 221.999 113 | intronic IL7R hom | rs1353252 | 0.647364 | ||
| 35857235 | C G | 221.999 83 | intronic IL7R hom | rs1494561 | 0.647564 | ||
| 35857262 | A G | 221.999 58 | intronic IL7R hom | rs1353250 | 0.647564 | ||
| 35860780 | G T | 21.0411 8 | intronic IL7R het | ||||
| 35861068 | T C | 221.999 44 | exonic IL7R hom | rs1494558 | 0.59984 | * | |
| 35861152 | C G | 176.009 30 | intronic IL7R het | rs11567705 | 0.233826 | ||
| 35861268 | T G | 38.7651 2 | intronic IL7R hom | rs969129 | 0.667332 | ||
| 35867343 | T C | 184.009 32 | intronic IL7R het | rs73750058 | 0.00838658 | ||
| 35870814 | A C | 32.7667 2 | intronic IL7R hom | rs10063445 | |||
| 35871010 | C T | 126.008 47 | intronic IL7R het | rs6893892 | 0.0247604 | ||
| 35871190 | G A | 221.999 98 | exonic IL7R hom | rs1494555 | 0.666933 | * | |
| 35871463 | T C | 111.008 20 | intronic IL7R het | rs9282751 | 0.0247604 | ||
| 35873899 | C A | 3.0136 3 | intronic IL7R het | ||||
| 35874575 | C T | 225.009 164 | exonic IL7R het | rs6897932 | 0.172524 | Multiple sclerosis,Type 1 diabetes | * |
| 35875593 | A T | 221.999 41 | intronic IL7R hom | rs987106 | 0.44988 | ||
| 35910419 | C T | 221.999 53 | intronic CAPSL hom | rs1445899 | 0.525359 | ||
| 35910529 | C T | 221.999 92 | exonic CAPSL hom | rs1445898 | 0.525359 | Type 1 diabetes | |
| 35921069 | T C | 221.999 28 | intronic CAPSL hom | rs6859892 | 0.859625 | ||
| 35937050 | T C | 11.3429 1 | intronic CAPSL hom | rs6890660 | 0.926917 | ||
| 74633975 | C T | 8.64911 1 | intronic HMGCR hom | ||||
| 74639269 | C T | 22.7872 2 | intronic HMGCR hom | rs149363137 | 0.0321486 | ||
| 74639546 | T C | 179.014 19 | intronic HMGCR hom | rs80116386 | 0.047524 | ||
| 74642848 | A T | 26.0177 6 | intronic HMGCR het | rs17244834 | 0.432308 | ||
| 74647886 | T C | 11.3429 1 | intronic HMGCR hom | ||||
| 74651084 | A G | 221.999 90 | intronic HMGCR hom | rs3846662 | 0.625 | LDL cholesterol,Cholesterol, total | |
| 74652326 | T G | 83.0076 10 | intronic HMGCR het | rs17244883 | 0.0247604 | ||
| 74655726 | C T | 58.0073 5 | intronic HMGCR het | rs3846663 | 0.405751 | cholesterol,Quantitative traits | |
| 74656539 | T C | 225.009 58 | UTR3 HMGCR (NM_000859:c.*372T>C, NM_001130996:c.*372T>C) het | rs12916 | 0.416134 | Cholesterol, total,LDL cholesterol |