1.Подготовка чтений
Изображения 'Per base sequence quality' и проекты FastQC чтений:
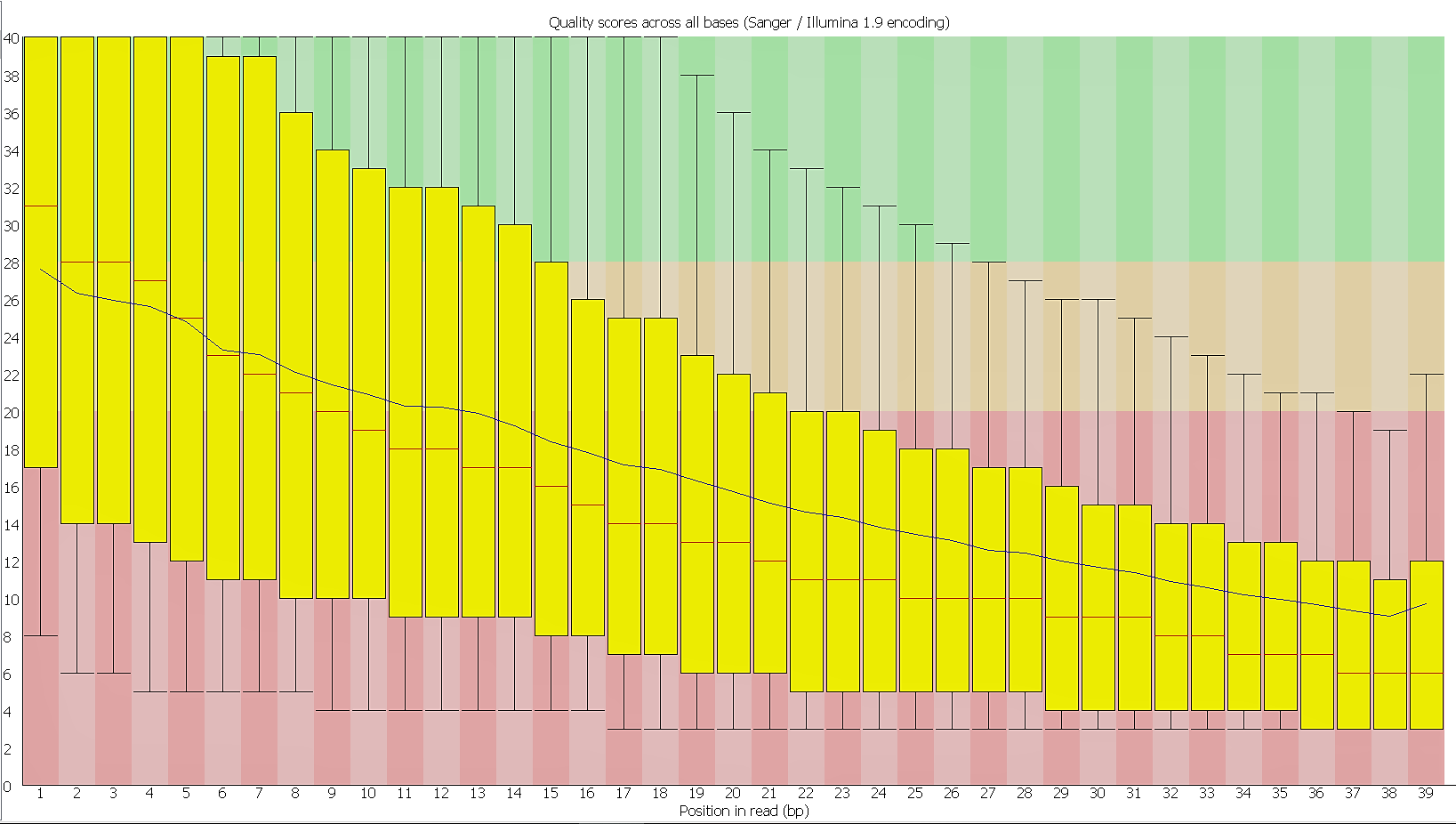
1) До очистки чтений:
-ссылка на FastQC-проект
- Изображение 'Per base sequence quality':
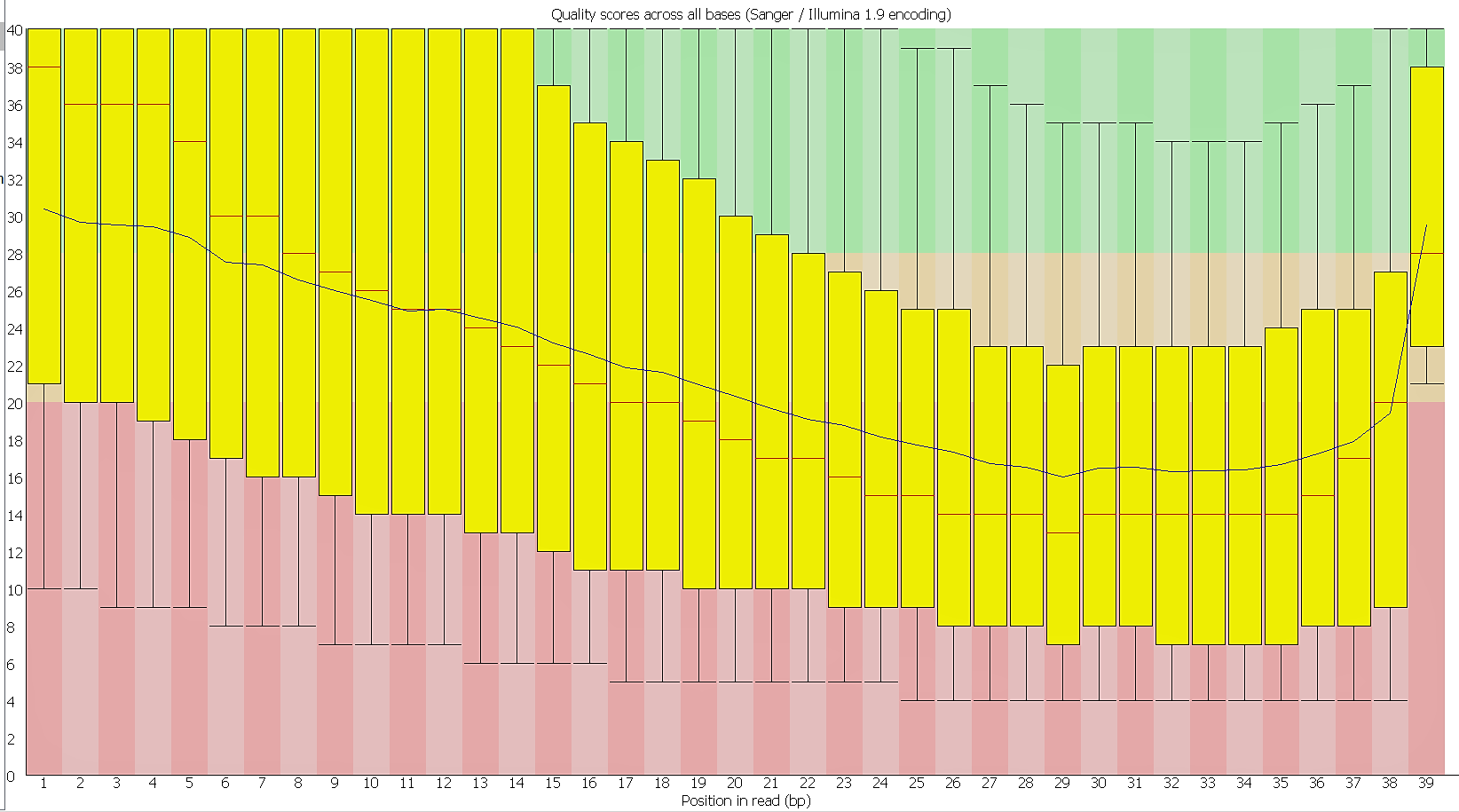
2) После очистки Trimmomatic:
-ссылка на FastQC-проект
- Изображение 'Per base sequence quality':
Итог:
- Число чтений
До очистки - 12950609
После - 6344028
-Длина чтений
До очистки - 39
После - 30-39
В итоге было удалено 6606578 чтений, получившихся после чистки длиной менее 30 пар нуклеотидов
- Использованные команды:
fastqc SRR.fastq
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR.fastq SRR_trim.fastq ILLUMINACLIP:adapters.fasta:2:7:7
TRAILING:20 MINLEN:30
2. Создание k-меров и построение генома
- N50 = 2960
-Максимальная длина контига 15418, покрытие- 51.135621 (ID 2)
На втором месте- длина 15093, покрытие 37.135096 (ID 36)
На третьем месте- длина 10741, покрытие 38.155386 (ID 62)
- Два контига с длиной 1 имеют покрытия 1760 и 31848 (ID 690 и 691 соответственно), в то время как медиана
для покрытий равняется 8. Контиг с ID 1 имеет длину 1 и покрытие 597662
Использованные команды:
velveth kmers 29 -fastq -short SRR_trim.fastq
velvetg kmers
Информация о контигах бралась из 'stats.txt'
3. Megablast. Hаложение контигов на геном
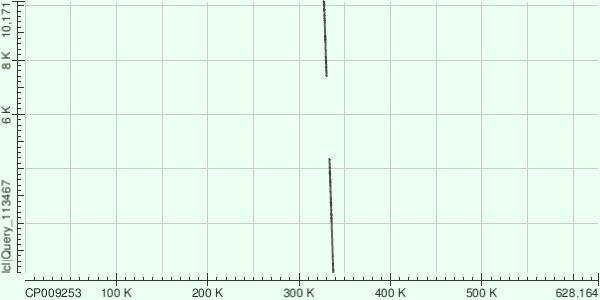
1) Для контига с ID 15:
- Dot matrix контига:
- Характеристика всего выравнивания:
- Total score = 3619, Query cover = 1%, E value = 0, Ident = 77%
- Контиг соответствует участку хромосомы с координатами [167:10171]
- Всего было построено 2 выравниваний со следующими координатами:
[167:4358]
[7402:10171]
- Все эти выравнивания имеют обратную направленность (-/+) и Expect = 0
- Процент гэпов в выравниваниях- 3%, Identities - 76% и 77%
- В итоге нельзя сказать, что данный контиг полностью лег на участок генома, так как все же слишком большой не схожий участок.
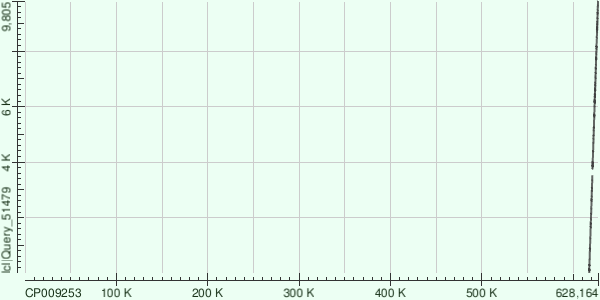
2) Для контига с ID 36:
- Dot matrix контига:
- Характеристика всего выравнивания:
- Total score = 5147, Query cover = 1%, E value = 0, Ident = 76%
- Контиг соответствует участку хромосомы с координатами [2:9805]
- Всего было построено 2 выравниваний со следующими координатами:
[3756:9805]
[2:3526]
- Все эти выравнивания имеют положительную направленность (+/+) и Expect = 0
- Процент гэпов в выравниваниях- 2% и 3%, Identities - 76% и 78%
- В итоге можно сказать, что данный контиг почти полностью лег на участок генома, так как не схожий участок не очень большой.
3) Для контига с ID 27:
- Dot matrix контига:
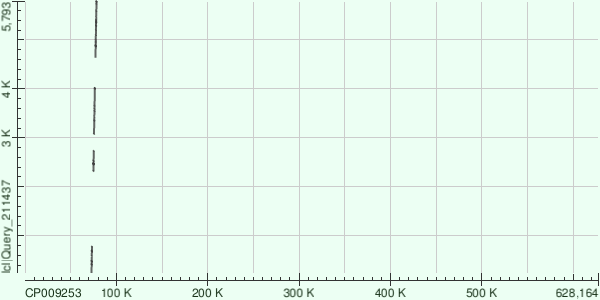
- Характеристика всего выравнивания:
- Total score = 1489, Query cover = 0%, E value = 1e-153, Ident = 77%
- Контиг соответствует участку хромосомы с координатами [240:5793]
- Всего было построено 4 выравниваний со следующими координатами:
[3077:4021]
[4635:5793]
[240:781]
[2319:2736]
- Все эти выравнивания имеют положительную направленность (+/+) и Expect от 1e-153 до 6e-87
- Процент гэпов в выравниваниях- 2% - 7%, Identities - 73% - 78%
- В итоге можно сказать, что данный контиг не лег полностью на участок генома, так как сликом много несовпадающих участков.




