Ссылки на файлы с результатом:
JalView проект выравнивания прямой и обратной последовательности
Обоснование решений для четырех
проблемных нуклеотидов или полиморфизмов:
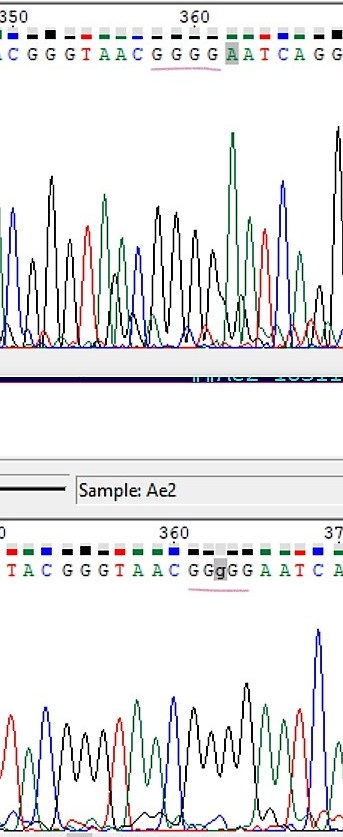
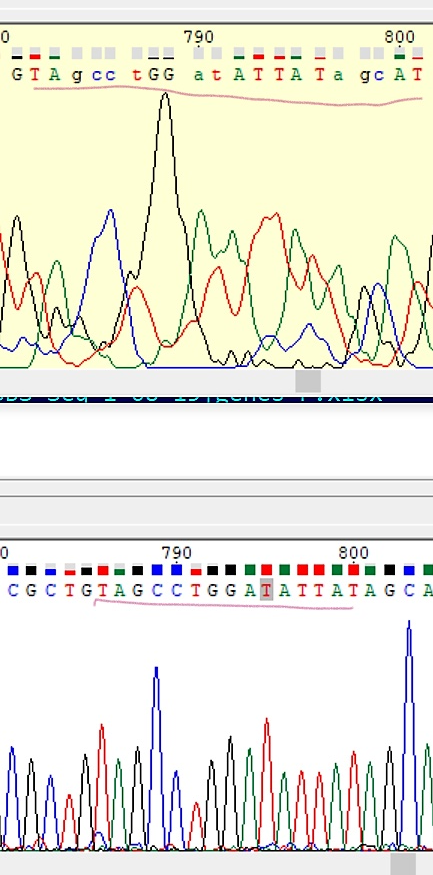
1) В этом месте (363 нуклеотид обратной последовательности) был пропущен гуанин. Я его добавила,
так как в прямой последовательности нет сомнений в его наличии в данном месте.
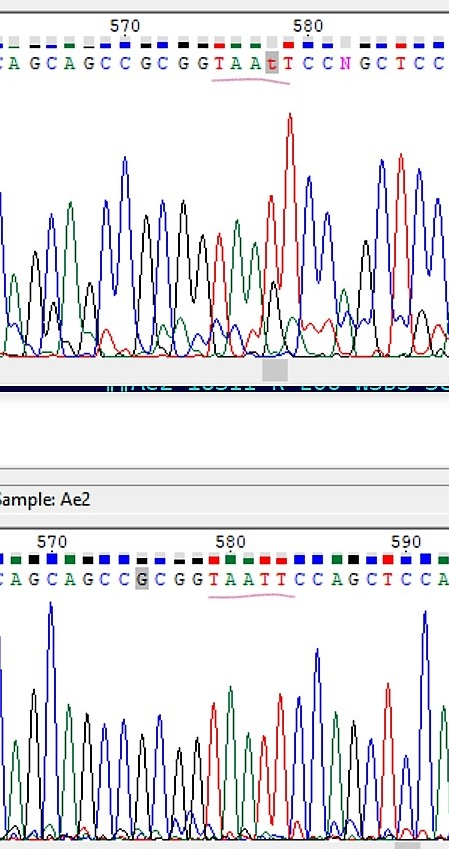
2) В 578 позиции прямой последователности стоял неизвестный нуклеотид (N). Но так как в обратной последователности на этом
месте явный тимин, то в пямой я сделала такую же замену (к тому же пик гуанина меньше середины пика тимина, что так же
поддтверждает, что такая замена имеет месте в этой позиции).
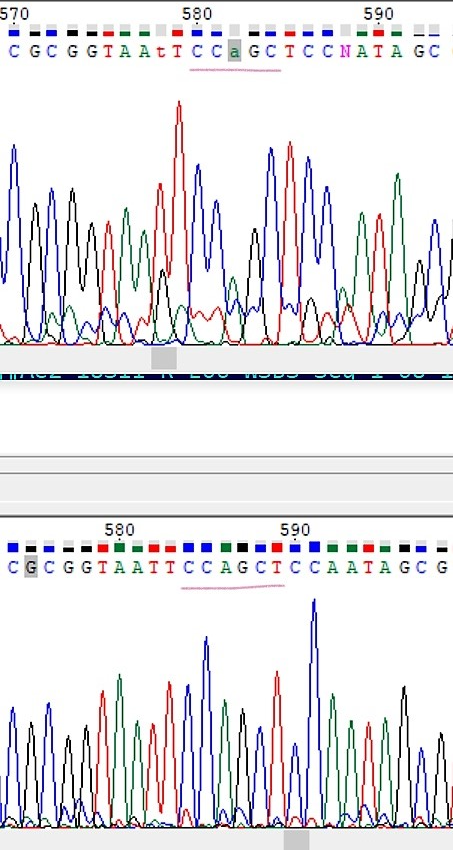
3) В 582 позиции прямой последовательности неизвестный нуклеотид я заменила на аденин, так как в обратной поседовательности
на этом месте точно стоит аденин, к тому же втрой пик очень сильно похож на шум.
4) Даже в таком запутанном участке была возможность разобраться, благодаря отличной хромaтограмме обратной последовательности.
Конечно, есть некая вероятность, что там мог быть полиморфизм, но она все же меньше, чем вероятность комплиментарности.
Характеристика хроматограммы в целом:
1) длины начального и конечного нечитаемых участков:
-прямая последовательность: 5' 1-52
3' 812-940
- обратная последовательность: 5' 816-946
3' 1-164
2) И в прямой, и в обратной последовательностях сигнал к шуму относится примерно 3:1
3) В целом хроматограмма мне показалась довольно неплохой, к тому же неясности прямой последовательности часто можно
было было прояснить, посмотрев на обратную.
Ссылки на исходные файлы в .ab1 формате
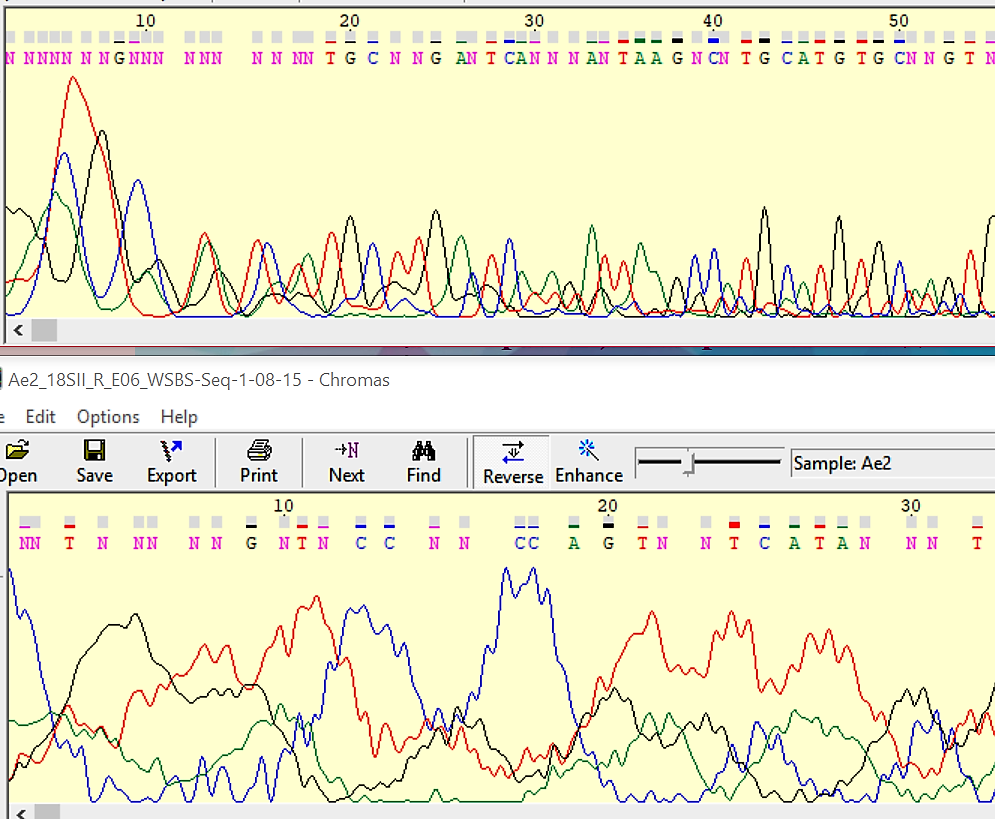
В качестве примера нечитаемого фрагмента я взяла начальный кусок данных мне хроматограмм.
В некоторых местах все же наблюдается комплиментарность, но в целом разобрать их невозможно.