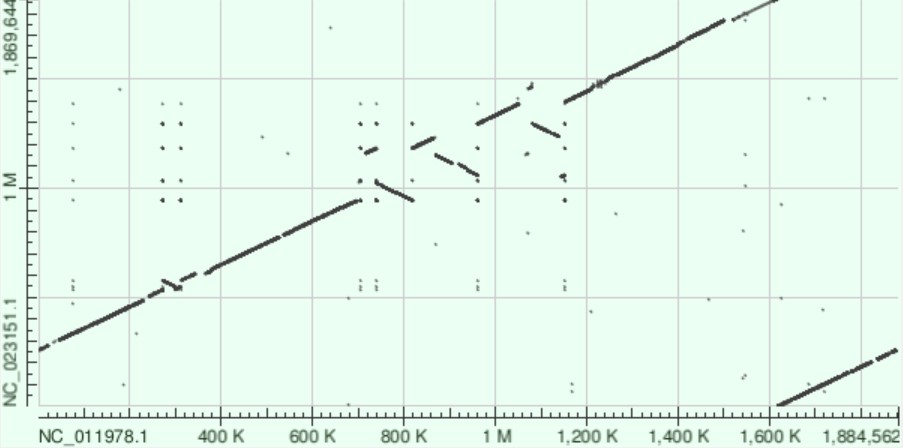
Для выравнивания были выбраны нуклеотидные последовательности двух видов термофильных бактерий одного рода: Thermotoga maritima (AC: NC_023151.1) и Thermotoga neapolitana (AC: NC_011978.1).
Геномы были найдены на сайте NCBI с помощью 'Browse by Organism'. Далее были построены карты локального сходства с помощью Megablast (рис.1) и blastn (рис.2).

Рис.1. DotPlot при использовании Megablast.
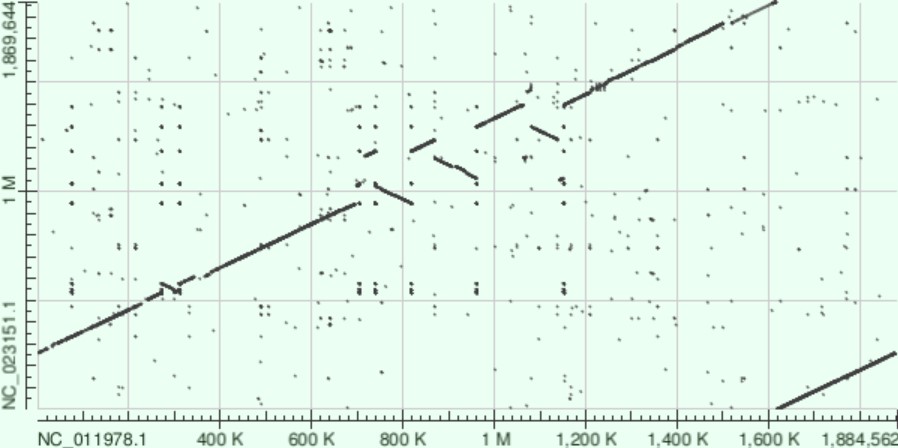
Рис.2. DotPlot при использовании blastn.
Можно сделать вывод, что точки начала плазмид были выбраны по разному. Участок в правом нижнем углу скорее всего является настоящим началом, а большая линия - его продолжением. Выбраны одинаковые цепи, а не комплементарные (т.к. прямая выравнивания идет по диагонали вверх).
Наблюдаются 4 инверсии на участках 290k-300k, 660k-820k, 860k-960k, 1m 90k-1m 140k (примерно). Кроме того, в megablast есть пробел в участке 510 k , который в blastn выровнялся. Скорее всего это mismatch.
В выдаче blastn появляется много черных точек. Это гомологичные повторы в геномах.