
Из базы данных Pfam был выбран домен белков теплового шока HSP70 (PF00012). HSP 70 связывается с другими белками в клетке, помогая им сохранять свою пространственную конформацию и предотвращая их неправильное сворачивание.
Выравнивание seed для этого семейства включает в себя 27 последовательностей и имеет длину 691 аминокислотный остаток. Файл был открыт в Jalview. При пороге Identity Treshold, равном 80%, нашлось несколько консервативных мотивов с высоким IC. Я выбрала мотив D[LF]GGGTFD.S, который встречается в 24 последовательностях.
Стоит отметить, что на второй позиции L, который содержится в 23 из 24 последовательностей, заменился в трех последовательностях, в которых не обнаружен мотив, на I и M, похожие по структуре. Аналогично в 5 позиции G во всех трех заменился на S, в 6 T заменился на S в двух из трех случаев, в 7 - F на Y в двух случаях. В 9 позиции у 26 последовательностей обнаружен V, A или I, которые также похожи по свойствам.

Рис 1. Паттерн, найденный в выравнивании. Окраска в стиле Clustal.
Был произведён поиск по данному мотиву в базе данных SwissProt на сайте MyHits. Была найдено 1141 находка. При этом среди этих находок были либо гомологи HSP70 (HscA, SSB, SSA4), либо непосредственно сам HSP70. Результат поиска можно увидеть здесь.
По выравниванию было построено дерево алгоритмом UPGMA. В итоге было выделено 7 групп: 5 тривиальных, в одной 5 последовательностей и в одной 17 последовательностей.
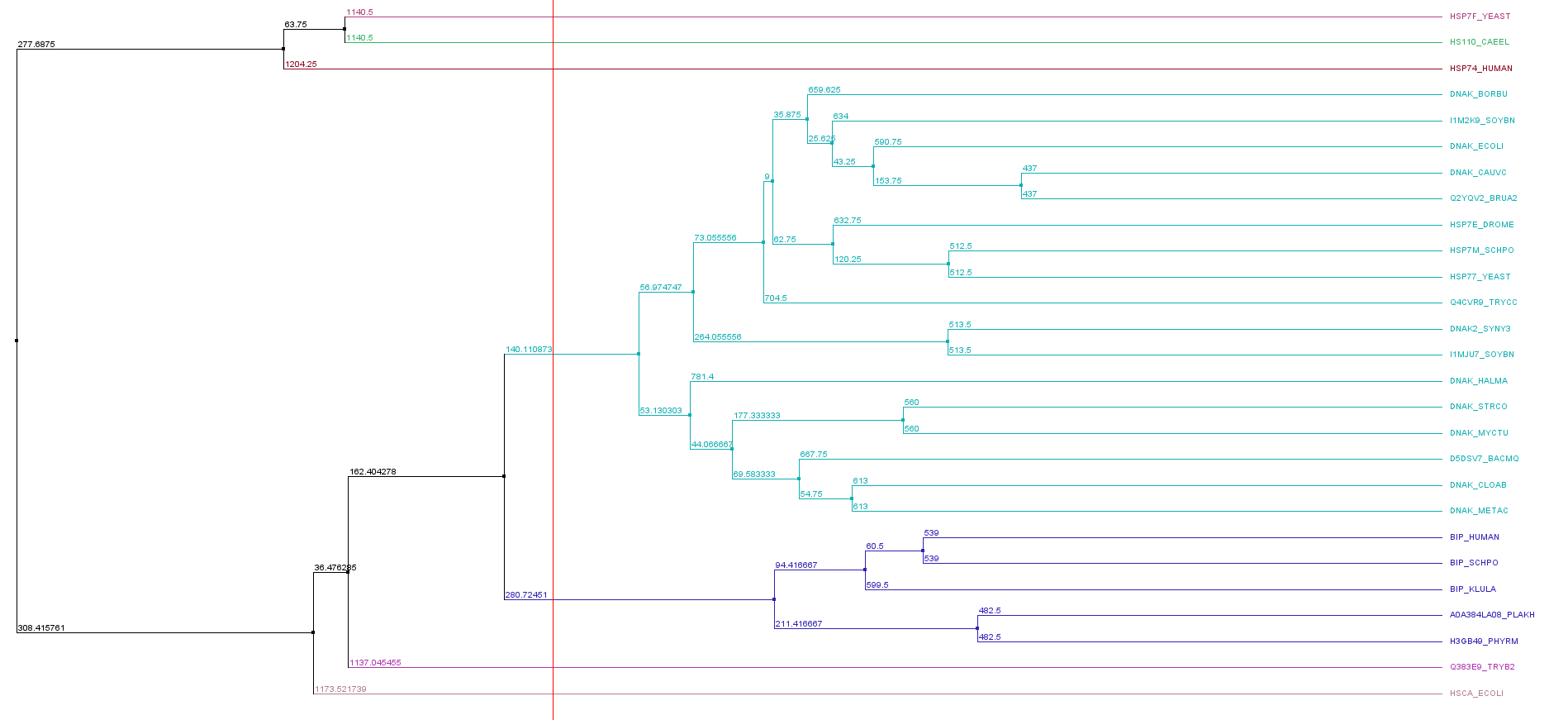
Рис 2. Дерево, построенное на основе выравнивания.
Я выбрала группу из 17 последовательностей, которая выделена на рис. 2 бирюзовым цветом.
Был найден мотив с паттерном LLDV[TS]PL[ST]LG.E[TV]. Данный мотив встречался 17 раз в выравнивании выбранной группы, а также 17 раз в выравнивании всех последовательностей. Таким образом, данный мотив специфичен для выбранной клады.
Файл с выравниванием в формате fasta.
Для анализа был выбран белок с AC Q7VDL2. Он является ингибитором клеточного деления, блокирующим образование полярных Z-кольцевых перегородок и принадлежит бактерии Prochlorococcus marinus (strain SARG / CCMP1375 / SS120).
Таблица 1. Результаты итераций PSI-BLAST.
| Номер итерации | Число находок выше порога | Идентификатор худшей находки выше порога | E-value находки | Идентификатор лучшей находки ниже порога | E-value находки |
|---|---|---|---|---|---|
| 1 | 146 | Q9AG20.1 | 0,005 | A8GFG7.1 | 0.005 |
| 2 | 188 | B6JKX0.1 | 7,00E-08 | - | - |
| 3 | 188 | Q9ZM51.1 | 2,00E-12 | A7H8E6.1 | 0.014 |
| 4 | 189 | A8MHK8.1 | 0,001 | A7H8E6.1 | 0.013 |
Уже на второй итерации оформилось семейство. На 4 итерации добавился посторонний белок, обладающий другой функцией. Об этом говорит очень малое различие E-value у него и лучшей находки ниже порога.
Для анализа была выбрана хромосома бактерии Bacillus Subtilis штамма 168. Она имеет длину 4220000 пар нуклеотидов. Ожидаемое число ТА в геноме составило 336259, реальное число - 218025. При этом p-value = 0.0 и такое отличие является статистически значимым.