
С помощью Excel был выбран подходящий по требованиям домен GAGA (GAGA factor). Члены этого семейства связываются с 5'-GAGAG-3' консенсусным сайтом связывания ДНК и содержат ядро цинкового пальца Cys2-His2. Ниже представлены некоторые характеристики данного семейства:
Для домена известны 26 архитектур. Для изучения была выбрана двухдоменная архитектура Q2PDY2_DROME, состоящая из BTB И GAGA. Она встречается в 77 из 186 последовательностей.

Был скачан файл со всеми последовательностями домена. Далее был составлен список их АС c помощью скрипта. Далее был составлен список всех АС с нужной нам доменной архитектурой и был создан файл с их последовательностями.
Полученный файл был выровнен в Jailview, также были удалены фрагменты до первого домена и после второго, лишние последовательности и последовательности с большим процентом идентичности. Результаты можно увидеть здесь.
Для построения профиля использовался пакет HMMER. Были выполнены следующие команды:
hmm2build HMM aln.fa
hmm2calibrate HMM
hmm2search --cpu=1 -E 1 HMM full.fasta> results.txt
В результате был получен НММ-профиль двухдоменой структуры длиной 374 и файл с результатами поиска профиля по набору белков с выбранным доменом.
Для анализа профиля была построена таблица со следующими важными для анализа столбцами:
C помощью формул для нее были посчитаны значения specificity, 1 - sensitivity и F1. По данным в таблице были построены графики, которые приведены ниже.
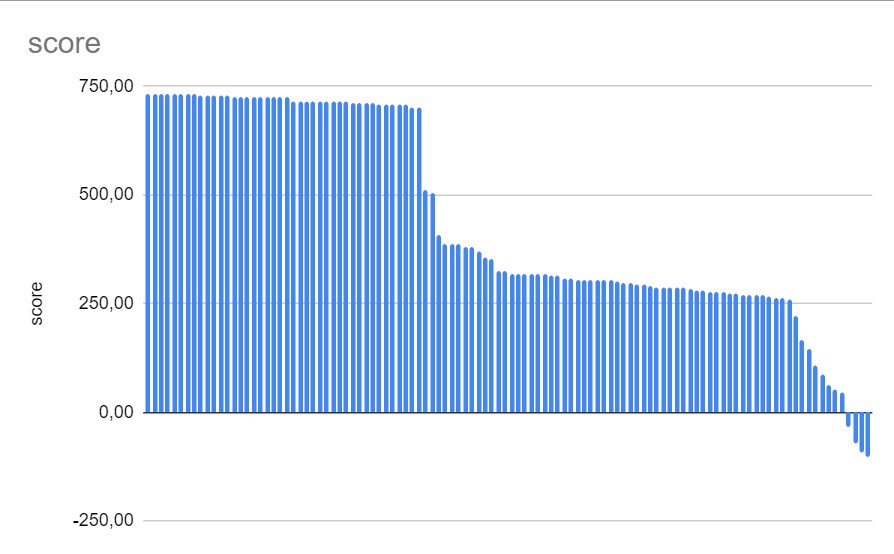
Рис.2. Распределение весов последовательностей.
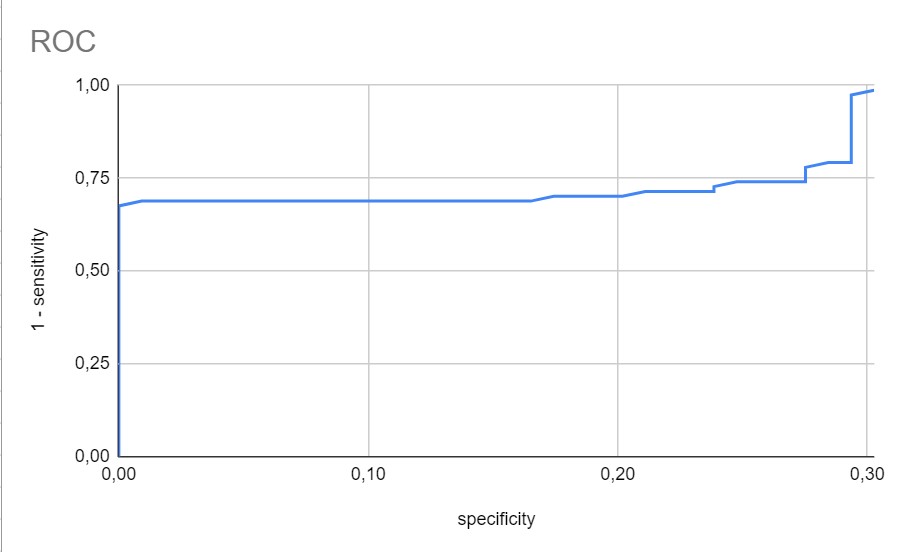
Рис.3. ROC-кривая.
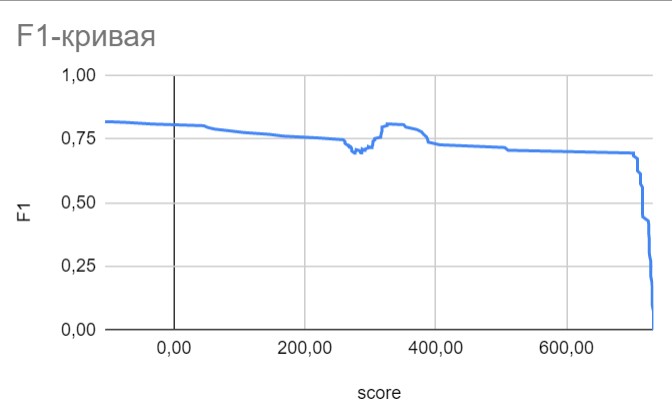
Рис.4. F1-кривая.
Исходя из графика зависимости F1 от веса, можно сказать, что порог веса, обеспечивающий наибольшие точность и полноту, равен 351. Точка максимума, равная 0,81, данного графика соответствует оптимальному соотношению sensitivity и specificity. Полученный порог прошли 54 последовательности из 77, которые имеют данную доменную архитектуру. Возможно, это произошло из-за слишком жесткой ревизии. При этом, на графике распределения весов последовательности этот порог не очевиден визуально.