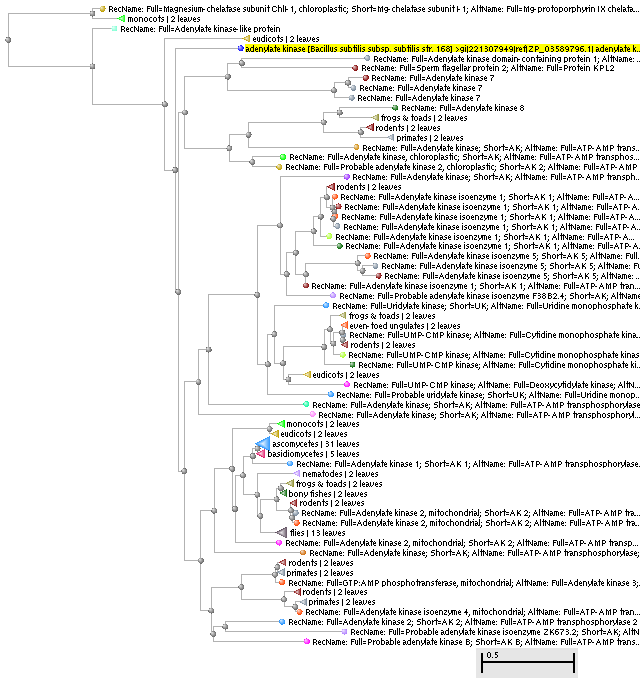

Рисунок 1. Распределение гомологов KAD_BACSU по таксонам.
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
1. Лучшая находка (с последовательностью исходного белка) |
|||
| Accession | P16304 | 3DL0_A (цепь А) | NP_388018.1 |
| E-value | 9e-157 | 1e-156 | 4e-155 |
| Вес (в битах) | 440 | 436 | 440 |
| Процент идентичности | 100% | 100% | 100% |
2. Число находок с E-value < 10–10 |
661 | 42 | 20000 |
3. "Худшая из удовлетворительных" находка (последняя в выдаче с E-value < 1) |
|||
| Номер находки в списке описаний | 737 | 45 | 20000 |
| Accession | Q9UZJ6.1 | 1Q3T | CCG18940.1 |
| E-value | 0,98 | 0,67 | 2e-61 |
| Вес (в битах) | 33,1 | 30,8 | 202 |
| % идентичности | 29% | 32% | . |
| % сходства | 47% | 56% | . |
| Длина выравнивания | 59 | 50 | . |
| Координаты выравнивания (от-до, в запросе и в находке) | 3-61 в запросе, 8-63 в находке | 1-48 в запросе, 17-66 в находке | . |
| Число гэпов | 3 | 2 | . |
| SwissProt | PDB | nr | |
| Число явных гомологов | 661 | 41 | 20000 |
| Общее количество находок | 797 | 60 | 20000 |
| E-value последней находки | 9,8 | 7,6 | 2e-61 |
| Лимитирующий фактор запроса | E-value | E-value | Предельный размер выдачи |
 |
 Рисунок 1. Распределение гомологов KAD_BACSU по таксонам. |
"Лучший" гомолог в Eukaryota (E-value < 0,001) |
|||
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr"* | |
| Номер находки в списке описаний | 1 | 1 | 1 |
| Accession | Q08480.1 | 3BE4_A | XP_001122889.2 |
| E-value | 1e-68 | 1e-63 | 3e-76 |
| Вес (в битах) | 215 | 199 | 240 |
| % идентичности | 47% | 48% | 54% |
| % сходства | 69% | 65% | 71% |
| Длина выравнивания | 212 | 211 | 215 |
| Координаты выравнивания (от-до, в запросе и в находке) | 2-213 в запросе, 32-243 в находке | 2-212 в запросе, 7-217 в находке | 4-216 в запросе, 1-215 в находке |
| Число гэпов | 0 | 0 | 0 |
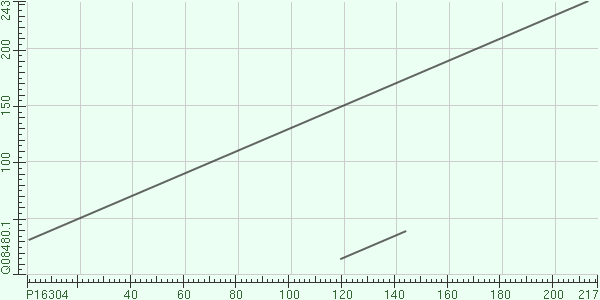 Рисунок 2.Выравнивание последовательностей при пороге e-value 10. |
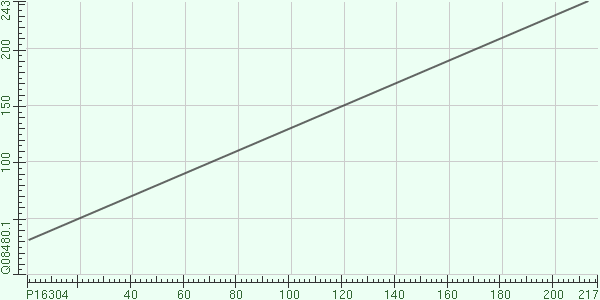 Рисунок 3.Выраванивание последовательностей при пороге e-value 0,01. |
| Матрица BLOSUM62 | Матрица BLOSUM90 | |
| Номер находки в списке описаний | 1 | 1 |
| Accession | Q08480.1 | Q08480.1 |
| E-value! | 1e-68 | 4e-65 |
| Вес (в битах)! | 215 | 220 |
| % идентичности | 47% | 47% |
| % сходства ! | 69% | 63% |
| Длина выравнивания | 212 | 212 |
| Координаты выравнивания (от-до, в запросе и в находке) | 2-213 в запросе, 32-243 в находке | 2-213 в запросе, 32-243 в находке |
| Число гэпов | 0 | 0 |
| Главная страница | Первый семестр | Второй семестр | Обо мне | Ссылки |