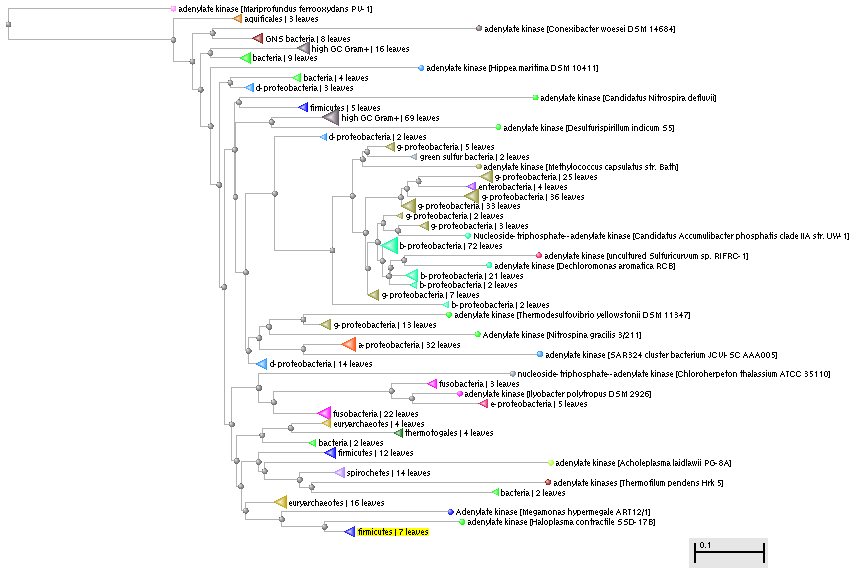

Рисунок 1. Филогенетическое дерево гомологов KAD_BACSU у прокариот.
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количесвто хитов |
| По прокариотам | blastp | RefSeq | см таблицу 1' | 1 | 20000 |
| По эукариотам | blastp | RefSeq | см таблицу 1' | 1 | 1542 |
| Запрос № | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 |
| Таксон | Nitrospinae Ignavibacteria Tenericutes Synergistetes |
Poribacteria Lentisphaerae Chlamydiae |
Thermodesulfobacteria Chrysiogenetes Deferribacteres Thermotogae Chloroflexi Aquificae Gemmatimonadetes Elusimicrobia Dictyoglomi Armatimonadetes Caldiserica Fibrobacteres Acidobacteria Nitrospirae |
Cyanobacteria | Fusobacteria | Spirochaetes | Chlorobi Bacteroidetes |
Planctomycetes | Deinococcus-Thermus | Thaumarchaeota Nanoarchaeota Korarchaeota Euryarchaeota Crenarchaeota |
a-Proteobacteria | b-Proteobacteria | d-Proteobacteria | e-Proteobacteria | g-Proteobacteria | Actinobacteria |
| Запрос № | 1 | 2 | 3 | 4 |
| Таксоны | Animlas | Fungi | Amoebozoa | Филумы запросов 1-3 + exclude |
 |
 Рисунок 1. Филогенетическое дерево гомологов KAD_BACSU у прокариот. |
 |
  |
| Рисунок 2. Филогенетическое дерево гомологов KAD_BACSU у эукариот. |
| Домен | Филум | Вид | Количество хитов на филум |
| Archeae | Crenarchaeota | Thermofilum pendens | 31 |
| Euryarcheota | Haloferax larsenii | 271 | |
| Methanosarcina mazei | |||
| Korarcheota | Korarchaeum cryptofilum | 1 | |
| Bacteria | Acidobacteria | Terriglobus saanensis | 6 |
| Actinobacteria | Actinomyces odontolyticus | 434 | |
| Turicella otitidis | |||
| Conexibacter woesei | |||
| Aquificae | Hydrogenivirga sp. | 12 | |
| Bacteroidetes (CFB) | Barnesiella intestinihominis | 240 | |
| Dyadobacter fermentans | |||
| Caldiserica | Caldisericum exile | 1 | |
| Chlamydiae | Diplosphaera colitermitum | 31 | |
| Chlorobi | Chlorobium phaeobacteroides | 12 | |
| Chloroflexi | Oscillochloris trichoides | 26 | |
| Chrysiogenetes | Desulfurispirillum indicum | 2 | |
| Cyanobacteria | Microcystis aeruginosa | 196 | |
| Oscillatoria sp. | |||
| Anabaena variabilis | |||
| Deferribacteres | Denitrovibrio acetiphilus | 6 | |
| Deinococcus-Thermus | Truepera radiovictrix | 27 | |
| Oceanithermus profundus | |||
| Dictyoglomi | Dictyoglomus thermophilum | 2 | |
| Elusimicrobia | Elusimicrobium minutum | 1 | |
| Fibrobacteres | Fibrobacter succinogenes | 2 | |
| Firmicutes | Bacillus subtilis | . | |
| Fusobacteria | Ilyobacter polytropus | 34 | |
| Leptotrichia goodfellowii | |||
| Gemmatimonadetes | Gemmatimonas aurantiaca | 1 | |
| Ignavibacteria | Ignavibacterium album | 3 | |
| Lentisphaerae | Lentisphaera araneosa | 1 | |
| Nitrospinae | Nitrospina gracilis | 1 | |
| Nitrospirae | Thermodesulfovibrio yellowstonii | 4 | |
| Planctomycetes | Blastopirellula marina | 42 | |
| a-Proteobacteria | Agrobacterium radiobacter | 500 | |
| Rickettsia africae | |||
| Commensalibacter intestini | |||
| b-Proteobacteria | Alcaligenes sp. | 195 | |
| Neisseria mucosa | |||
| Nitrosospira multiformis | |||
| d-Proteobacteria | Desulfovibrio magneticus | 91 | |
| Corallococcus coralloides | |||
| Bacteriovorax marinus | |||
| e-Proteobacyeria | Helicobacter cetorum | 236 | |
| Arcobacter nitrofigilis | |||
| Nautilia profundicola | |||
| g-Proteobacteria | Escherichia coli | 500 | |
| Vibrio parahaemolyticus | |||
| Aeromonas aquariorum | |||
| Spirochetes | Treponema primitia | 78 | |
| Leptospira borgpetersenii | |||
| Synergistetes | Aminobacterium colombiense | 14 | |
| Tenericutes | Mycoplasma penetrans | 61 | |
| Thermodesulfobacteria | Thermodesulfatator indicus | 3 | |
| Thermotogae | Thermotoga neapolitana | 17 | |
| Eukarya | Amoebozoa | Dictyostelium purpureum | 32 |
| Entamoeba invadens | |||
| Acanthamoeba castellanii | |||
| Viridiplantae | Oryza sativa | 235 | |
| Ricinus communis | |||
| Physcomitrella patens | |||
| Volvox carteri | |||
| Kinetoplastida | Trypanosoma cruzi | 57 | |
| Apicomplexa | Plasmodium vivax | 53 | |
| Ciliophora | Paramecium tetraurelia | 50 | |
| Diatoms | Thalassiosira pseudonana | 15 | |
| Oomycota | Phytophthora infestans | 11 | |
| Diplomonada | Giardia lamblia | 3 | |
| Animals | Xenopus laevis (mit) | 500 | |
| Danio rerio (mit) | |||
| Bos taurus (mit) | |||
| Homo sapiens (mit) | |||
| Ciona intestinalis (mit) | |||
| Apis mellifera | |||
| Ixodes scapularis | |||
| Trichinella spiralis | |||
| Hydra magnipapillata (mit) | |||
| Strongylocentrotus purpuratus (mit) | |||
| Saccoglossus kowalevski | |||
| Amphimedon queenslandica (mit) | |||
| Fungi | Kazachstania africana | 268 | |
| Aspergillus fumigatus | |||
| Chaetomium globosum | |||
| Magnaporthe oryzae | |||
| Coprinopsis cinerea | |||
| Puccinia graminis | |||
| Encephalitozoon hellem |

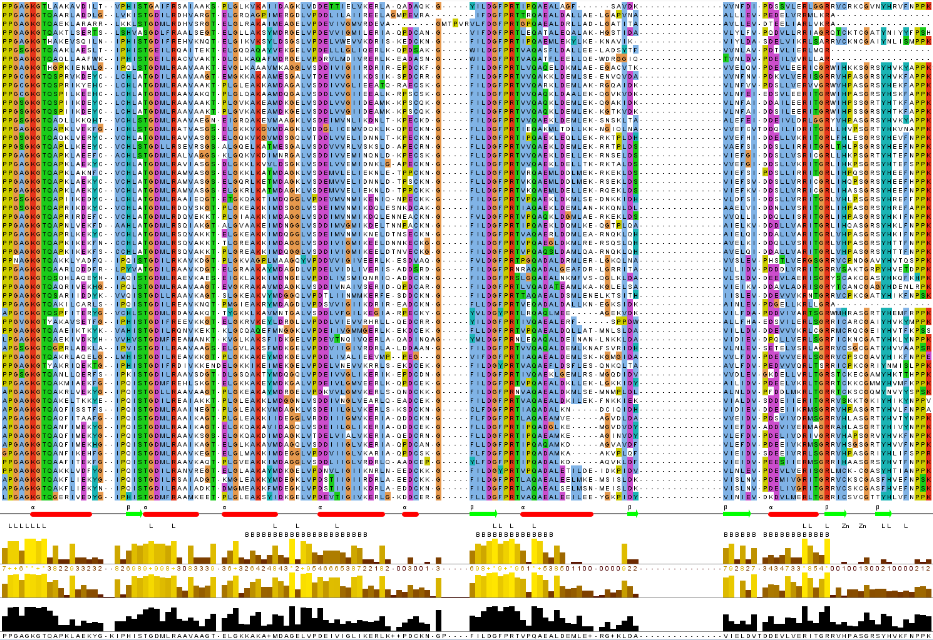
 |
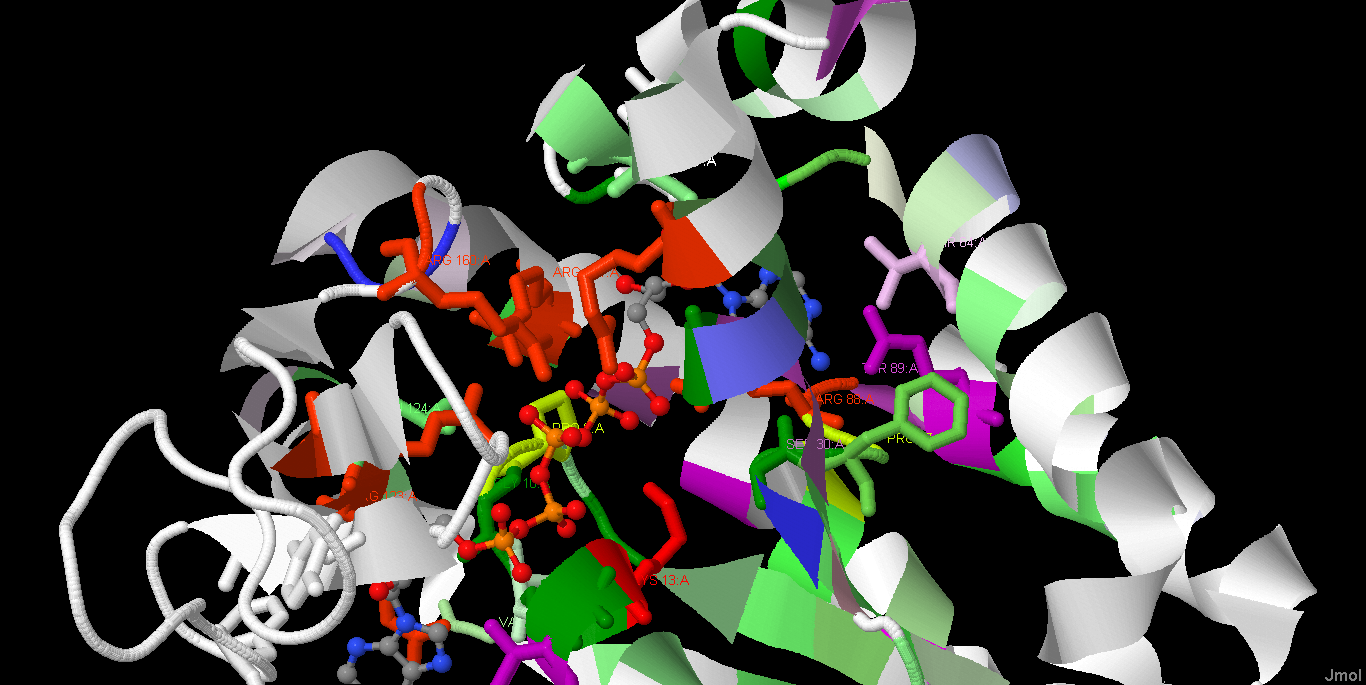 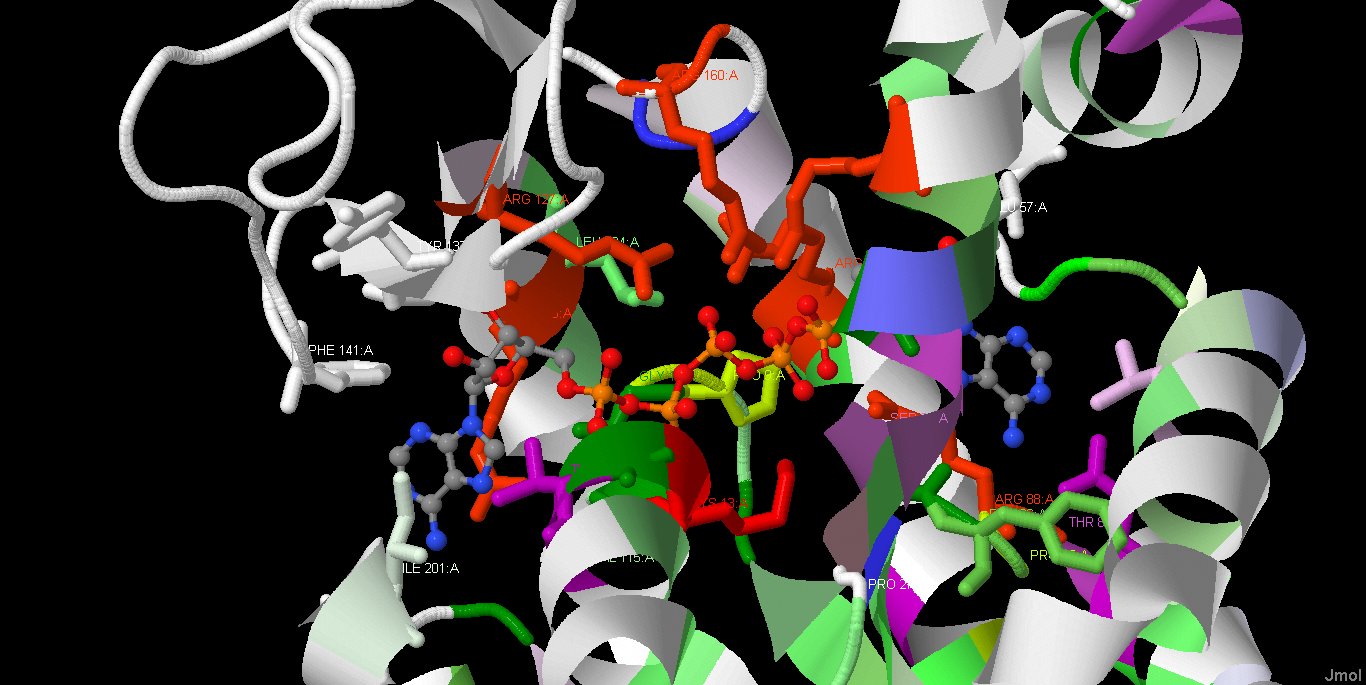 |
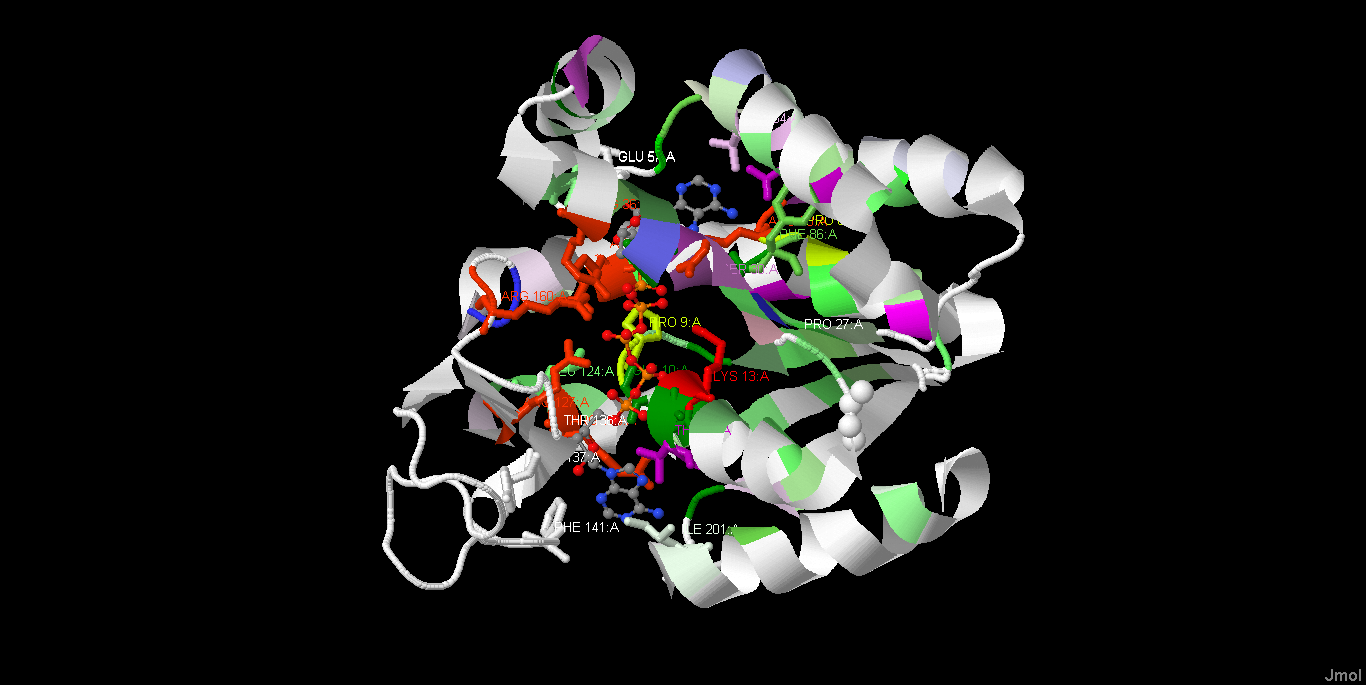
 | Рисунок 6. Область связывания иона цинка в цепи А. Стили использованы такие же, как на рисунке 5. Цинк обозначен синим. |

| Главная страница | Первый семестр | Второй семестр | Обо мне | Ссылки |