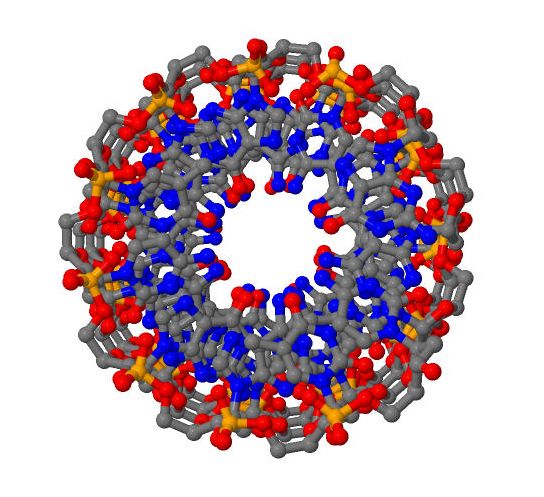
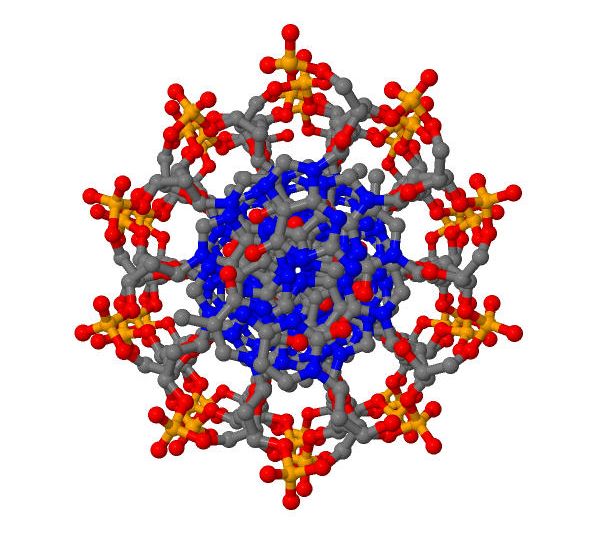
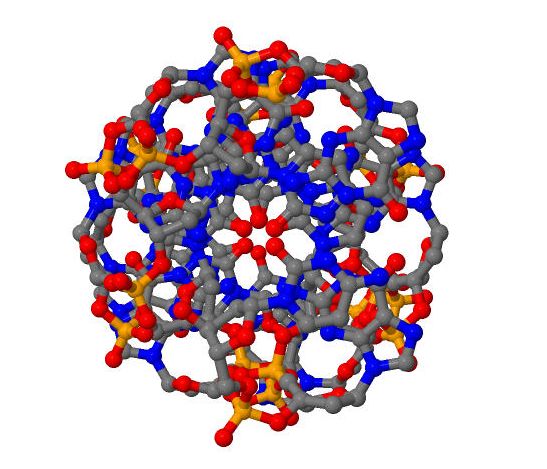
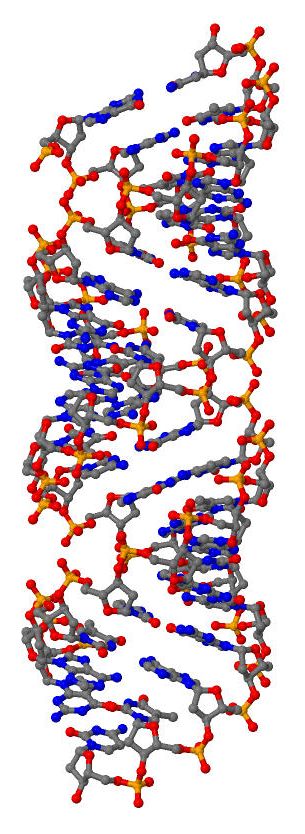
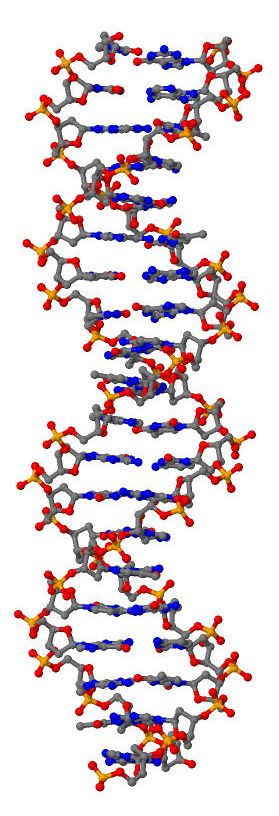

ДНК может существовать в форме А-, В- или Z-спирали. Для построения трехмерных моделей этих структур используются программы пакета 3DNA. В данном случае были построены три структуры с помощью команды "fiber":
fiber -a gatc-a.pdb
fiber -b gatc-b.pdb
fiber -z gatc-z.pdbТаким образом были построены А и В-формы ДНК, состоящие из пятикратных повторов последовательности GATС, и Z-форма, состоящая из 10 G-C пар (fiber не дает альтернативы). Полученные структуры представлены на рисунке 1.
 |
 |
 |
 |
 |
 |
| А-форма | B-форма | Z-форма |
 |  |
 |  |
 |
 |
 |
 |
 |
 |
 |
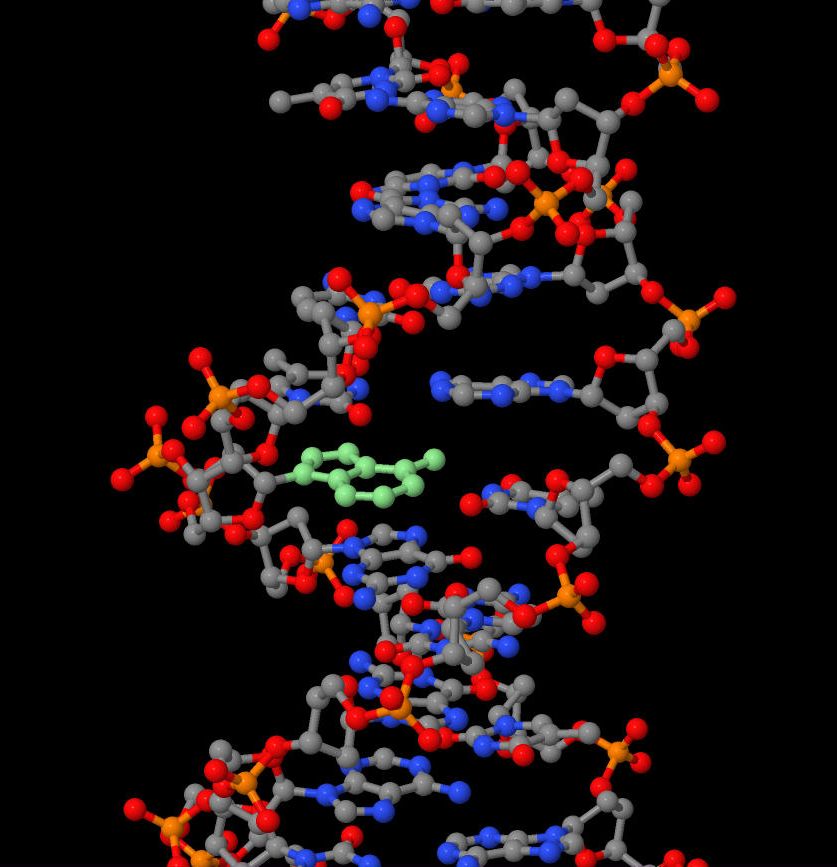
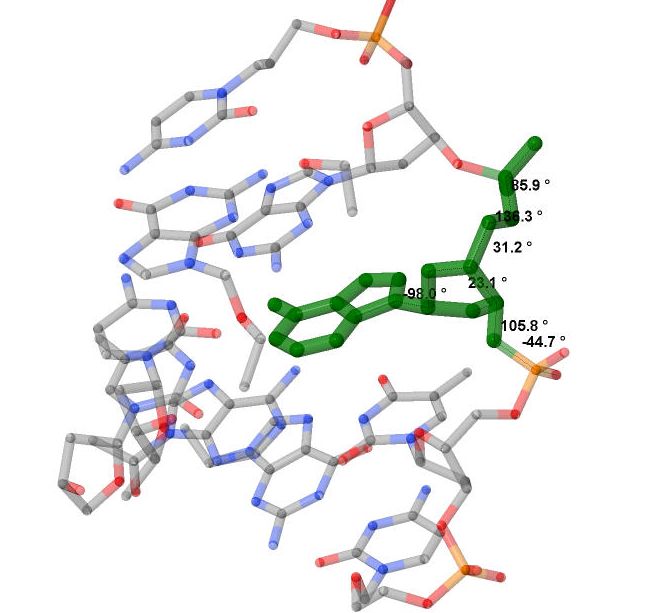
| Рис. 6. Слева зеленым цветом показано расположение выбранного аденина в положении 10 структуры B-спирали. Справа красным выделены атомы, обращенные к большой бороздке, синим - к малой. |
 |
 |
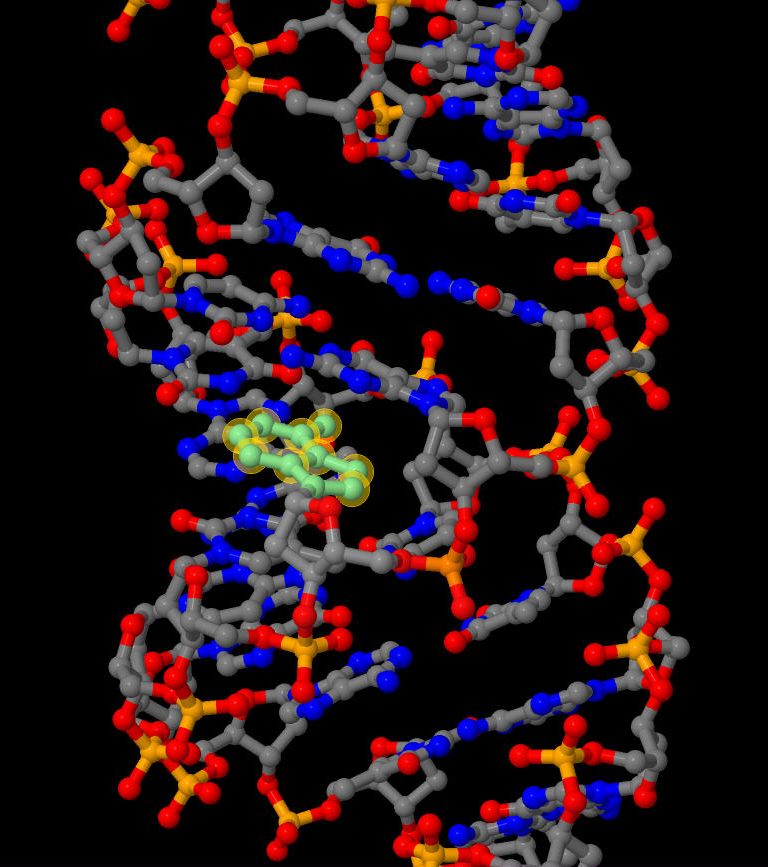
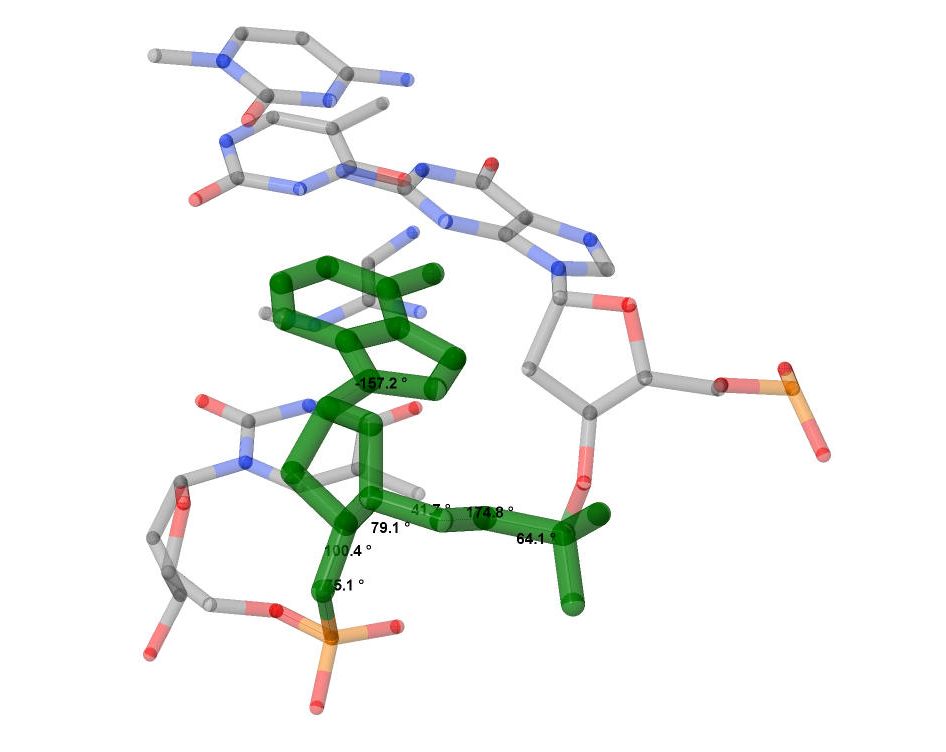
| Рис. 7. Слева зеленым цветом показано расположение выбранного аденина в положении 30 структуры A-спирали. Справа красным выделены атомы, обращенные к большой бороздке, синим - к малой. |
 |
 |
 |
 |
 |
 |
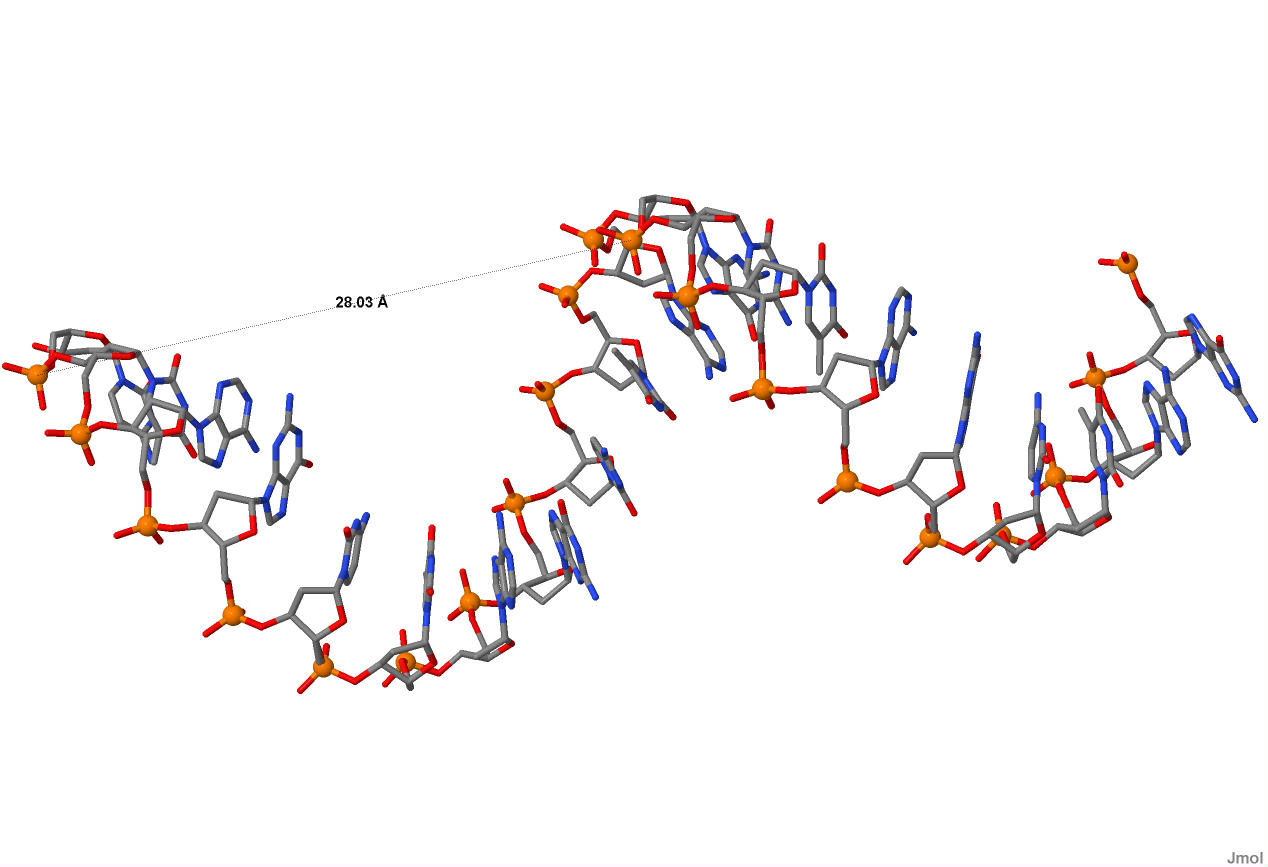
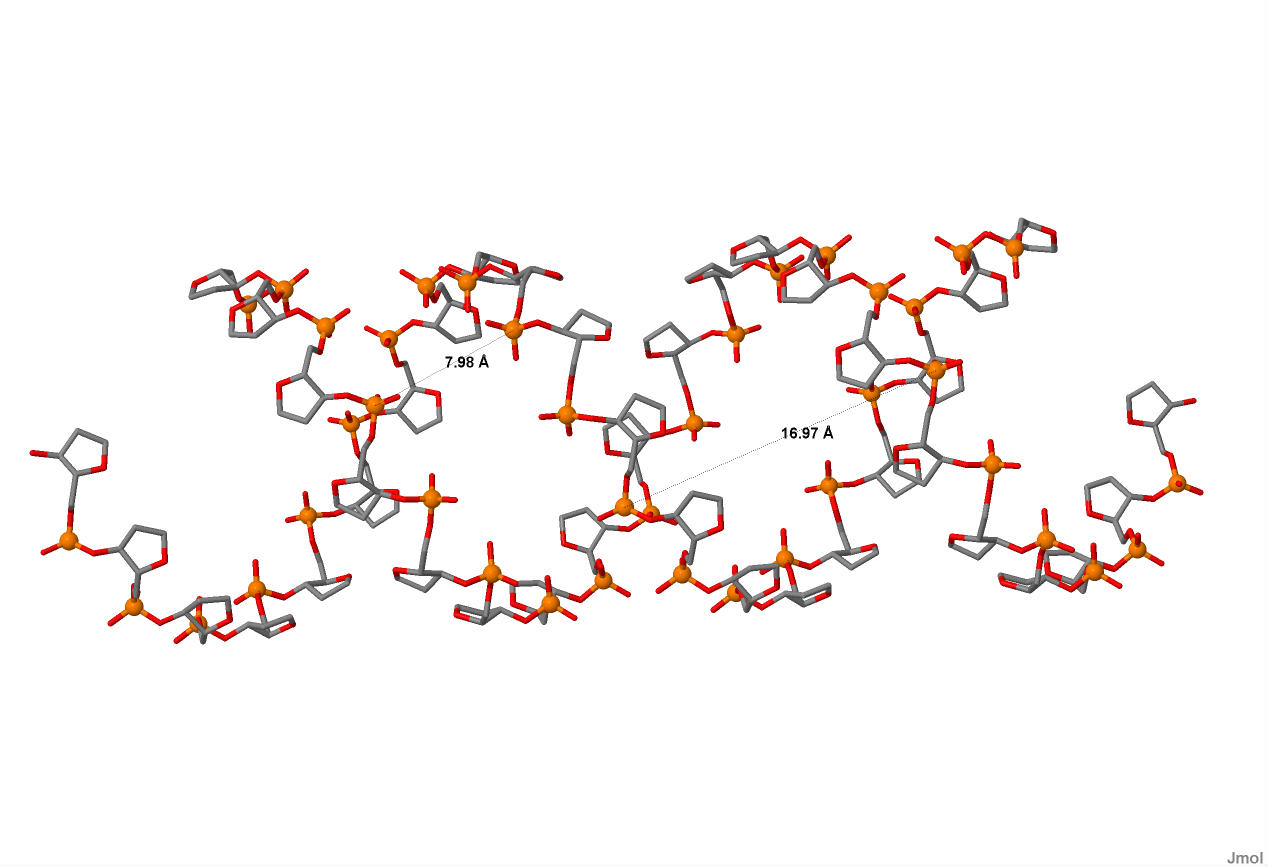
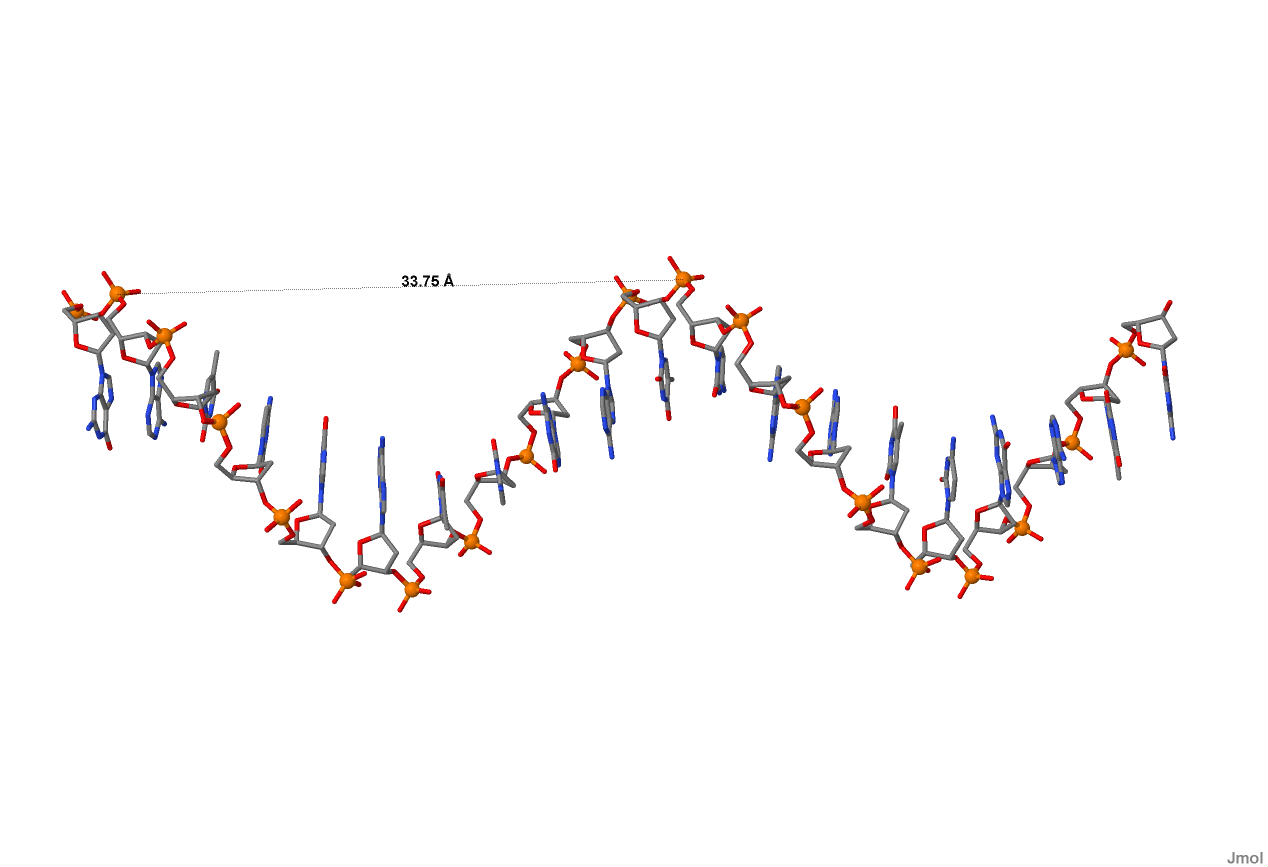
| Спиральный параметр | А-форма | В-форма | Z-форма |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (A) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
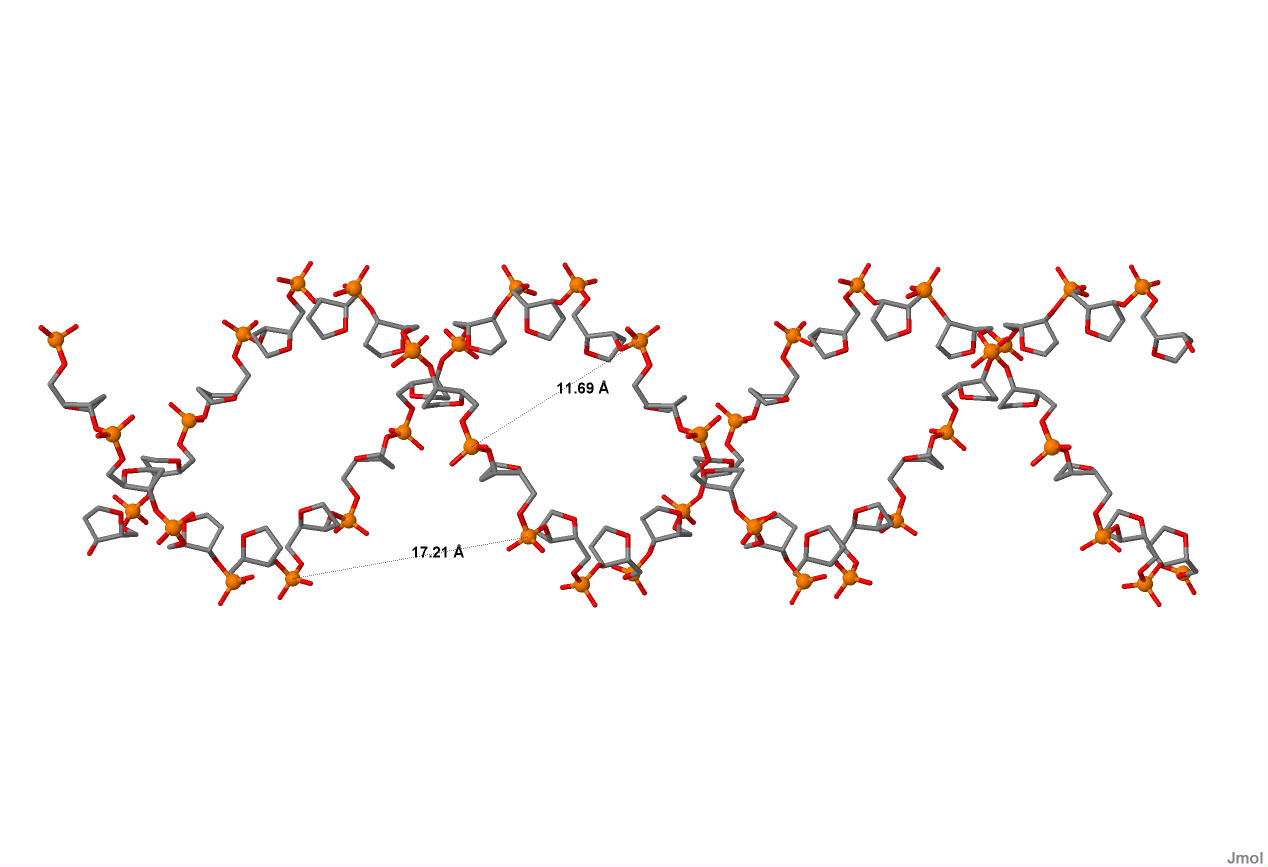
| Ширина большой бороздки | 16.97 | 17.21 | 18.3 |
| Ширина малой бороздки | 7.98 | 11.69 | 9.87 |
 |
 |
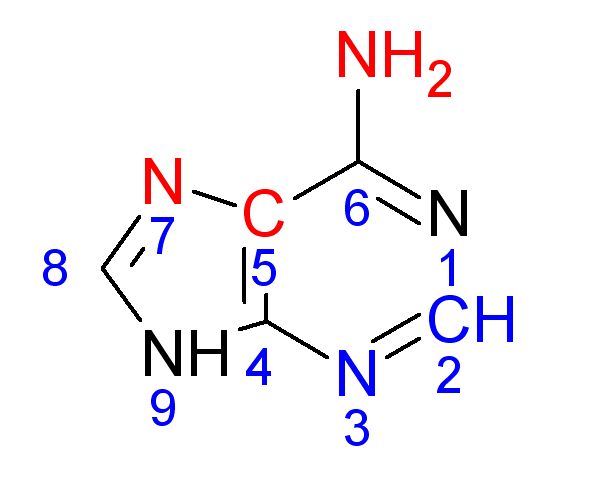
| Форма | α | β | γ | δ | ε | ζ | χ |
| А | 64,1 | 174,8 | 41,7 | 79,1 | 100,4 | -75,1 | -157,2 |
| В | 85,9 | 136,3 | 31,2 | 23,1 | 105,8 | -44,7 | -98,3 |
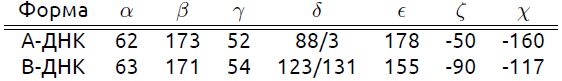 |
remediator --old "XXXX.pdb" > "XXXX_old.pdb"Это необходимо, потому что 3DNA работает только сос старым форматом. Теперь о структурах, записанных в pdb-файлах можно получить довольно много информации с помощью команд find_pair и analyze:
find_pair -t XXXX.pdb stdout | analyzeПроанализируем сначала файлы gatc-X_old.pdb.
| Форма | α | β | γ | δ | ε | ζ | χ |
| А | -57,7 | 174,8 | 41,7 | 79,1 | -147,8 | -75,1 | -157,2 |
| В | -29,9 | 136,3 | 31,2 | 143,3 | -140,8 | -160,5 | -98,3 |
| α | β | γ | δ | ε | ζ | χ |
| (-63,4)-(152,8) | (-176,4)-(179,4) | (51,4)-(178,9) | (81,5)-(88,5) | (-178,0)-(-147,2) | (-81,3)-(-64,7) | (-163,5)-(149,2) |
| α | β | γ | δ | ε | ζ | χ |
| -32,28 | 156,88 | 35,82 | 137,32 | -97,44 | -107,14 | -114,32 |
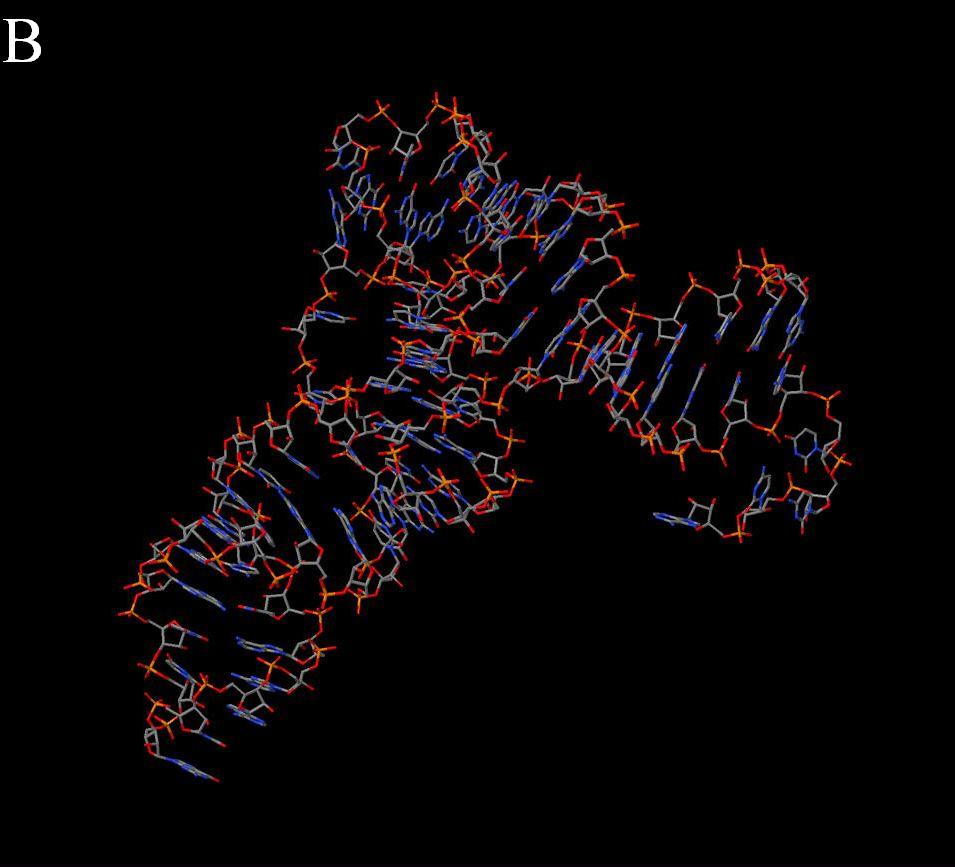
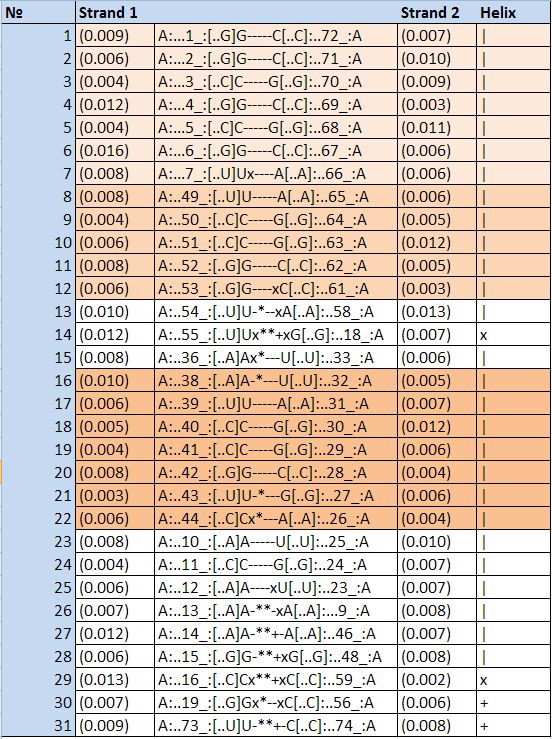 | Рис. 12. Пары оснований тРНК из структуры 1U0B, цветом выделены стебли. |
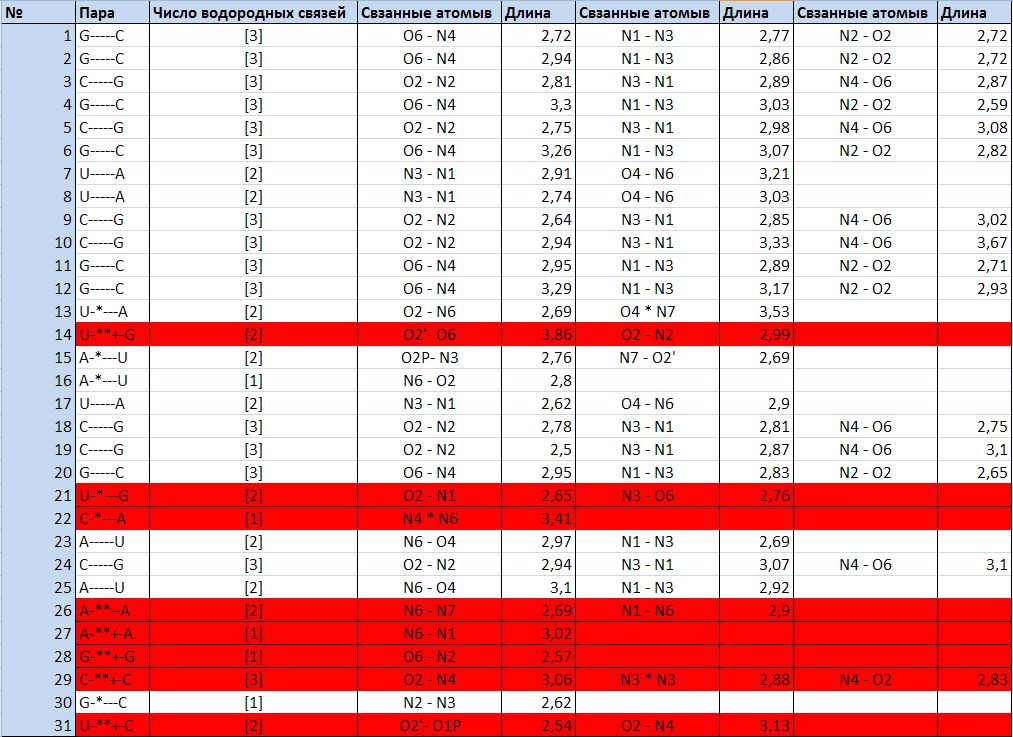 | Рис. 13. Неканонические пары оснований выделены красным |
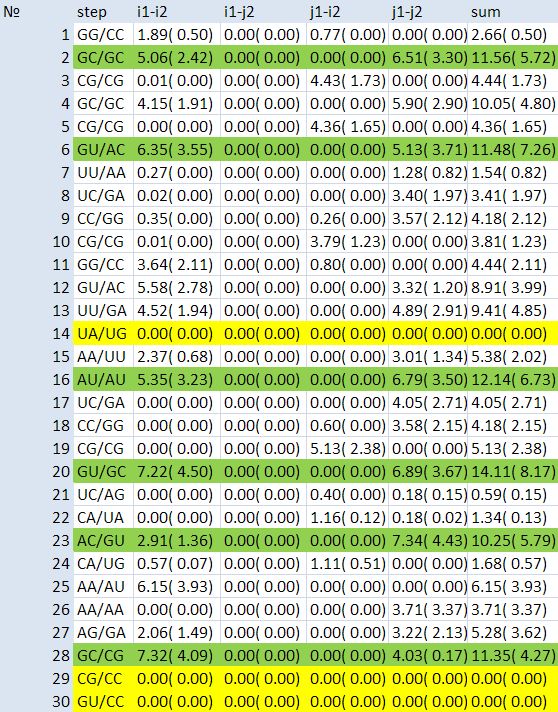 |
Рис. 14. Зеленым выделены наиболее перекрывающиеся основания, желтым - наименее перекрывающиеся. |
ex_str -X stacking.pdb stepX.pdb
stack2img -cdolt stepX.pdb stepX.ps
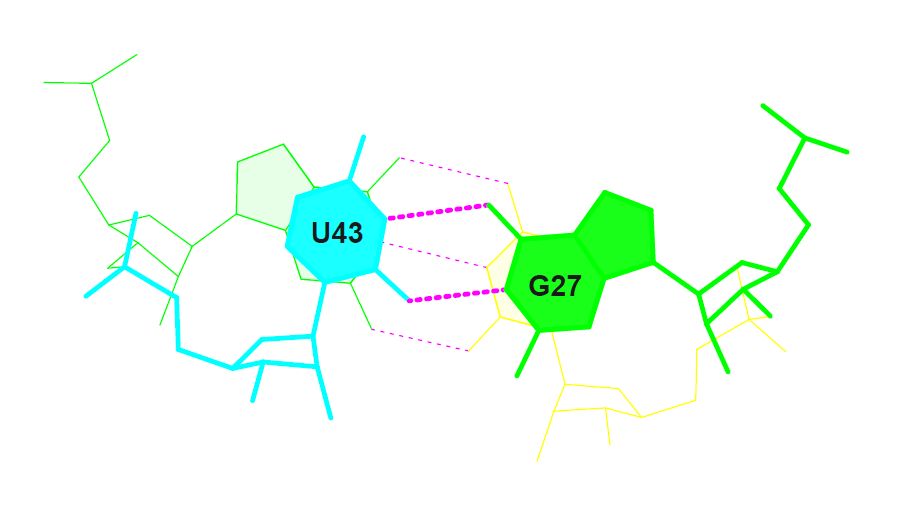 |
 |
| Главная страница | Первый семестр | Второй семестр | Третий семестр | Обо мне | Ссылки |