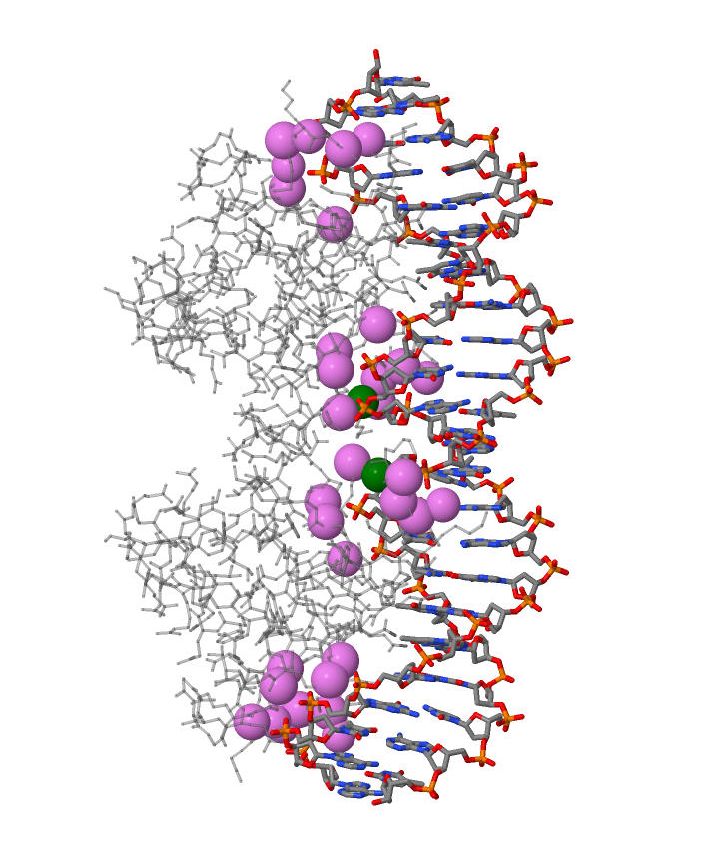
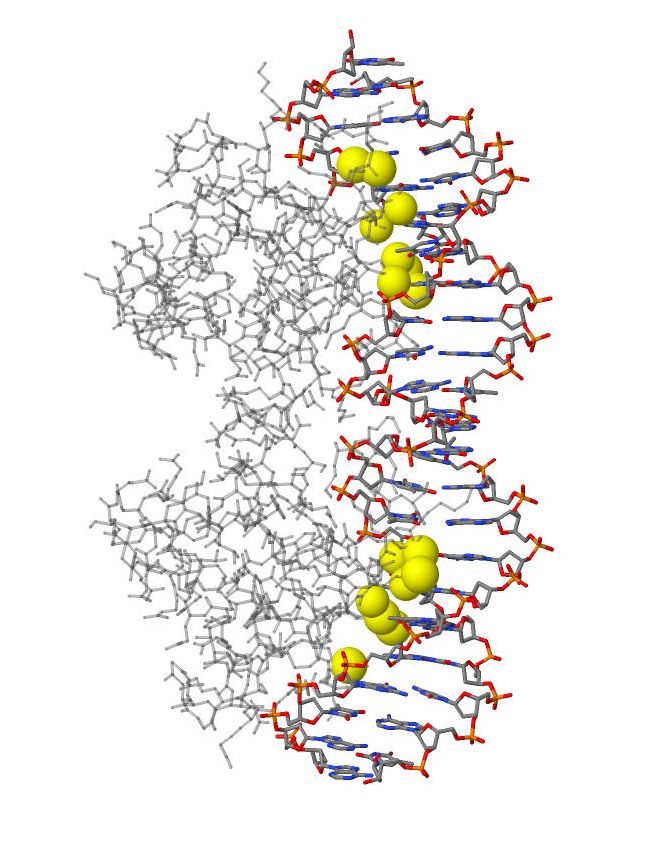
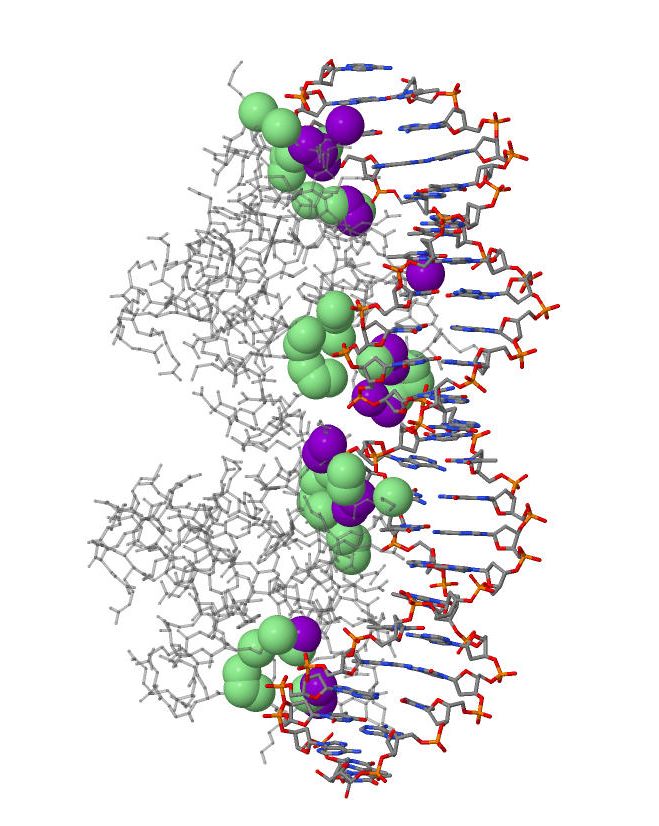
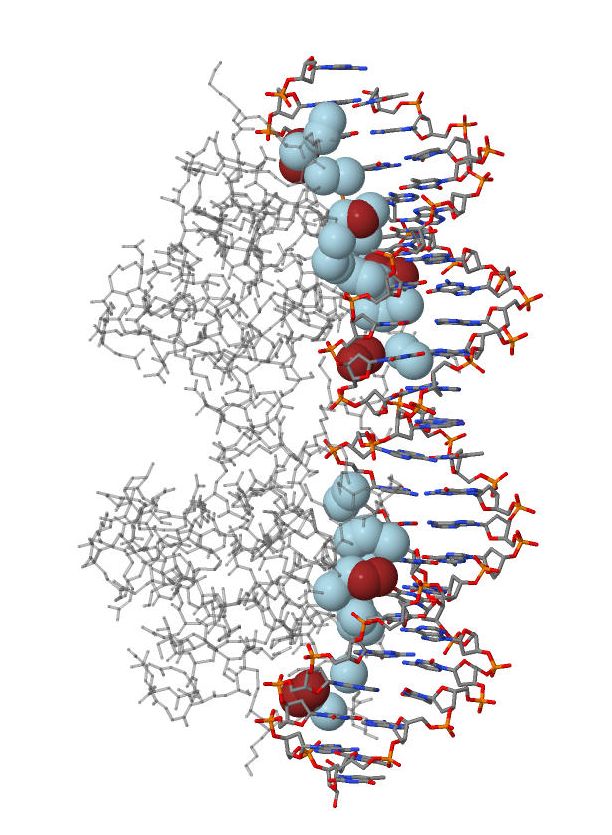
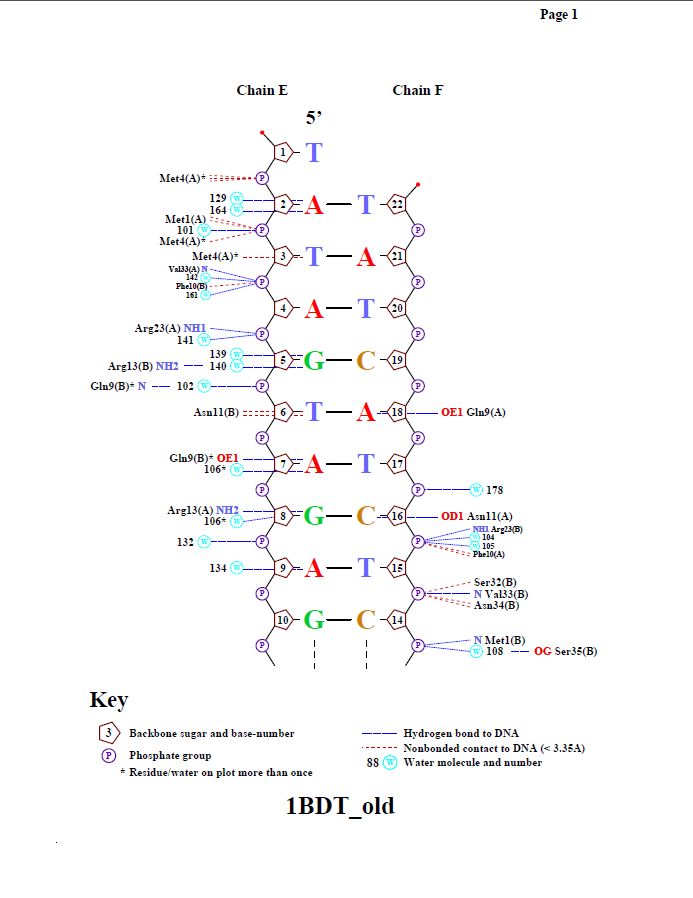
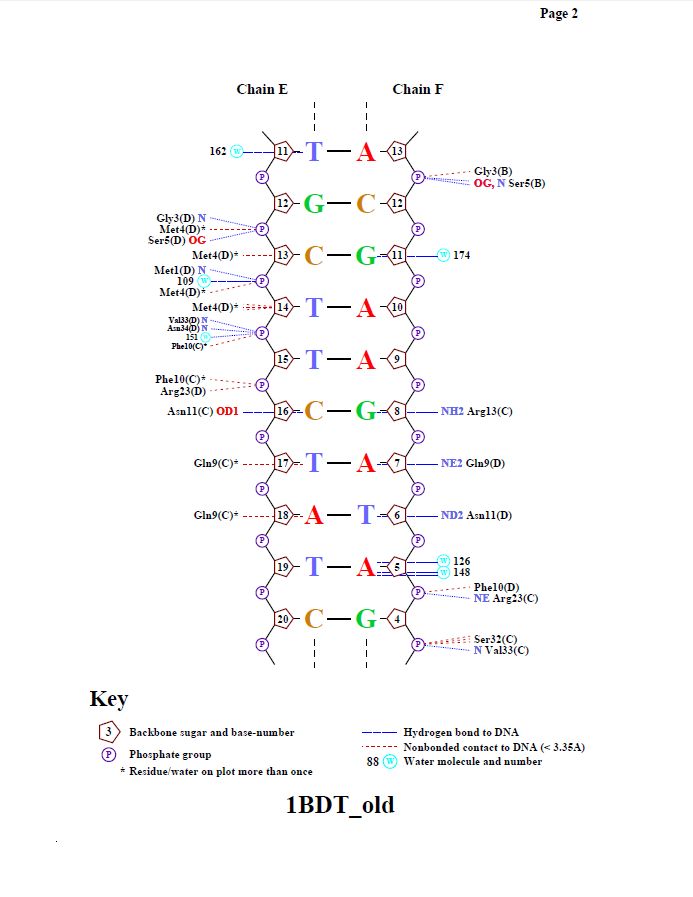
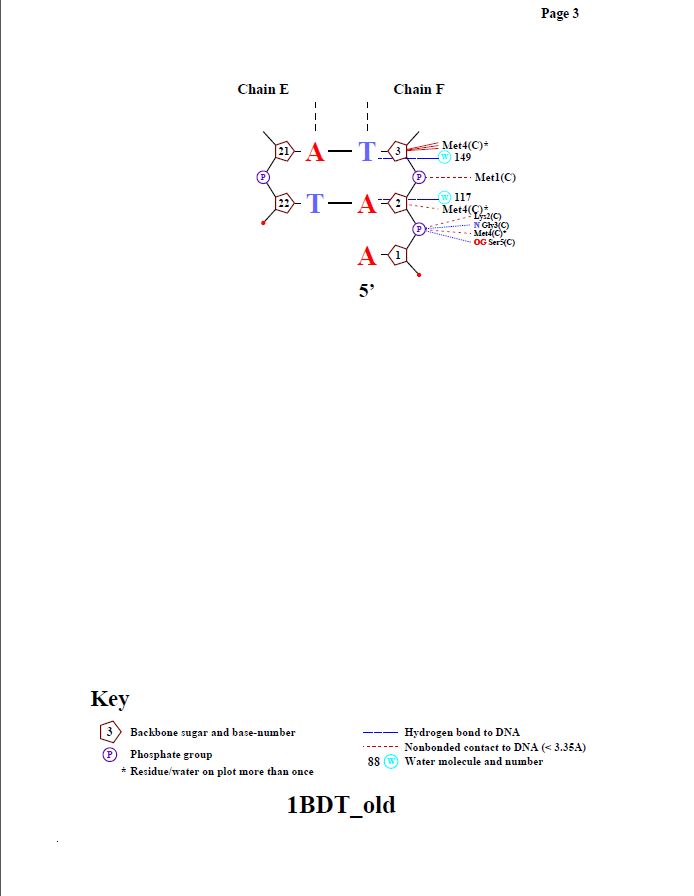
В этом разделе изучается ДНК-белковый комплекс структуры 1BDT.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 2 | 29 | 31 |
| остатками фосфорной кислоты | 35 | 41 | 76 |
| остатками азотистых оснований со стороны большой бороздки | 16 | 53 | 69 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 11 | 11 |
 |
 |
 |
 |
| Полярные. Зеленые - с дезоксирибозой, розовые - с фосфатами. | Полярные. Желтые - атомами оснований большой бороздки. | Неполярные. Фиолетовые - с дезоксирибозой, зеленые - с фосфатами. | Неполярные. Голыбые - с атомами оснований большой бороздки, коричневые - малой бороздки. |
nucplot 1BDT_old.pdb
 |
 |
 |
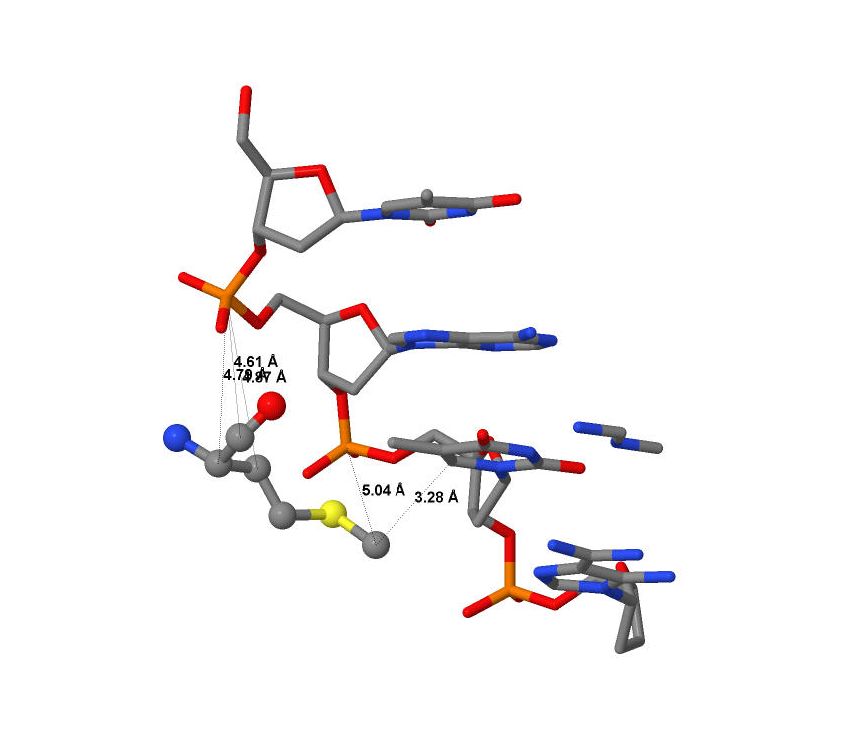 | 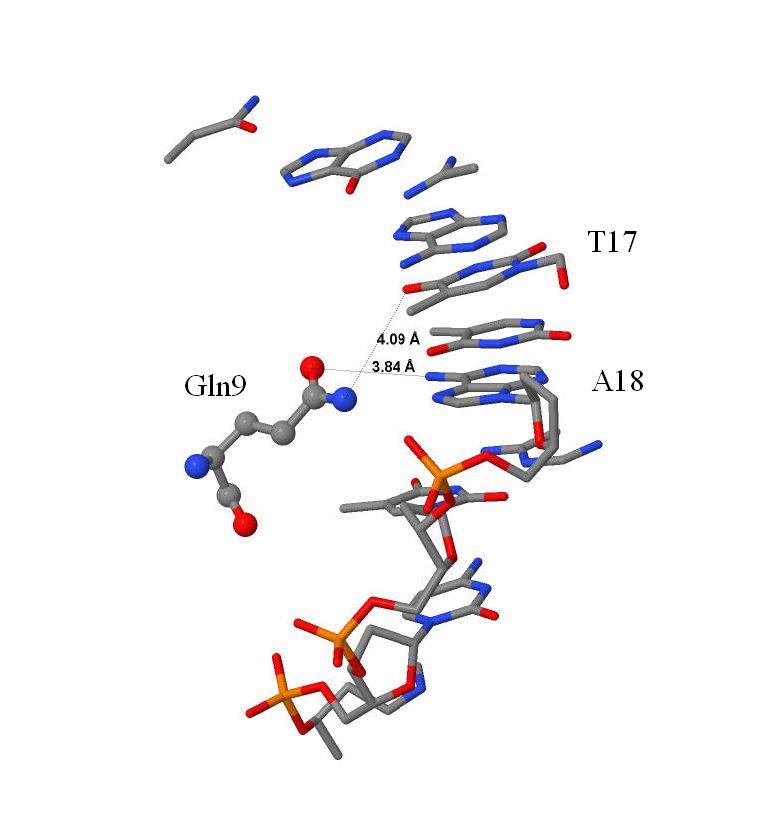 |
| Рис. 3. Слева четвертый метионин и его контакты с ДНК, справа - контакты девятого глутамина с ДНК. |
SEQUENCE: Score 48: 21/32 ( 65%) matches, 3 gaps
1 ggcgcgttaacaaagcggttatg--tag-cggatt 32
|||||||| | || | ||| ||||||
70 ccgcgcaaggcctcagcttggcctgatctgcctaa 36
В алгоритме Зукера можно менять множество разных параметров входа, но здесь имело смысл менять только параметр "P", который отражает отклонение энергии структуры от оптимального. После попыток применить разные значения параметра я сделал вывод, что стоит оставить P=0 и получил изображение вторичной структуры, приведенное ниже.
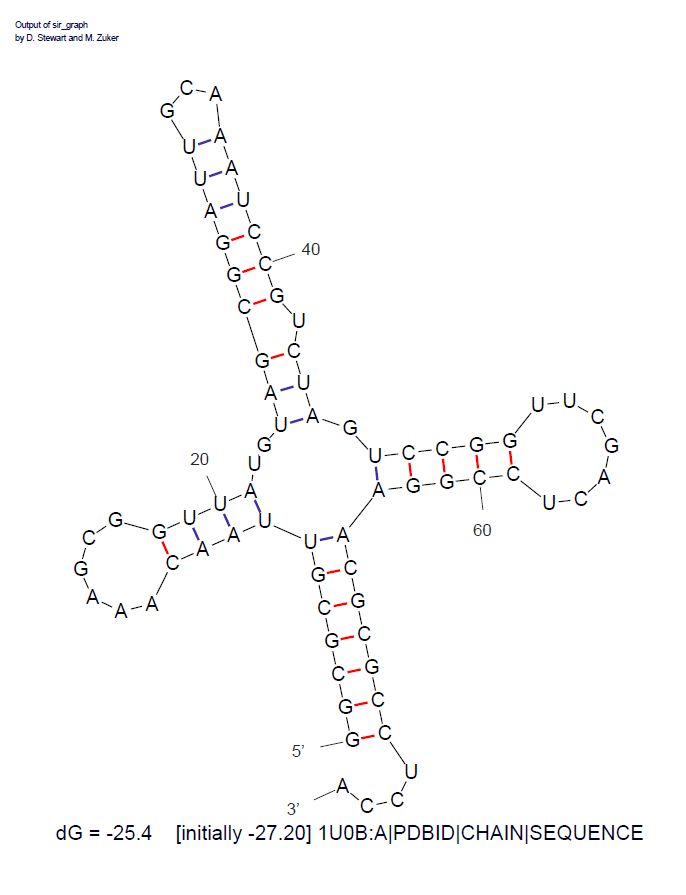 | Рис. 4. Структура тРНК, полученная алгоритмом Зукера. |
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-1-7-3' 5'-66-72-3' Всего 7 пар |
Предсказано 7 пар из 7 реальных | Предсказано 7 пар из 7 реальных |
| D-стебель | 5'-10-12-3' 5'-23-25-3' Всего 3 пары |
Предскзано 0 пар из 3 реальных | Предскзано 4 пар из 3 реальных** |
| T-стебель | 5'-49-53-3' 5'-61-65-3' Всего 5 пар |
Предсказано 0 пар из 5 реальных | Предсказано 5 пар из 5 реальных |
| Антикодоновый стебель | 5'-38-44-3' 5'-26-32-3' Всего 7 пар |
Предсказано 6 пар из 7 реальных | Предсказано 9 пар из 7 реальных* |
| Общее число канонических пар нуклеотидов | 20 | 13 | 22 |
| Главная страница | Первый семестр | Второй семестр | Третий семестр | Обо мне | Ссылки |